مƒ‡م‚ھم‚م‚·مƒھمƒœو ¸é…¸ 
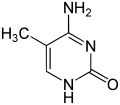
مƒ‡م‚ھم‚م‚·مƒھمƒœو ¸é…¸ï¼ˆمƒ‡م‚ھم‚م‚·مƒھمƒœمپ‹مپڈمپ•م‚“م€پ英: deoxyribonucleic acidم€پDNA[1])مپ¯م€پ2وœ¬مپ®مƒمƒھمƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰éژ–مپŒن؛’مپ„مپ«ه·»مپچمپ¤مپ„مپ¦ن؛Œé‡چم‚‰مپ›م‚“م‚’ه½¢وˆگمپ—مپ¦مپ„م‚‹مƒمƒھمƒمƒ¼مپ§مپ‚م‚‹م€‚مپ“مپ®مƒمƒھمƒمƒ¼مپ¯م€پمپ™مپ¹مپ¦مپ®و—¢çں¥مپ®ç”ں物مپ¨ه¤ڑمپڈمپ®م‚¦م‚¤مƒ«م‚¹مپ®ç™؛ç”ںم€پو©ں能م€پوˆگé•·م€پمپٹم‚ˆمپ³ç”ںو®–مپ®مپںم‚پمپ®éپ؛ن¼çڑ„ه‘½ن»¤م‚’ن¼éپ”مپ™م‚‹م€‚DNAمپ¯مƒھمƒœو ¸é…¸ï¼ˆè‹±: ribonucleic acidم€پRNA)مپ¨مپ¨م‚‚مپ«و ¸é…¸مپ¨ç·ڈ称مپ•م‚Œم‚‹م€‚و ¸é…¸مپ¯م‚؟مƒ³مƒ‘م‚¯è³ھم€پè„‚è³ھم€پ複هگˆه¤ڑç³–مپ¨ن¸¦م‚“مپ§م€پمپ™مپ¹مپ¦مپ®و—¢çں¥مپ®ç”ںه‘½ن½“مپ«مپ¨مپ£مپ¦ن¸چهڈ¯و¬ مپھ4ه¤§ç”ںن½“é«کهˆ†هگمپ®مپ²مپ¨مپ¤مپ§مپ‚م‚‹م€‚ DNAمپ®ن؛Œوœ¬éژ–مپ¯م€پمƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰مپ¨ه‘¼مپ°م‚Œم‚‹م‚ˆم‚ٹهچکç´”مپھهچکé‡ڈن½“هچکن½چمپ‹م‚‰و§‹وˆگمپ•م‚Œمپ¦مپ„م‚‹مپ“مپ¨مپ‹م‚‰م€پمƒمƒھمƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰مپ¨ه‘¼مپ°م‚Œم‚‹[2][3]م€‚هگ„مƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰مپ¯م€پ4مپ¤مپ®çھ’ç´ هگ«وœ‰و ¸é…¸ه،©هں؛(م‚·مƒˆم‚·مƒ³: Cم€پم‚°م‚¢مƒ‹مƒ³: Gم€پم‚¢مƒ‡مƒ‹مƒ³: Aم€پمƒپمƒںمƒ³: T)مپ®مپ†مپ،مپ®1مپ¤م€پمƒ‡م‚ھم‚م‚·مƒھمƒœمƒ¼م‚¹مپ¨ه‘¼مپ°م‚Œم‚‹ç³–م€پمپٹم‚ˆمپ³مƒھمƒ³é…¸هں؛مپ§و§‹وˆگمپ•م‚Œمپ¦مپ„م‚‹م€‚مپ‚م‚‹مƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰مپ®ç³–مپ¨م€پو¬،مپ®مƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰مپ®مƒھمƒ³é…¸مپŒه…±وœ‰çµگهگˆï¼ˆمƒ›م‚¹مƒ›م‚¸م‚¨م‚¹مƒ†مƒ«çµگهگˆمپ¨ه‘¼مپ°م‚Œم‚‹ï¼‰مپ«م‚ˆمپ£مپ¦éژ–çٹ¶مپ«çµگهگˆمپ—م€پç³–-مƒھمƒ³é…¸مپŒن؛¤ن؛’مپ«ç¹°م‚ٹè؟”مپ•م‚Œم‚‹ن¸»éژ–مپŒه½¢وˆگمپ•م‚Œم‚‹م€‚ن؛Œوœ¬مپ®مƒمƒھمƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰éژ–مپ®çھ’ç´ ه،©هں؛مپ¯م€په،©هں؛ه¯¾هگˆه‰‡ï¼ˆAمپ¨Tم€پCمپ¨G)مپ«ه¾“مپ£مپ¦و°´ç´ çµگهگˆمپ§çµگهگˆمپ—م€پن؛Œوœ¬éژ–DNAم‚’ه½¢وˆگمپ™م‚‹م€‚çھ’ç´ ه،©هں؛مپ¯م€پهچکç’°مپ®مƒ”مƒھمƒںم‚¸مƒ³مپ¨ن؛Œé‡چç’°مپ®مƒ—مƒھمƒ³مپ¨مپ„مپ†2مپ¤مپ®م‚°مƒ«مƒ¼مƒ—مپ«هˆ†é،مپ•م‚Œم‚‹م€‚DNAمپ§مپ¯م€پمƒپمƒںمƒ³مپ¨م‚·مƒˆم‚·مƒ³مپŒمƒ”مƒھمƒںم‚¸مƒ³م€پم‚¢مƒ‡مƒ‹مƒ³مپ¨م‚°م‚¢مƒ‹مƒ³مپŒمƒ—مƒھمƒ³مپ§مپ‚م‚‹م€‚ ن؛Œوœ¬éژ–DNAمپ®ن¸،éژ–مپ¯هگŒن¸€مپ®ç”ں物ه¦çڑ„وƒ…ه ±م‚’ن؟هکمپ—مپ¦مپ„م‚‹م€‚مپ“مپ®وƒ…ه ±مپ¯2وœ¬مپ®éژ–مپŒهˆ†é›¢مپ™م‚‹مپ¨مپچمپ«è¤‡è£½مپ•م‚Œم‚‹م€‚DNAمپ®ه¤§éƒ¨هˆ†ï¼ˆمƒ’مƒˆمپ§مپ¯98ï¼…ن»¥ن¸ٹ)مپ¯مƒژمƒ³م‚³مƒ¼مƒ‡م‚£مƒ³م‚°مپ§مپ‚م‚ٹم€پمپ“م‚Œم‚‰مپ®éƒ¨هˆ†مپ¯م‚؟مƒ³مƒ‘م‚¯è³ھé…چهˆ—مپ®مƒ‘م‚؟مƒ¼مƒ³مپ¨مپ—مپ¦مپ¯و©ں能مپ—مپھمپ„م€‚DNAمپ®2وœ¬مپ®éژ–مپ¯ن؛’مپ„مپ«هڈچه¯¾و–¹هگ‘مپ«èµ°مپ£مپ¦مپ„م‚‹مپںم‚پم€پ逆ه¹³è،Œمپ«مپھمپ£مپ¦مپ„م‚‹م€‚مپم‚Œمپم‚Œمپ®ç³–مپ«مپ¯4種é،مپ®و ¸é…¸ه،©هں؛(مپ¾مپںمپ¯ه،©هں؛)مپ®مپ†مپ،مپ®1مپ¤مپŒçµگهگˆمپ—مپ¦مپ„م‚‹م€‚éپ؛ن¼وƒ…ه ±م‚’م‚³مƒ¼مƒ‰ï¼ˆç¬¦هڈ·هŒ–)مپ™م‚‹مپ®مپ¯م€پن¸»éژ–مپ«و²؟مپ£مپںمپ“م‚Œم‚‰4種é،مپ®و ¸é…¸ه،©هں؛مپ®é…چهˆ—مپ§مپ‚م‚‹م€‚RNA(مƒھمƒœو ¸é…¸ï¼‰éژ–مپ¯DNAéژ–م‚’鋳ه‹مپ¨مپ—مپ¦è»¢ه†™مپ¨ه‘¼مپ°م‚Œم‚‹éپژ程مپ§ن½œم‚‰م‚Œم€پمپمپ®éڑ›مپ«DNAه،©هں؛مپ¯ه¯¾ه؟œمپ™م‚‹ه،©هں؛مپ¨ن؛¤وڈ›مپ•م‚Œم‚‹مپŒم€پمƒپمƒںمƒ³ï¼ˆT)مپ®ه ´هگˆمپ¯ن¾‹ه¤–مپ§م€پRNAمپ¯م‚¦مƒ©م‚·مƒ«ï¼ˆU)مپ¨ن؛¤وڈ›مپ™م‚‹[4]م€‚مپ“م‚Œم‚‰مپ®RNAéژ–مپ¯ç؟»è¨³مپ¨ه‘¼مپ°م‚Œم‚‹éپژ程مپ§م€پéپ؛ن¼وڑ—هڈ·مپ«هں؛مپ¥مپ„مپ¦م‚؟مƒ³مƒ‘م‚¯è³ھمپ®م‚¢مƒںمƒژé…¸é…چهˆ—م‚’و±؛ه®ڑمپ™م‚‹م€‚ çœںو ¸ç´°èƒمپ§مپ¯م€پDNAمپ¯وں“色ن½“مپ¨ه‘¼مپ°م‚Œم‚‹é•·مپ„و§‹é€ ن½“مپ«çµ„ç¹”هŒ–مپ•م‚Œمپ¦مپ„م‚‹م€‚مپ“م‚Œم‚‰مپ®وں“色ن½“مپ¯م€پé€ڑه¸¸مپ®ç´°èƒهˆ†è£‚مپ®ه‰چمپ«DNA複製éپژ程مپ§è¤‡è£½مپ•م‚Œم€پمپم‚Œمپم‚Œمپ®ه¨کç´°èƒمپ«ه®Œه…¨مپھوں“色ن½“مپ®é›†هگˆم‚’وڈگن¾›مپ™م‚‹م€‚çœںو ¸ç”ں物(ه‹•ç‰©م€پو¤چ物م€پçœںèڈŒé،م€پهژںç”ںç”ں物)مپ¯DNAمپ®ه¤§éƒ¨هˆ†م‚’و ¸DNAمپ¨مپ—مپ¦ç´°èƒو ¸ه†…مپ«ن؟هکمپ—م€پن¸€éƒ¨م‚’مƒںمƒˆم‚³مƒ³مƒ‰مƒھم‚¢DNAمپ¨مپ—مپ¦مƒںمƒˆم‚³مƒ³مƒ‰مƒھم‚¢ه†…م€پمپ‚م‚‹مپ„مپ¯è‘‰ç·‘ن½“DNAمپ¨مپ—مپ¦è‘‰ç·‘ن½“ه†…مپ«ن؟هکمپ—مپ¦مپ„م‚‹[5]م€‚ه¯¾ç…§çڑ„مپ«م€پهژںو ¸ç”ں物(細èڈŒمپ¨هڈ¤ç´°èڈŒï¼‰مپ¯DNAم‚’ç´°èƒè³ھه†…مپ®ç’°çٹ¶وں“色ن½“مپ«مپ®مپ؟ن؟هکمپ—مپ¦مپ„م‚‹م€‚çœںو ¸ç”ں物مپ®وں“色ن½“ه†…مپ§مپ¯م€پمƒ’م‚¹مƒˆمƒ³مپھمپ©مپ®م‚¯مƒمƒمƒپمƒ³م‚؟مƒ³مƒ‘م‚¯è³ھمپŒDNAم‚’ه°ڈمپ•مپڈمپ¾مپ¨م‚پمپ¦çµ„ç¹”هŒ–مپ—مپ¦مپ„م‚‹م€‚مپ“م‚Œم‚‰مپ®ç·»ه¯†مپھو§‹é€ مپ¯م€پDNAمپ¨ن»–مپ®م‚؟مƒ³مƒ‘م‚¯è³ھمپ¨مپ®ç›¸ن؛’ن½œç”¨م‚’ه°ژمپچم€پDNAمپ®مپ©مپ®éƒ¨هˆ†مپŒè»¢ه†™مپ•م‚Œم‚‹مپ‹م‚’هˆ¶ه¾،مپ™م‚‹مپ®مپ«ه½¹ç«‹مپ£مپ¦مپ„م‚‹م€‚ 特و€§ DNAمپ¯مƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰مپ¨ه‘¼مپ°م‚Œم‚‹هڈچه¾©هچکن½چمپ‹م‚‰مپھم‚‹é•·مپ„مƒمƒھمƒمƒ¼مپ§مپ‚م‚‹[6][7]م€‚DNAمپ®و§‹é€ مپ¯مپمپ®é•·مپ•مپ«و²؟مپ£مپ¦ه‹•çڑ„مپ§مپ‚م‚ٹم€په¯†مپھمƒ«مƒ¼مƒ—م‚’ن½œمپ£مپںم‚ٹم€پن»–مپ®ه½¢çٹ¶مپ«ه·»مپچمپ¤مپڈمپ“مپ¨مپŒمپ§مپچم‚‹[8]م€‚مپ©مپ®ç”ں物種مپ«مپٹمپ„مپ¦م‚‚م€پDNAمپ¯و°´ç´ çµگهگˆمپ§çµگهگˆمپ—مپں2وœ¬مپ®م‚‰مپ›م‚“çٹ¶مپ®éژ–مپ§و§‹وˆگمپ•م‚Œمپ¦مپ„م‚‹م€‚ن¸،و–¹مپ®éژ–مپ¨م‚‚م€پهگŒمپک軸مپ«م‚‰مپ›م‚“çٹ¶مپ«ه·»مپ‹م‚Œم€پمƒ”مƒƒمƒپم‚‚هگŒمپکمپ§34م‚ھمƒ³م‚°م‚¹مƒˆمƒمƒ¼مƒ (3.4 nm)مپ§مپ‚م‚‹م€‚ن¸€ه¯¾مپ®éژ–مپ®هچٹه¾„مپ¯10 أ… (1.0 nm)مپ§مپ‚م‚‹[9]م€‚هˆ¥مپ®ç ”究مپ«م‚ˆم‚‹مپ¨م€پهˆ¥مپ®و؛¶و¶²ن¸مپ§و¸¬ه®ڑمپ—مپںه ´هگˆم€پDNAéژ–مپ®ه¹…مپ¯22–26 أ… (2.2–2.6 nm)م€پ1مƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰هچکن½چمپ®é•·مپ•مپ¯3.3 أ… (0.33 nm)مپ§مپ‚مپ£مپں[10]م€‚مپ»مپ¨م‚“مپ©مپ®DNAمپ®وµ®هٹ›ه¯†ه؛¦مپ¯1.7 g/cm3مپ§مپ‚م‚‹[11]م€‚ é€ڑه¸¸م€پDNAمپ¯ن¸€وœ¬مپ®éژ–مپ¨مپ—مپ¦هکهœ¨مپ™م‚‹مپ®مپ§مپ¯مپھمپڈم€پن¸€ه¯¾مپ®éژ–مپŒمپ—مپ£مپ‹م‚ٹمپ¨çµگهگˆمپ—مپ¦هکهœ¨مپ™م‚‹[9][12]م€‚مپ“مپ®2وœ¬مپ®é•·مپ„éژ–مپ¯ن؛’مپ„مپ«ه·»مپچمپ¤مپ„مپ¦ن؛Œé‡چم‚‰مپ›م‚“م‚’ه½¢وˆگمپ—مپ¦مپ„م‚‹م€‚مƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰مپ«مپ¯م€پDNAهˆ†هگمپ®ن¸»éژ–مپ®ن¸€éƒ¨ï¼ˆéژ–م‚’و§‹وˆگمپ™م‚‹ï¼‰مپ¨و ¸é…¸ه،©هں؛(م‚‰مپ›م‚“ه†…部مپ§م‚‚مپ†ن¸€و–¹مپ®DNAéژ–مپ¨ç›¸ن؛’ن½œç”¨مپ™م‚‹ï¼‰مپ®ن¸،و–¹مپŒهگ«مپ¾م‚Œمپ¦مپ„م‚‹م€‚ç³–مپ¨çµگهگˆمپ—مپںو ¸é…¸ه،©هں؛مپ¯مƒŒم‚¯مƒ¬م‚ھم‚·مƒ‰ï¼ˆè‹±: nucleoside)مپ¨ه‘¼مپ°م‚Œم€پمپ“م‚Œمپ«ه¯¾مپ—ç³–مپ¨1مپ¤ن»¥ن¸ٹمپ®مƒھمƒ³é…¸هں؛مپ¨çµگهگˆمپ—مپںه،©هں؛مپ¯مƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰ï¼ˆè‹±: nucleotide)مپ¨ه‘¼مپ°م‚Œم‚‹م€‚(DNAمپ®م‚ˆمپ†مپ«ï¼‰è¤‡و•°مپ®مƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰مپŒçµگهگˆمپ—مپںç”ںن½“é«کهˆ†هگم‚’مƒمƒھمƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰مپ¨ه‘¼مپ¶[13]م€‚ DNAéژ–مپ®ن¸»éژ–مپ¯مƒھمƒ³é…¸هں؛مپ¨ç³–هں؛مپŒن؛¤ن؛’مپ«çµگهگˆمپ—مپ¦مپ§مپچمپ¦مپ„م‚‹[14]م€‚DNAمپ®ç³–مپ¯2-مƒ‡م‚ھم‚م‚·مƒھمƒœمƒ¼م‚¹مپ§م€پمƒڑمƒ³مƒˆمƒ¼م‚¹ï¼ˆç‚ç´ و•°5م€پن؛”ç‚糖)مپ®ن¸€ç¨®مپ§مپ‚م‚‹م€‚ç³–مپ¨ç³–مپ¯م€پéڑ£وژ¥مپ™م‚‹ç³–ç’°مپ®3ن½چمپ¨5ن½چمپ®ç‚ç´ هژںهگé–“مپ«مƒ›م‚¹مƒ›م‚¸م‚¨م‚¹مƒ†مƒ«çµگهگˆم‚’ه½¢وˆگمپ™م‚‹مƒھمƒ³é…¸هں؛مپ«م‚ˆمپ£مپ¦çµگهگˆمپ—مپ¦مپ„م‚‹م€‚مپ“م‚Œم‚‰مپ®ç‚ç´ مپ¯مپم‚Œمپم‚Œم€پ3'وœ«ç«¯ï¼ˆthree prime end)م€پ5'وœ«ç«¯ï¼ˆfive prime end)مپ¨ه‘¼مپ°م‚Œم‚‹م€‚مƒ—مƒ©م‚¤مƒ è¨کهڈ·ï¼ˆ')مپ¯م€پمƒ‡م‚ھم‚م‚·مƒھمƒœمƒ¼م‚¹مپŒم‚°مƒھم‚³م‚·مƒ‰çµگهگˆم‚’ه½¢وˆگمپ™م‚‹ه،©هں؛مپ®ç‚ç´ هژںهگمپ¨هŒ؛هˆ¥مپ™م‚‹مپںم‚پمپ«ن½؟م‚ڈم‚Œم‚‹[12]م€‚ مپ“مپ®م‚ˆمپ†مپ«DNAéژ–مپ«مپ¯é€ڑه¸¸م€پمƒھمƒœمƒ¼م‚¹مپ®5'ç‚ç´ مپ«çµگهگˆمپ—مپںمƒھمƒ³é…¸هں؛(5'مƒ›م‚¹مƒ›مƒھمƒ«ï¼‰م‚’وŒپمپ¤وœ«ç«¯مپ¨م€پمƒھمƒœمƒ¼م‚¹مپ®3'ç‚ç´ مپ«çµگهگˆمپ—مپںéپٹ離مƒ’مƒ‰مƒم‚م‚·هں؛(3'مƒ’مƒ‰مƒم‚م‚·ï¼‰م‚’وŒپمپ¤وœ«ç«¯مپŒمپ‚م‚‹م€‚ç³–-مƒھمƒ³é…¸éھ¨و ¼مپ«و²؟مپ£مپں3’مپ¨5'ç‚ç´ مپ®é…چهگ‘مپ¯م€پهگ„DNAéژ–مپ«و–¹هگ‘و€§ï¼ˆو¥µو€§مپ¨م‚‚ه‘¼مپ°م‚Œم‚‹ï¼‰م‚’ن¸ژمپˆم‚‹م€‚و ¸é…¸مپ®ن؛Œé‡چم‚‰مپ›م‚“مپ§مپ¯م€پن¸€و–¹مپ®éژ–مپ®مƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰مپ®و–¹هگ‘مپ¨م‚‚مپ†ن¸€و–¹مپ®éژ–مپ®مƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰مپ®و–¹هگ‘مپ¯هڈچه¯¾مپ§م€پ逆ه¹³è،Œمپ«مپھمپ£مپ¦مپ„م‚‹م€‚DNAéژ–مپ®éه¯¾ç§°وœ«ç«¯مپ«مپ¤مپ„مپ¦مپ¯م€پ5'وœ«ç«¯و–¹هگ‘مپ¨3'وœ«ç«¯و–¹هگ‘مپ¨مپ„مپ†و–¹هگ‘و€§م‚’وœ‰مپ—م€پ5'وœ«ç«¯مپ¯مƒھمƒ³é…¸هں؛م‚’وœ‰مپ—م€پ3'وœ«ç«¯مپ¯مƒ’مƒ‰مƒم‚م‚·هں؛م‚’وœ‰مپ™م‚‹مپ¨ه‘¼مپ°م‚Œم‚‹م€‚DNAمپ¨RNAمپ®ه¤§مپچمپھéپ•مپ„مپ®ن¸€مپ¤مپ¯ç³–مپ§م€پDNAمپ®2-مƒ‡م‚ھم‚م‚·مƒھمƒœمƒ¼م‚¹مپŒRNAمپ§مپ¯مƒڑمƒ³مƒˆمƒ¼م‚¹ç³–مپ®مƒھمƒœمƒ¼م‚¹مپ«ç½®مپچوڈ›مپˆم‚‰م‚Œمپ¦مپ„م‚‹[12]م€‚  DNAن؛Œé‡چم‚‰مپ›م‚“مپ¯م€پمƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰é–“مپ®و°´ç´ çµگهگˆمپ¨م€پèٹ³é¦™و—ڈو€§و ¸é…¸ه،©هں؛é–“مپ®ه،©هں؛م‚¹م‚؟مƒƒم‚مƒ³م‚°ç›¸ن؛’ن½œç”¨مپ¨مپ„مپ†م€پن¸»مپ«2مپ¤مپ®هٹ›مپ«م‚ˆمپ£مپ¦ه®‰ه®ڑهŒ–مپ•م‚Œمپ¦مپ„م‚‹[16]م€‚DNAمپ«هگ«مپ¾م‚Œم‚‹4مپ¤مپ®ه،©هں؛مپ¯م€پم‚¢مƒ‡مƒ‹مƒ³ï¼ˆA)م€پم‚·مƒˆم‚·مƒ³ï¼ˆC)م€پم‚°م‚¢مƒ‹مƒ³ï¼ˆG)م€پمƒپمƒںمƒ³ï¼ˆT)مپ§مپ‚م‚‹م€‚مپ“م‚Œم‚‰مپ®4مپ¤مپ®ه،©هں؛مپ¯م€پم‚¢مƒ‡مƒژم‚·مƒ³ن¸€مƒھمƒ³é…¸مپ§ç¤؛مپ—مپںم‚ˆمپ†مپ«م€پç³–-مƒھمƒ³é…¸مپ«çµگهگˆمپ—مپ¦ه®Œه…¨مپھمƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰م‚’ه½¢وˆگمپ™م‚‹م€‚م‚¢مƒ‡مƒ‹مƒ³مپ¯مƒپمƒںمƒ³مپ¨ه¯¾مپ«مپھم‚ٹم€پم‚°م‚¢مƒ‹مƒ³مپ¯م‚·مƒˆم‚·مƒ³مپ¨ه¯¾مپ«مپھم‚ٹم€پمپم‚Œمپم‚Œ A-T مپ¨ G-C مپ®ه،©هں؛ه¯¾م‚’ه½¢وˆگمپ™م‚‹[17][18]م€‚ و ¸é…¸ه،©هں؛مپ®هˆ†é،و ¸é…¸ه،©هں؛مپ¯م€پ5ه“،مپٹم‚ˆمپ³6ه“،مپ®ç¸®هگˆè¤‡ç´ ç’°ه¼ڈهŒ–هگˆç‰©مپ§مپ‚م‚‹مƒ—مƒھمƒ³ A مپ¨ G مپ¨م€پ6ه“،ç’°مپ®مƒ”مƒھمƒںم‚¸مƒ³ C مپ¨ T مپ®2種é،مپ«هˆ†é،مپ•م‚Œم‚‹[12]م€‚第5مپ®مƒ”مƒھمƒںم‚¸مƒ³و ¸é…¸ه،©هں؛مپ§مپ‚م‚‹م‚¦مƒ©م‚·مƒ«ï¼ˆU)مپ¯é€ڑه¸¸م€پRNAه†…مپ§مƒپمƒںمƒ³مپ®ن»£م‚ڈم‚ٹم‚’و‹…مپ„م€پمپمپ®ç’°ن¸ٹمپ«مƒ،مƒپمƒ«هں؛م‚’وŒپمپںمپھمپ„点مپ§مƒپمƒںمƒ³مپ¨ç•°مپھم‚‹م€‚RNAمپ¨DNAمپ«هٹ مپˆمپ¦م€په¤ڑمپڈمپ®ن؛؛ه·¥و ¸é…¸é،ن¼¼ن½“مپŒو ¸é…¸مپ®ç‰¹و€§م‚’ç ”ç©¶مپ™م‚‹مپںم‚پم€پمپ‚م‚‹مپ„مپ¯مƒگم‚¤م‚ھمƒ†م‚¯مƒژمƒم‚¸مƒ¼مپ§ن½؟用مپ™م‚‹مپںم‚پمپ«ن½œوˆگمپ•م‚Œمپ¦مپچمپں[19]م€‚ éو¨™و؛–ه،©هں؛DNAمپ«مپ¯ن؟®é£¾ه،©هں؛مپŒهکهœ¨مپ™م‚‹م€‚مپ“مپ®مپ†مپ،وœ€هˆمپ«èھچèکمپ•م‚Œمپںمپ®مپ¯5-مƒ،مƒپمƒ«م‚·مƒˆم‚·مƒ³مپ§م€پ1925ه¹´مپ«م€پçµگو ¸èڈŒï¼ˆMycobacterium tuberculosis)مپ®م‚²مƒژمƒ مپ‹م‚‰ç™؛見مپ•م‚Œمپں[20]م€‚ç´°èڈŒم‚¦م‚¤مƒ«م‚¹ï¼ˆمƒگم‚¯مƒ†مƒھم‚ھمƒ•م‚،مƒ¼م‚¸ï¼‰مپ«مپ“مپ†مپ—مپںéو¨™و؛–ه،©هں؛(英: noncanonical base)مپŒهکهœ¨مپ™م‚‹çگ†ç”±مپ¯م€پç´°èڈŒمپ«هکهœ¨مپ™م‚‹هˆ¶é™گé…µç´ م‚’éپ؟مپ‘م‚‹مپںم‚پمپ§مپ‚م‚‹م€‚مپ“مپ®é…µç´ ç³»مپ¯م€په°‘مپھمپڈمپ¨م‚‚部هˆ†çڑ„مپ«مپ¯م€پç´°èڈŒم‚’م‚¦م‚¤مƒ«م‚¹و„ںوں“مپ‹م‚‰ن؟è·مپ™م‚‹هˆ†هگه…چç–«ç³»مپ¨مپ—مپ¦هƒچمپڈ[21]م€‚م‚ˆم‚ٹن¸€èˆ¬çڑ„مپھن؟®é£¾DNAه،©هں؛مپ§مپ‚م‚‹م‚·مƒˆم‚·مƒ³مپ¨م‚¢مƒ‡مƒ‹مƒ³مپ®ن؟®é£¾مپ¯م€په‹•و¤چ物مپ«مپٹمپ‘م‚‹éپ؛ن¼هگç™؛çڈ¾مپ®م‚¨مƒ”م‚¸م‚§مƒچمƒ†م‚£مƒƒم‚¯هˆ¶ه¾،(ه¾Œوˆگçڑ„èھ؟و•´ï¼‰مپ«مپٹمپ„مپ¦م€پé‡چè¦پمپھه½¹ه‰²م‚’وœمپںمپ—مپ¦مپ„م‚‹[22]م€‚ DNAمپ«مپ¯ه¤ڑمپڈمپ®éو¨™و؛–ه،©هں؛مپŒهکهœ¨مپ™م‚‹مپ“مپ¨مپŒçں¥م‚‰م‚Œمپ¦مپ„م‚‹[23]م€‚مپ“م‚Œم‚‰مپ®مپ»مپ¨م‚“مپ©مپ¯م€پم‚¦مƒ©م‚·مƒ«م‚’هگ«م‚€و¨™و؛–ه،©هں؛(英: canonical base)مپŒن؟®é£¾مپ•م‚Œمپںم‚‚مپ®مپ§مپ‚م‚‹م€‚
ن¸»و؛مپ¨ه‰¯و؛ ن؛Œوœ¬مپ®م‚‰مپ›م‚“éژ–مپŒDNAمپ®ن¸»éژ–م‚’ه½¢وˆگمپ—مپ¦مپ„م‚‹م€‚م‚‚مپ†ن¸€مپ¤مپ®ن؛Œé‡چم‚‰مپ›م‚“مپŒم€پمپمپ®éژ–مپ¨éژ–مپ®é–“مپ«مپ‚م‚‹ç©؛éڑ™م€پمپ‚م‚‹مپ„مپ¯و؛م‚’مپںمپ©مپ£مپ¦è¦‹مپ„مپ مپ•م‚Œم‚‹م€‚مپ“م‚Œم‚‰مپ®ç©؛éڑ™مپ¯ه،©هں؛ه¯¾مپ«éڑ£وژ¥مپ—مپ¦مپٹم‚ٹم€پçµگهگˆéƒ¨ن½چمپ¨مپھم‚‹هڈ¯èƒ½و€§مپŒمپ‚م‚‹م€‚éژ–مپ¯ن؛’مپ„مپ«ه¯¾ç§°مپ«é…چç½®مپ•م‚Œمپ¦مپ„مپھمپ„مپںم‚پم€پو؛مپ®ه¤§مپچمپ•مپ¯ن¸چه‡ç‰مپ§مپ‚م‚‹م€‚ن¸»و؛(مپ—م‚…مپ“مپ†ï¼‰مپ®ه¹…مپ¯22م‚ھمƒ³م‚°م‚¹مƒˆمƒمƒ¼مƒ (2.2 nm)مپ§م€په‰¯و؛(مپµمپڈمپ“مپ†ï¼‰مپ®ه¹…مپ¯12 أ… (1.2 nm)مپ§مپ‚م‚‹[24]م€‚ن¸»و؛مپ®و–¹مپŒه¹…مپŒه؛ƒمپ„مپںم‚پم€په،©هں؛مپ®ç«¯مپ¯ه‰¯و؛م‚ˆم‚ٹم‚‚ن¸»و؛مپ®و–¹مپŒè؟‘مپ¥مپچم‚„مپ™مپ„م€‚مپمپ®çµگوœم€پن؛Œوœ¬éژ–DNAمپ®ç‰¹ç•°çڑ„é…چهˆ—مپ«çµگهگˆمپ§مپچم‚‹è»¢ه†™ه› هگمپھمپ©مپ®م‚؟مƒ³مƒ‘م‚¯è³ھمپ¯م€پé€ڑه¸¸م€پن¸»و؛مپ«éœ²ه‡؛مپ—مپںه،©هں؛مپ®هپ´é¢مپ«وژ¥è§¦مپ™م‚‹ه‚¾هگ‘مپŒمپ‚م‚‹[25]م€‚مپ“مپ®م‚ˆمپ†مپھçٹ¶و³پمپ¯ç´°èƒه†…مپ®DNAمپ®ç•°ه¸¸مپھم‚³مƒ³مƒ›مƒ،مƒ¼م‚·مƒ§مƒ³ï¼ˆç«‹ن½“é…چه؛§ï¼‰مپ«م‚ˆمپ£مپ¦ç•°مپھم‚‹مپŒم€پن¸»و؛مپ¨ه‰¯و؛مپ¯DNAم‚’é€ڑه¸¸مپ®Bه‹مپ«ه·»مپچوˆ»مپ—مپںه ´هگˆمپ«è¦‹م‚‰م‚Œم‚‹ه¹…مپ®éپ•مپ„م‚’هڈچوک مپ™م‚‹م‚ˆمپ†ه¸¸مپ«ه‘½هگچمپ•م‚Œمپ¦مپ„م‚‹م€‚ ه،©هں؛ه¯¾هگˆâ†’詳細مپ¯م€Œه،©هں؛ه¯¾م€چم‚’هڈ‚ç…§
(ن¸ٹ) 3مپ¤مپ®و°´ç´ çµگهگˆم‚’وŒپمپ¤GCه،©هں؛ه¯¾م€‚(ن¸‹) 2مپ¤مپ®و°´ç´ çµگهگˆم‚’وŒپمپ¤ATه،©هں؛ه¯¾م€‚ç ´ç·ڑمپ¯ه،©هں؛ه¯¾é–“مپ®éه…±وœ‰و°´ç´ çµگهگˆم‚’ç¤؛مپ™م€‚ DNAمپ®ن؛Œé‡چم‚‰مپ›م‚“مپ§مپ¯م€پن¸€و–¹مپ®éژ–ن¸ٹمپ«مپ‚م‚‹مپم‚Œمپم‚Œمپ®و ¸é…¸ه،©هں؛مپŒم€پم‚‚مپ†ن¸€و–¹مپ®éژ–ن¸ٹمپ®مپںمپ ن¸€ç¨®é،مپ®و ¸é…¸ه،©هں؛مپ¨çµگهگˆمپ™م‚‹م€‚مپ“م‚Œمپ¯ç›¸è£œçڑ„ه،©هں؛ه¯¾ه½¢وˆگ(英: complementary base pairing)مپ¨ه‘¼مپ°م‚Œم‚‹م€‚مƒ—مƒھمƒ³مپ¨مƒ”مƒھمƒںم‚¸مƒ³مپ¯ه¯¾هگˆمپ—مپ¦و°´ç´ çµگهگˆم‚’ه½¢وˆگمپ—م€پم‚¢مƒ‡مƒ‹مƒ³مپ¨مƒپمƒںمƒ³مپ¯2وœ¬م€پم‚·مƒˆم‚·مƒ³مپ¨م‚°م‚¢مƒ‹مƒ³مپ¯3وœ¬مپ®و°´ç´ çµگهگˆم‚’ه½¢وˆگمپ™م‚‹م€‚مپ“مپ®م‚ˆمپ†مپ«م€پن؛Œé‡چم‚‰مپ›م‚“م‚’وŒںم‚“مپ§ï¼ˆ6ç‚ç´ ç’°مپ‹م‚‰6ç‚ç´ ç’°مپ¸ï¼‰2مپ¤مپ®مƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰مپŒçµگهگˆه¯¾م‚’ه½¢وˆگمپ™م‚‹é…چç½®مپ¯م€پمƒ¯مƒˆم‚½مƒ³مƒ»م‚¯مƒھمƒƒم‚¯ه،©هں؛ه¯¾مپ¨ه‘¼مپ°م‚Œم‚‹م€‚GCهگ«é‡ڈمپ®é«کمپ„DNAمپ¯GCهگ«é‡ڈمپ®ن½ژمپ„DNAم‚ˆم‚ٹم‚‚ه®‰ه®ڑمپ§مپ‚م‚‹م€‚مƒ•مƒ¼م‚°م‚¹مƒ†م‚£مƒ¼مƒ³ه،©هں؛ه¯¾ï¼ˆè‹±: Hoogsteen base pairم€پ6ç‚ç´ ç’°مپ¨5ç‚ç´ ç’°مپ®و°´ç´ çµگهگˆï¼‰مپ¯م€په،©هں؛ه¯¾ه½¢وˆگمپ®مپ¾م‚Œمپھه¤‰ç¨®مپ§مپ‚م‚‹[26]م€‚ه…±وœ‰çµگهگˆمپ¨ç•°مپھم‚ٹم€پو°´ç´ çµگهگˆمپ¯و¯”較çڑ„ç°،هچکمپ«هˆ‡و–مپ—مپںم‚ٹه†چçµگهگˆمپ—مپںم‚ٹمپ™م‚‹مپ“مپ¨مپŒمپ§مپچم‚‹م€‚مپمپ®مپںم‚پن؛Œé‡چم‚‰مپ›م‚“م‚’و§‹وˆگمپ™م‚‹DNAمپ®ن؛Œوœ¬éژ–مپ¯م€پو©ںو¢°çڑ„مپھهٹ›م‚„é«کو¸©مپ«م‚ˆمپ£مپ¦مƒ•م‚،م‚¹مƒٹمƒ¼مپ®م‚ˆمپ†مپ«ه¼•مپچ離مپ™مپ“مپ¨مپŒمپ§مپچم‚‹[27]م€‚مپ“مپ®ه،©هں؛ه¯¾مپ®ç›¸è£œو€§مپ®çµگوœم€پDNAم‚‰مپ›م‚“مپ®ن؛Œوœ¬éژ–é…چهˆ—مپ®مپ™مپ¹مپ¦مپ®وƒ…ه ±مپŒمپم‚Œمپم‚Œمپ®éژ–مپ«è¤‡è£½مپ•م‚Œم€پمپ“م‚Œمپ¯DNA複製مپ«ن¸چهڈ¯و¬ مپ§مپ‚م‚‹م€‚相補çڑ„مپھه،©هں؛ه¯¾é–“مپ®مپ“مپ®هڈ¯é€†çڑ„مپ§ç‰¹ç•°çڑ„مپھ相ن؛’ن½œç”¨مپ¯م€پç”ں物مپ«مپٹمپ‘م‚‹DNAمپ®مپ™مپ¹مپ¦مپ®و©ں能مپ«مپ¨مپ£مپ¦é‡چè¦پمپ§مپ‚م‚‹[7]م€‚ ssDNAمپ¨dsDNAن¸ٹè؟°مپ—مپںم‚ˆمپ†مپ«م€پمپ»مپ¨م‚“مپ©مپ®DNAهˆ†هگمپ¯ه®ںéڑ›مپ«مپ¯2وœ¬مپ®مƒمƒھمƒمƒ¼éژ–مپ§مپ‚م‚ٹم€پéه…±وœ‰çµگهگˆمپ«م‚ˆمپ£مپ¦م‚‰مپ›م‚“çٹ¶مپ«çµگهگˆمپ—مپ¦مپ„م‚‹م€‚مپ“مپ®ن؛Œوœ¬éژ–DNAو§‹é€ (英: double-stranded DNAم€پdsDNA)مپ¯م€پن¸»مپ«éژ–ه†…ه،©هں؛م‚¹م‚؟مƒƒم‚مƒ³م‚°ç›¸ن؛’ن½œç”¨ï¼ˆG,Cم‚¹م‚؟مƒƒم‚¯مپŒوœ€م‚‚ه¼·مپ„)مپ«م‚ˆمپ£مپ¦ç¶وŒپمپ•م‚Œمپ¦مپ„م‚‹م€‚مپ“مپ®2وœ¬مپ®éژ–مپ¯م€پèچ解(melting)مپ¨ه‘¼مپ°م‚Œم‚‹éپژ程م‚’経مپ¦هˆ†é›¢مپ—م€پ2وœ¬مپ®ن¸€وœ¬éژ–DNAهˆ†هگ(英: single-stranded DNAم€پssDNA)م‚’ه½¢وˆگمپ™م‚‹مپ“مپ¨مپŒمپ‚م‚‹م€‚èچ解مپ¯م€پé«کو¸©م€پن½ژه،©م€پé«کpHمپ®و،ن»¶ن¸‹مپ§èµ·مپ“م‚‹ï¼ˆن½ژpHم‚‚DNAم‚’èچ解مپ•مپ›م‚‹مپŒم€پDNAمپ¯é…¸-脱مƒ—مƒھمƒ³هŒ–مپ«م‚ˆم‚ٹن¸چه®‰ه®ڑمپ§مپ‚م‚‹مپںم‚پم€پن½ژpHمپ¯مپ»مپ¨م‚“مپ©è،Œم‚ڈم‚Œمپھمپ„)م€‚ dsDNAه‹مپ®ه®‰ه®ڑو€§مپ¯م€پGCهگ«وœ‰ï¼ˆG,Cه،©هں؛ه¯¾مپ®ه‰²هگˆï¼‰مپ مپ‘مپ§مپھمپڈم€پé…چهˆ—(م‚¹م‚؟مƒƒم‚مƒ³م‚°مپ¯é…چهˆ—特異çڑ„مپ§مپ‚م‚‹مپںم‚پ)مپٹم‚ˆمپ³é•·مپ•ï¼ˆهˆ†هگمپŒé•·مپ„مپ»مپ©ه®‰ه®ڑ)مپ«م‚‚ن¾هکمپ™م‚‹م€‚ه®‰ه®ڑو€§مپ¯مپ•مپ¾مپ–مپ¾مپھو–¹و³•مپ§و¸¬ه®ڑمپ§مپچم‚‹م€‚ن¸€èˆ¬çڑ„مپھو–¹و³•مپ¯èچ解و¸©ه؛¦ï¼ˆTmه€¤مپ¨م‚‚ه‘¼مپ°م‚Œم‚‹ï¼‰مپ§مپ‚م‚ٹم€پن؛Œوœ¬éژ–هˆ†هگمپ®50%مپŒن¸€وœ¬éژ–هˆ†هگمپ«ه¤‰وڈ›مپ•م‚Œم‚‹و¸©ه؛¦مپ§مپ‚م‚‹م€‚èچ解و¸©ه؛¦مپ¯DNAمپ®م‚¤م‚ھمƒ³ه¼·ه؛¦مپ¨و؟ƒه؛¦مپ«ن¾هکمپ™م‚‹م€‚مپ—مپںمپŒمپ£مپ¦م€پGCه،©هں؛ه¯¾مپ®ه‰²هگˆمپ¨DNAن؛Œé‡چم‚‰مپ›م‚“مپ®ه…¨é•·مپ®ن¸،و–¹مپŒم€پDNAمپ®ن؛Œوœ¬éژ–é–“مپ®çµگهگˆمپ®ه¼·مپ•م‚’و±؛ه®ڑمپ™م‚‹م€‚GCهگ«é‡ڈمپŒé«کمپڈé•·مپ„DNAم‚‰مپ›م‚“مپ¯ç›¸ن؛’ن½œç”¨مپŒه¼·مپ„éژ–مپŒه¤ڑمپڈم€پATهگ«é‡ڈمپŒé«کمپڈçںمپ„DNAم‚‰مپ›م‚“مپ¯ç›¸ن؛’ن½œç”¨مپŒه¼±مپ„éژ–مپŒه¤ڑمپ„[28]م€‚ç”ں物ه¦مپ§مپ¯م€پDNAن؛Œé‡چم‚‰مپ›م‚“مپ®مپ†مپ،هˆ†é›¢مپ—م‚„مپ™مپ„部هˆ†م€پمپںمپ¨مپˆمپ°ن¸€éƒ¨مپ®مƒ—مƒمƒ¢مƒ¼م‚؟مƒ¼مپ«هگ«مپ¾م‚Œم‚‹ TATAAT مƒ—مƒھمƒ–مƒژمƒ¼مƒ»مƒœمƒƒم‚¯م‚¹مپھمپ©مپ¯م€پéژ–م‚’ه¼•مپچ離مپ—م‚„مپ™مپڈمپ™م‚‹مپںم‚پمپ«ATهگ«é‡ڈمپŒé«کمپڈمپھم‚‹ه‚¾هگ‘مپŒمپ‚م‚‹[29]م€‚ ه®ں験ه®¤مپ§مپ¯م€پو°´ç´ çµگهگˆمپ®هچٹهˆ†م‚’هˆ‡و–مپ™م‚‹مپ®مپ«ه؟…è¦پمپھèچ解و¸©ه؛¦Tmم‚’و±‚م‚پم‚‹مپ“مپ¨مپ«م‚ˆم‚ٹم€پمپ“مپ®ç›¸ن؛’ن½œç”¨مپ®ه¼·مپ•م‚’و¸¬ه®ڑمپ™م‚‹مپ“مپ¨مپŒمپ§مپچم‚‹م€‚DNAن؛Œé‡چم‚‰مپ›م‚“ه†…مپ®ه،©هں؛ه¯¾مپŒمپ™مپ¹مپ¦èچ解مپ™م‚‹مپ¨éژ–مپ¯هˆ†é›¢مپ—م€پو؛¶و¶²ن¸مپ«ه®Œه…¨مپ«ç‹¬ç«‹مپ—مپں2مپ¤مپ®هˆ†هگمپ¨مپ—مپ¦هکهœ¨مپ™م‚‹م€‚مپ“م‚Œم‚‰مپ®ن¸€وœ¬éژ–DNAهˆ†هگمپ«مپ¯هچکن¸€مپ®ه…±é€ڑه½¢çٹ¶مپ¯هکهœ¨مپ—مپھمپ„مپŒم€پمپ„مپڈمپ¤مپ‹مپ®م‚³مƒ³مƒ›مƒ،مƒ¼م‚·مƒ§مƒ³مپ¯ن»–مپ®م‚‚مپ®م‚ˆم‚ٹم‚‚ه®‰ه®ڑمپ—مپ¦مپ„م‚‹[30]م€‚ هگ«وœ‰é‡ڈ →詳細مپ¯م€Œو ¸ه‹م€چم‚’هڈ‚ç…§ مƒ’مƒˆمپ®ه ´هگˆم€پç´°èƒ1ه€‹مپ‚مپںم‚ٹم€په¥³و€§مپ®ن؛Œه€چن½“و ¸م‚²مƒژمƒ مپ®ç·ڈé•·مپ¯6.37م‚®م‚¬ه،©هں؛ه¯¾ï¼ˆGbp)مپ«هڈٹمپ³م€پé•·مپ•مپ¯208.23 cmم€پè³ھé‡ڈمپ¯6.51 pgمپ§مپ‚م‚‹[31]م€‚ç”·و€§مپ®ه€¤مپ¯مپم‚Œمپم‚Œم€پ6.27 Gbpم€پ205.00 cmم€پ6.41 pgمپ§مپ‚م‚‹[31]م€‚هگ„DNAمƒمƒھمƒمƒ¼مپ¯م€پ1ç•ھوں“色ن½“مپ®م‚ˆمپ†مپ«و•°ه„„م‚‚مپ®مƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰م‚’هگ«م‚€مپ“مپ¨مپŒمپ‚م‚‹م€‚1ç•ھوں“色ن½“مپ¯ç´„2ه„„2هچƒن¸‡ه،©هں؛ه¯¾مپ‹م‚‰مپھم‚‹مƒ’مƒˆوœ€ه¤§مپ®وں“色ن½“مپ§م€پمپ¾مپ£مپ™مپگمپ«ن¼¸مپ°مپ™مپ¨85 mmمپ®é•·مپ•مپ«مپھم‚‹[32]م€‚ çœںو ¸ç”ں物مپ«مپ¯م€پو ¸DNAمپ®مپ»مپ‹مپ«مƒںمƒˆم‚³مƒ³مƒ‰مƒھم‚¢DNA(mtDNA)م‚‚مپ‚م‚ٹم€پمƒںمƒˆم‚³مƒ³مƒ‰مƒھم‚¢مپ§ن½؟م‚ڈم‚Œم‚‹ç‰¹ه®ڑمپ®م‚؟مƒ³مƒ‘م‚¯è³ھم‚’م‚³مƒ¼مƒ‰مپ—مپ¦مپ„م‚‹م€‚mtDNAمپ¯é€ڑه¸¸م€پو ¸DNAمپ«و¯”مپ¹مپ¦و¯”較çڑ„ه°ڈمپ•مپ„م€‚مپںمپ¨مپˆمپ°م€پمƒ’مƒˆمپ®مƒںمƒˆم‚³مƒ³مƒ‰مƒھم‚¢DNAمپ¯é–‰مپکمپںç’°çٹ¶هˆ†هگم‚’ه½¢وˆگمپ—م€پمپم‚Œمپم‚Œمپ®هˆ†هگمپ¯16,569ه€‹[33][34]مپ®DNAه،©هں؛ه¯¾م‚’هگ«مپ؟[35]م€پمپمپ†مپ—مپںهگ„هˆ†هگمپ«مپ¯é€ڑه¸¸م€پمƒںمƒˆم‚³مƒ³مƒ‰مƒھم‚¢éپ؛ن¼هگمپ®ه®Œه…¨مپھ集هگˆمپŒهگ«مپ¾م‚Œم‚‹م€‚مƒ’مƒˆمپ®هگ„مƒںمƒˆم‚³مƒ³مƒ‰مƒھم‚¢مپ«مپ¯م€پمپ“مپ®م‚ˆمپ†مپھmtDNAهˆ†هگمپŒه¹³ه‡مپ—مپ¦ç´„5ه€‹هگ«مپ¾م‚Œمپ¦مپ„م‚‹[35]م€‚هگ„مƒ’مƒˆç´°èƒمپ¯ç´„100ه€‹مپ®مƒںمƒˆم‚³مƒ³مƒ‰مƒھم‚¢م‚’هگ«م‚€مپ®مپ§م€پمƒ’مƒˆç´°èƒمپ‚مپںم‚ٹمپ®mtDNAهˆ†هگمپ®ç·ڈو•°مپ¯ç´„500ه€‹مپ¨مپھم‚‹[35]م€‚مپںمپ مپ—م€پç´°èƒمپ‚مپںم‚ٹمپ®مƒںمƒˆم‚³مƒ³مƒ‰مƒھم‚¢مپ®é‡ڈم‚‚ç´°èƒمپ®ç¨®é،مپ«م‚ˆمپ£مپ¦ç•°مپھم‚ٹم€پهچµç´°èƒمپ«مپ¯10ن¸‡ه€‹مپ®مƒںمƒˆم‚³مƒ³مƒ‰مƒھم‚¢مپŒهگ«مپ¾م‚Œم‚‹مپ“مپ¨مپŒمپ‚م‚ٹم€پمƒںمƒˆم‚³مƒ³مƒ‰مƒھم‚¢م‚²مƒژمƒ (細èƒمپ®DNAمپ®وœ€ه¤§90ï¼…م‚’و§‹وˆگمپ™م‚‹ï¼‰مپ®وœ€ه¤§150ن¸‡م‚³مƒ”مƒ¼مپ«ç›¸ه½“مپ™م‚‹[36]م€‚ م‚»مƒ³م‚¹مپ¨م‚¢مƒ³مƒپم‚»مƒ³م‚¹â†’詳細مپ¯م€Œم‚»مƒ³م‚¹ (هˆ†هگç”ں物ه¦)م€چم‚’هڈ‚ç…§
مپ‚م‚‹DNAé…چهˆ—مپŒم€پم‚؟مƒ³مƒ‘م‚¯è³ھمپ«ç؟»è¨³مپ•م‚Œم‚‹مƒ،مƒƒم‚»مƒ³م‚¸مƒ£مƒ¼RNAمپ®م‚³مƒ”مƒ¼مپ¨هگŒمپکمپ§مپ‚م‚‹ه ´هگˆم€پم€Œم‚»مƒ³م‚¹é…چهˆ—م€چ(英: sense sequence)مپ¨ه‘¼مپ°م‚Œم‚‹[37]م€‚هڈچه¯¾هپ´مپ®éژ–مپ®é…چهˆ—مپ¯م€Œم‚¢مƒ³مƒپم‚»مƒ³م‚¹é…چهˆ—م€چ(英: antisense sequence)مپ¨ه‘¼مپ°م‚Œم‚‹م€‚م‚»مƒ³م‚¹é…چهˆ—مپ¨م‚¢مƒ³مƒپم‚»مƒ³م‚¹é…چهˆ—مپ¯م€پهگŒمپکDNAéژ–مپ®ç•°مپھم‚‹éƒ¨هˆ†مپ«هکهœ¨مپ™م‚‹مپ“مپ¨مپŒمپ‚م‚‹ï¼ˆمپ™مپھم‚ڈمپ،م€پن¸،و–¹مپ®éژ–مپŒم‚»مƒ³م‚¹é…چهˆ—مپ¨م‚¢مƒ³مƒپم‚»مƒ³م‚¹é…چهˆ—مپ®ن¸،و–¹م‚’هگ«م‚€ï¼‰م€‚هژںو ¸ç”ں物مپ§م‚‚çœںو ¸ç”ں物مپ§م‚‚م‚¢مƒ³مƒپم‚»مƒ³م‚¹RNAé…چهˆ—مپŒن½œم‚‰م‚Œم‚‹مپŒم€پمپ“م‚Œم‚‰مپ®RNAمپ®و©ں能مپ¯ه®Œه…¨مپ«مپ¯è§£وکژمپ•م‚Œمپ¦مپ„مپھمپ„[38]م€‚ن¸€مپ¤مپ®وڈگو،ˆمپ¯م€پم‚¢مƒ³مƒپم‚»مƒ³م‚¹RNAمپŒRNA-RNAه،©هں؛ه¯¾ه½¢وˆگم‚’é€ڑمپکمپ¦éپ؛ن¼هگç™؛çڈ¾مپ®èھ؟節مپ«é–¢ن¸ژمپ—مپ¦مپ„م‚‹مپ¨مپ„مپ†م‚‚مپ®مپ§مپ‚م‚‹[39]م€‚ هژںو ¸ç”ں物م‚„çœںو ¸ç”ں物مپ®DNAé…چهˆ—م€پمپمپ—مپ¦مƒ—مƒ©م‚¹مƒںمƒ‰م‚„م‚¦م‚¤مƒ«م‚¹مپ§مپ¯م‚ˆم‚ٹه¤ڑمپڈمپ®DNAé…چهˆ—مپŒم€پم‚ھمƒ¼مƒگمƒ¼مƒ©مƒƒمƒ—éپ؛ن¼هگ(英: overlapping gene)م‚’وŒپمپ¤مپ“مپ¨مپ«م‚ˆمپ£مپ¦م‚»مƒ³م‚¹éژ–مپ¨م‚¢مƒ³مƒپم‚»مƒ³م‚¹éژ–مپ®هŒ؛هˆ¥م‚’مپ‚مپ„مپ¾مپ„مپ«مپ—مپ¦مپ„م‚‹[40]م€‚مپ“مپ®م‚ˆمپ†مپھه ´هگˆم€پDNAé…چهˆ—مپ®ن¸مپ«مپ¯م€پن¸€و–¹مپ®éژ–مپ«و²؟مپ£مپ¦èھمپ¾م‚Œم‚‹مپ¨ن¸€و–¹مپ®م‚؟مƒ³مƒ‘م‚¯è³ھم‚’م‚³مƒ¼مƒ‰مپ—م€پم‚‚مپ†ن¸€و–¹مپ®éژ–مپ«و²؟مپ£مپ¦é€†و–¹هگ‘مپ«èھمپ¾م‚Œم‚‹مپ¨م‚‚مپ†ن¸€و–¹مپ®م‚؟مƒ³مƒ‘م‚¯è³ھم‚’م‚³مƒ¼مƒ‰مپ™م‚‹مپ¨مپ„مپ†م€پن؛Œé‡چمپ®ه½¹ه‰²م‚’وœمپںمپ™م‚‚مپ®مپŒمپ‚م‚‹م€‚ç´°èڈŒمپ§مپ¯م€پمپ“مپ®é‡چ畳مپŒéپ؛ن¼هگ転ه†™مپ®èھ؟節مپ«é–¢ن¸ژمپ—مپ¦مپ„م‚‹هڈ¯èƒ½و€§مپŒمپ‚م‚‹[41]م€‚ن¸€و–¹م€پم‚¦م‚¤مƒ«م‚¹مپ§مپ¯م€پم‚ھمƒ¼مƒگمƒ¼مƒ©مƒƒمƒ—éپ؛ن¼هگمپ«م‚ˆمپ£مپ¦م€په°ڈمپ•مپھم‚¦م‚¤مƒ«م‚¹م‚²مƒژمƒ ه†…مپ«م‚³مƒ¼مƒ‰مپ§مپچم‚‹وƒ…ه ±é‡ڈم‚’ه¢—هٹ مپ•مپ›م‚‹[42]م€‚ م‚¹مƒ¼مƒ‘مƒ¼م‚³م‚¤مƒ«â†’詳細مپ¯م€ŒDNA超م‚‰مپ›م‚“م€چم‚’هڈ‚ç…§
DNAمپ¯م€پDNAم‚¹مƒ¼مƒ‘مƒ¼م‚³م‚¤مƒ«ï¼ˆè‹±: DNA supercoilingم€پDNA超م‚‰مپ›م‚“)مپ¨ه‘¼مپ°م‚Œم‚‹éپژ程مپ§م€پمƒمƒ¼مƒ—مپ®م‚ˆمپ†مپ«مپمپکم‚Œم‚‹مپ“مپ¨مپŒمپ‚م‚‹م€‚DNAمپŒم€Œه¼›ç·©مپ—مپںم€چçٹ¶و…‹مپ§مپ¯م€پéژ–مپ¯é€ڑه¸¸10.4ه،©هں؛ه¯¾مپ”مپ¨مپ«ن؛Œé‡چم‚‰مپ›م‚“مپ®è»¸مپ®ه‘¨م‚ٹم‚’ن¸€ه‘¨مپ™م‚‹مپŒم€پDNAمپŒمپمپکم‚Œم‚‹مپ¨éژ–مپ¯م‚ˆم‚ٹمپچمپ¤مپڈم€پمپ‚م‚‹مپ„مپ¯م‚ˆم‚ٹç·©مپڈه·»مپ‹م‚Œم‚‹[43]م€‚DNAمپŒم‚‰مپ›م‚“مپ®و–¹هگ‘مپ«مپمپکم‚Œمپ¦مپ„م‚‹ه ´هگˆم€پمپ“م‚Œمپ¯و£مپ®م‚¹مƒ¼مƒ‘مƒ¼م‚³م‚¤مƒ«مپ¨ه‘¼مپ°م‚Œم€په،©هں؛هگŒه£«مپ¯م‚ˆم‚ٹè؟‘مپڈمپ«é…چç½®مپ•م‚Œم‚‹م€‚م‚‚مپ—هڈچه¯¾و–¹هگ‘مپ«مپمپکم‚Œمپ¦مپ„م‚‹مپھم‚‰م€پمپ“م‚Œمپ¯è² مپ®م‚¹مƒ¼مƒ‘مƒ¼م‚³م‚¤مƒ«مپ¨ه‘¼مپ°م‚Œم€په،©هں؛هگŒه£«مپ¯م‚ˆم‚ٹ離م‚Œم‚„مپ™مپڈمپھم‚‹م€‚è‡ھ然界مپ§مپ¯م€پمپ»مپ¨م‚“مپ©مپ®DNAمپ¯م€پمƒˆمƒم‚¤م‚½مƒ،مƒ©مƒ¼م‚¼مپ¨ه‘¼مپ°م‚Œم‚‹é…µç´ مپ«م‚ˆمپ£مپ¦ه°ژه…¥مپ•م‚Œم‚‹م€پم‚ڈمپڑمپ‹مپ«è² مپ®م‚¹مƒ¼مƒ‘مƒ¼م‚³م‚¤مƒ«م‚’وŒپمپ£مپ¦مپ„م‚‹[44]م€‚مپ“م‚Œم‚‰مپ®é…µç´ مپ¯م€پ転ه†™م‚„DNA複製مپھمپ©مپ®éپژ程مپ§DNAéژ–مپ«ç”ںمپکم‚‹مپمپکم‚Œه؟œهٹ›م‚’ç·©ه’Œمپ™م‚‹مپںم‚پمپ«م‚‚ه؟…è¦پمپ§مپ‚م‚‹[45]م€‚ ن»£و›؟DNAو§‹é€â†’詳細مپ¯م€Œو ¸é…¸مپ®هˆ†هگو§‹é€ : مƒ‡م‚ھم‚م‚·مƒھمƒœو ¸é…¸مپ®و§‹é€ م€چم€پم€ŒDNAمپ®هˆ†هگمƒ¢مƒ‡مƒ«م€چم€پمپٹم‚ˆمپ³م€Œو ¸é…¸و§‹é€ م€چم‚’هڈ‚ç…§
 DNAمپ¯م€پA-DNA(Aه‹DNA)م€پB-DNA(Bه‹DNA)م€پZ-DNA(Zه‹DNA)مپھمپ©مپ®ه¤ڑمپڈمپ®èµ·مپ“م‚ٹمپ†م‚‹م‚³مƒ³مƒ›مƒ،مƒ¼م‚·مƒ§مƒ³مپ§هکهœ¨مپ™م‚‹مپŒم€پو©ں能çڑ„مپھç”ں物مپ§ç›´وژ¥è¦³ه¯ںمپ•م‚Œمپ¦مپ„م‚‹مپ®مپ¯B-DNAمپ¨Z-DNAمپ«é™گم‚‰م‚Œم‚‹[14]م€‚DNAمپŒهڈ–م‚‹م‚³مƒ³مƒ›مƒ،مƒ¼م‚·مƒ§مƒ³مپ¯م€پو°´ه’Œمƒ¬مƒ™مƒ«م€پDNAé…چهˆ—م€پم‚¹مƒ¼مƒ‘مƒ¼م‚³م‚¤مƒ«مپ®é‡ڈمپ¨و–¹هگ‘م€په،©هں؛مپ®هŒ–ه¦ن؟®é£¾م€پ金ه±م‚¤م‚ھمƒ³مپ®ç¨®é،مپ¨و؟ƒه؛¦م€پو؛¶و¶²ن¸مپ®مƒمƒھم‚¢مƒںمƒ³مپ®وœ‰ç„،مپ«ن¾هکمپ™م‚‹[46]م€‚ A-DNAم€پمپٹم‚ˆمپ³B-DNAمپ®Xç·ڑه›وٹکمƒ‘م‚؟مƒ¼مƒ³مپ«مپ¤مپ„مپ¦وœ€هˆمپ«ç™؛è،¨مپ•م‚Œمپںه ±ه‘ٹمپ§مپ¯م€پمƒ‘م‚؟مƒ¼م‚½مƒ³é–¢و•°مپ«هں؛مپ¥مپڈ解وگمپŒن½؟用مپ•م‚Œم€پDNAمپ®é…چهگ‘ç¹ٹç¶مپ«é™گم‚‰م‚Œمپںو§‹é€ وƒ…ه ±مپ—مپ‹ه¾—م‚‰م‚Œمپھمپ‹مپ£مپں[47][48]م€‚1953ه¹´م€پم‚¦م‚£مƒ«م‚مƒ³م‚¹م‚‰مپ«م‚ˆمپ£مپ¦م€پé«کو°´ه’ŒDNAç¹ٹç¶مپ® in vivo(ç”ںن½“ه†…)B-DNA Xç·ڑه›وٹکو•£ن¹±مƒ‘م‚؟مƒ¼مƒ³مپ«مپ¤مپ„مپ¦م€پمƒ™مƒƒم‚»مƒ«é–¢و•°مپ®2ن¹—مپ¨مپ„مپ†è¦³ç‚¹مپ‹م‚‰هˆ¥مپ®è§£وگو³•مپŒوڈگو،ˆمپ•م‚Œمپں[49]م€‚هگŒمپکم‚¸مƒ£مƒ¼مƒٹمƒ«مپ§م€پم‚¸م‚§مƒ¼مƒ م‚؛مƒ»مƒ¯مƒˆم‚½مƒ³مپ¨مƒ•مƒ©مƒ³م‚·م‚¹مƒ»م‚¯مƒھمƒƒم‚¯مپŒم€پDNAمپ®Xç·ڑه›وٹکمƒ‘م‚؟مƒ¼مƒ³مپ®هˆ†هگمƒ¢مƒ‡مƒھمƒ³م‚°è§£وگم‚’ç™؛è،¨مپ—م€پمپمپ®و§‹é€ مپŒن؛Œé‡چم‚‰مپ›م‚“مپ§مپ‚م‚‹مپ“مپ¨م‚’وڈگو،ˆمپ—مپں[9]م€‚ B-DNAمپ¯ç´°èƒه†…مپ§è¦‹م‚‰م‚Œم‚‹و،ن»¶ن¸‹مپ§وœ€م‚‚مپ‚م‚ٹمپµم‚Œمپ¦مپ„م‚‹مپŒ[50]م€پمپ“م‚Œمپ¯وکژç¢؛مپ«ه®ڑ義مپ•م‚Œمپںم‚³مƒ³مƒ›مƒ،مƒ¼م‚·مƒ§مƒ³مپ§مپ¯مپھمپڈم€پç´°èƒه†…مپ§è¦‹م‚‰م‚Œم‚‹é«کو°´ه’Œمƒ¬مƒ™مƒ«مپ§ç”ںمپکم‚‹é–¢é€£مپ™م‚‹DNAم‚³مƒ³مƒ›مƒ،مƒ¼م‚·مƒ§مƒ³مپ®ن¸€ç¾¤مپ§مپ‚م‚‹[51]م€‚مپم‚Œم‚‰مپ«ه¯¾ه؟œمپ™م‚‹Xç·ڑه›وٹکمپ¨Xç·ڑو•£ن¹±مپ®مƒ‘م‚؟مƒ¼مƒ³مپ¯م€پمپ‹مپھم‚ٹمپ®ç¨‹ه؛¦مپ®ç„،秩ه؛ڈم‚’ن¼´مپ†هˆ†هگو؛–çµگو™¶مپ«ç‰¹ه¾´çڑ„مپ§مپ‚م‚‹[52][53]م€‚ B-DNAمپ¨و¯”較مپ™م‚‹مپ¨م€پA-DNAمپ¯وµ…مپڈه؛ƒمپ„ه‰¯و؛مپ¨ç‹مپڈو·±مپ„ن¸»و؛م‚’وŒپمپ¤م€پم‚ˆم‚ٹه¹…مپ®ه؛ƒمپ„هڈ³ه·»مپچم‚‰مپ›م‚“مپ§مپ‚م‚‹م€‚Aه‹مپ¯م€پéç”ںçگ†ه¦çڑ„و،ن»¶ن¸‹مپ§مپ¯م€پ部هˆ†çڑ„مپ«è„±و°´مپ—مپںDNA試و–™ن¸مپ«ç”ںمپکم‚‹مپŒم€پç´°èƒه†…مپ§مپ¯DNAéژ–مپ¨RNAéژ–مپ®و··وˆگمƒڑم‚¢مƒھمƒ³م‚°م‚„م€پé…µç´ -DNA複هگˆن½“مپ«ç”ںمپکم‚‹مپ“مپ¨مپŒمپ‚م‚‹[54][55]م€‚ه،©هں؛مپŒمƒ،مƒپمƒ«هŒ–مپ§هŒ–ه¦ن؟®é£¾مپ•م‚ŒمپںDNAم‚»م‚°مƒ،مƒ³مƒˆمپ¯م€پم‚ˆم‚ٹه¤§مپچمپھم‚³مƒ³مƒ›مƒ،مƒ¼م‚·مƒ§مƒ³ه¤‰هŒ–م‚’èµ·مپ“مپ—م€پZ-DNAم‚’هڈ–م‚‹مپ“مپ¨مپŒمپ‚م‚‹م€‚مپ“مپ®ه ´هگˆم€پéژ–مپ¯م‚‰مپ›م‚“軸م‚’ن¸ه؟ƒمپ«ه·¦ه·»مپچمپ®م‚‰مپ›م‚“م‚’وڈڈمپچم€پم‚ˆم‚ٹن¸€èˆ¬çڑ„مپھBه‹مپ¨مپ¯و£هڈچه¯¾مپ¨مپھم‚‹[56]م€‚مپ“مپ®م‚ˆمپ†مپھ特異مپھو§‹é€ مپ¯م€پ特異çڑ„مپھZ-DNAçµگهگˆم‚؟مƒ³مƒ‘م‚¯è³ھمپ«م‚ˆمپ£مپ¦èھچèکمپ•م‚Œم€پ転ه†™هˆ¶ه¾،مپ«é–¢ن¸ژمپ—مپ¦مپ„م‚‹هڈ¯èƒ½و€§مپŒمپ‚م‚‹[57]م€‚ ن»£و›؟DNAهŒ–ه¦ه®‡ه®™ç”ں物ه¦è€…مپںمپ،مپ¯é•·ه¹´مپ«م‚ڈمپںم‚ٹم€پçڈ¾هœ¨çں¥م‚‰م‚Œمپ¦مپ„م‚‹ç”ںه‘½مپ¨مپ¯و ¹وœ¬çڑ„مپ«ç•°مپھم‚‹ç”ںهŒ–ه¦çڑ„مپٹم‚ˆمپ³هˆ†هگه¦çڑ„مƒ—مƒم‚»م‚¹م‚’用مپ„م‚‹م€پهœ°çگƒن¸ٹمپ®ه¾®ç”ں物ç”ں物هœڈ(ه½±مپ®ç”ں物هœڈ)مپ®هکهœ¨م‚’وڈگو،ˆمپ—مپ¦مپچمپںم€‚مپمپ®وڈگو،ˆمپ®ن¸€مپ¤مپ¯م€پDNAن¸مپ®مƒھمƒ³مپ®ن»£م‚ڈم‚ٹمپ«مƒ’ç´ م‚’ن½؟用مپ™م‚‹ç”ںه‘½ن½“مپ®هکهœ¨مپ§مپ‚مپ£مپںم€‚2010ه¹´م€پGFAJ-1مپ¨مپ„مپ†ç´°èڈŒمپ«مپٹمپ‘م‚‹مپمپ®هڈ¯èƒ½و€§مپŒه ±ه‘ٹمپ•م‚ŒمپںمپŒ[58][59]م€پمپ“مپ®ç ”究مپ¯è«–ن؛‰م‚’ه‘¼مپ³[59][60]م€پç´°èڈŒمپŒDNAéھ¨و ¼م‚„ن»–مپ®ç”ںن½“هˆ†هگمپ¸مپ®مƒ’ç´ مپ®هڈ–م‚ٹè¾¼مپ؟م‚’ç©چو¥µçڑ„مپ«ه¦¨مپ’مپ¦مپ„م‚‹مپ“مپ¨م‚’ç¤؛ه”†مپ™م‚‹è¨¼و‹ مپŒç¤؛مپ•م‚Œمپں[61]م€‚ ه››é‡چéژ–و§‹é€â†’詳細مپ¯م€Œم‚°م‚¢مƒ‹مƒ³ه››é‡چéژ–م€چم‚’هڈ‚ç…§
 ç·ڑçٹ¶وں“色ن½“مپ®وœ«ç«¯مپ«مپ¯م€پمƒ†مƒمƒ،م‚¢مپ¨ه‘¼مپ°م‚Œم‚‹ç‰¹و®ٹمپھDNAé کهںںمپŒمپ‚م‚‹م€‚مƒ†مƒمƒ،م‚¢مپ®ن¸»مپھه½¹ه‰²مپ¯م€پé€ڑه¸¸DNAم‚’複製مپ™م‚‹é…µç´ مپ¯وں“色ن½“مپ®3'وœ«ç«¯مپ®ç«¯éƒ¨م‚’م‚³مƒ”مƒ¼مپ§مپچمپھمپ„مپںم‚پم€پç´°èƒمپŒمƒ†مƒمƒ،مƒ©مƒ¼م‚¼مپ¨مپ„مپ†é…µç´ م‚’ن½؟用مپ—مپ¦وں“色ن½“وœ«ç«¯م‚’複製مپ§مپچم‚‹م‚ˆمپ†مپ«مپ™م‚‹مپ“مپ¨مپ§مپ‚م‚‹[63]م€‚مپ“م‚Œم‚‰مپ®ç‰¹و®ٹمپھوں“色ن½“م‚مƒ£مƒƒمƒ—مپ¯DNAوœ«ç«¯م‚’ن؟è·مپ—م€پç´°èƒمپ®DNAن؟®ه¾©ç³»مپŒمپم‚Œم‚‰م‚’ن؟®و£مپ™مپ¹مپچوگچه‚·مپ¨مپ—مپ¦و‰±مپ†مپ“مپ¨م‚’éک²مپگمپ®مپ«م‚‚ه½¹ç«‹مپ¤[64]م€‚مƒ’مƒˆç´°èƒمپ§مپ¯مƒ†مƒمƒ،م‚¢مپ¯é€ڑه¸¸م€پهچکç´”مپھ TTAGGG é…چهˆ—مپŒو•°هچƒه›ç¹°م‚ٹè؟”مپ•م‚Œمپںن¸€وœ¬éژ–DNAمپ§مپ‚م‚‹[65]م€‚ مپ“م‚Œم‚‰مپ®م‚°م‚¢مƒ‹مƒ³مپ«ه¯Œم‚“مپ é…چهˆ—مپ¯ن»–مپ®DNAهˆ†هگمپ«è¦‹م‚‰م‚Œم‚‹é€ڑه¸¸مپ®ه،©هں؛ه¯¾مپ§مپ¯مپھمپڈم€پ4ه،©هں؛هچکن½چمپŒç©چمپ؟é‡چمپھمپ£مپںو§‹é€ م‚’ه½¢وˆگمپ™م‚‹مپ“مپ¨مپ«م‚ˆمپ£مپ¦وں“色ن½“وœ«ç«¯م‚’ه®‰ه®ڑهŒ–مپ•مپ›م‚‹هڈ¯èƒ½و€§مپŒمپ‚م‚‹م€‚مپ“مپ“مپ§مپ¯4مپ¤مپ®م‚°م‚¢مƒ‹مƒ³ه،©هں؛مپŒم€پم‚°م‚¢مƒ‹مƒ³مƒ†مƒˆمƒ©مƒƒمƒ‰ï¼ˆè‹±: guanine tetrad)مپ¨ه‘¼مپ°م‚Œم‚‹ه¹³é¢م‚’ه½¢وˆگمپ—مپ¦مپ„م‚‹م€‚مپمپ—مپ¦م€پمپ“م‚Œم‚‰مپ®4ه،©هں؛هچکن½چمپ®ه¹³é¢مپŒç©چمپ؟é‡چمپھم‚ٹم€په®‰ه®ڑمپ—مپںم‚°م‚¢مƒ‹مƒ³ه››é‡چéژ–و§‹é€ م‚’ه½¢وˆگمپ™م‚‹[66]م€‚مپ“م‚Œم‚‰مپ®و§‹é€ مپ¯م€په،©هں؛مپ®ç«¯هگŒه£«مپ®و°´ç´ çµگهگˆمپ¨م€پهگ„4ه،©هں؛هچکن½چمپ®ن¸ه؟ƒمپ«مپ‚م‚‹é‡‘ه±م‚¤م‚ھمƒ³مپ®م‚مƒ¬مƒ¼مƒˆهŒ–مپ«م‚ˆمپ£مپ¦ه®‰ه®ڑهŒ–مپ—مپ¦مپ„م‚‹[67]م€‚ن»–مپ®و§‹é€ م‚’ه½¢وˆگمپ™م‚‹مپ“مپ¨م‚‚هڈ¯èƒ½مپ§م€پن¸ه¤®مپ«مپ‚م‚‹4ه،©هں؛مپ®é›†مپ¾م‚ٹمپ¯م€په،©هں؛مپ®ه‘¨ه›²مپ«وٹکم‚ٹمپںمپںمپ¾م‚Œمپںهچکéژ–مپ‹م€پمپم‚Œمپم‚ŒمپŒن¸ه¤®مپ®و§‹é€ مپ«1ه،©هں؛مپڑمپ¤ه¯„ن¸ژمپ™م‚‹مپ„مپڈمپ¤مپ‹مپ®ç•°مپھم‚‹ه¹³è،Œéژ–مپ®مپ„مپڑم‚Œمپ‹مپ‹م‚‰ه½¢وˆگمپ•م‚Œم‚‹م€‚ مپ“مپ®م‚ˆمپ†مپھç©چه±¤و§‹é€ مپ«هٹ مپˆمپ¦م€پمƒ†مƒمƒ،م‚¢مپ¯مƒ†مƒمƒ،م‚¢مƒ«مƒ¼مƒ—(Tمƒ«مƒ¼مƒ—)مپ¨ه‘¼مپ°م‚Œم‚‹ه¤§مپچمپھمƒ«مƒ¼مƒ—و§‹é€ م‚‚ه½¢وˆگمپ™م‚‹م€‚مپ“مپ“مپ§مپ¯م€پن¸€وœ¬éژ–DNAمپŒمƒ†مƒمƒ،م‚¢çµگهگˆم‚؟مƒ³مƒ‘م‚¯è³ھمپ«م‚ˆمپ£مپ¦ه®‰ه®ڑهŒ–مپ•م‚Œمپںه¤§مپچمپھه††م‚’وڈڈمپڈم‚ˆمپ†مپ«ه·»مپچمپ¤مپ„مپ¦مپ„م‚‹[68]م€‚Tمƒ«مƒ¼مƒ—مپ®وœ€ه…ˆç«¯مپ§مپ¯ن¸€وœ¬éژ–مƒ†مƒمƒ،م‚¢DNAمپŒمƒ†مƒمƒ،م‚¢éژ–مپ«م‚ˆمپ£مپ¦ن؛Œوœ¬éژ–DNAمپ®é کهںںمپ«ن؟وŒپمپ•م‚Œم€پن؛Œé‡چم‚‰مپ›م‚“DNAم‚’هˆ†é›¢مپ—م€پن؛Œوœ¬éژ–مپ®ن¸€و–¹مپ¨ه،©هں؛ه¯¾م‚’ه½¢وˆگمپ™م‚‹م€‚مپ“مپ®ن¸‰é‡چéژ–و§‹é€ مپ¯م€پç½®وڈ›مƒ«مƒ¼مƒ—مپ‚م‚‹مپ„مپ¯Dمƒ«مƒ¼مƒ—مپ¨ه‘¼مپ°م‚Œم‚‹[66]م€‚
هˆ†وDNAمپ¯م€پ複و•°مپ®وم‚’هگ«م‚€مƒچمƒƒمƒˆمƒ¯مƒ¼م‚¯م‚’ه½¢وˆگمپ™م‚‹مپ“مپ¨مپŒمپ‚م‚‹ هˆ†ه²گDNA→詳細مپ¯م€Œهˆ†ه²گDNAو³•م€چمپٹم‚ˆمپ³م€ŒDNAمƒٹمƒژمƒ†م‚¯مƒژمƒم‚¸مƒ¼م€چم‚’هڈ‚ç…§
DNAمپ§مپ¯م€پ相補çڑ„مپ§مپ‚م‚‹مپ¹مپچن؛Œوœ¬éژ–DNAمپ®وœ«ç«¯éƒ¨مپ«ç›¸è£œçڑ„مپ§مپھمپ„é کهںںمپŒهکهœ¨مپ™م‚‹مپ¨م€Œمپ»مپ¤م‚Œم€چم‚’ç”ںمپکم‚‹م€‚مپ—مپ‹مپ—第ن¸‰مپ®DNAéژ–مپŒه°ژه…¥مپ•م‚Œم€پو—¢هکمپ®ن؛Œوœ¬éژ–مپ®مپ»مپ¤م‚Œé کهںںمپ¨و··وˆگمپ§مپچم‚‹éڑ£وژ¥é کهںںم‚’هگ«م‚€ه ´هگˆم€پهˆ†ه²گDNA(英: branched DNA)مپŒç”ںمپکم‚‹هڈ¯èƒ½و€§مپŒمپ‚م‚‹م€‚هˆ†ه²گDNAمپ®وœ€م‚‚هچکç´”مپھن¾‹مپ¯3وœ¬مپ®DNAéژ–مپ®مپ؟مپ§مپ‚م‚‹مپŒم€پمپ•م‚‰مپھم‚‹éژ–مپ¨è¤‡و•°مپ®هˆ†ه²گم‚’هگ«م‚€è¤‡هگˆن½“م‚‚هڈ¯èƒ½مپ§مپ‚م‚‹[69]م€‚هˆ†ه²گDNAمپ¯م€په¹¾ن½•ه¦çڑ„ه½¢çٹ¶م‚’و§‹ç¯‰مپ™م‚‹مپںم‚پمپ«مƒٹمƒژمƒ†م‚¯مƒژمƒم‚¸مƒ¼مپ§ن½؟用مپ™م‚‹مپ“مپ¨مپŒمپ§مپچم‚‹م€‚ن»¥ن¸‹مپ®وٹ€è،“مپ«مپٹمپ‘م‚‹ç”¨é€”مپ®ç¯€م‚‚هڈ‚ç…§مپ®مپ“مپ¨م€‚ ن؛؛ه·¥ه،©هں؛→詳細مپ¯م€Œو ¸é…¸م‚¢مƒٹمƒم‚°م€چم‚’هڈ‚ç…§
مپ„مپڈمپ¤مپ‹مپ®ن؛؛ه·¥ه،©هں؛مپŒهگˆوˆگمپ•م‚Œم€پمƒڈمƒپمƒ¢م‚¸DNA(英: Hachimoji DNA)مپ¨ه‘¼مپ°م‚Œم‚‹8ه،©هں؛مپ®و ¸é…¸م‚¢مƒٹمƒم‚°مپ«çµ„مپ؟è¾¼م‚€مپ“مپ¨مپ«وˆگهٹںمپ—مپںم€‚Sم€پBم€پPم€پZمپ¨ه‘½هگچمپ•م‚Œمپںمپ“م‚Œم‚‰مپ®ن؛؛ه·¥ه،©هں؛مپ¯م€پن؛ˆو¸¬هڈ¯èƒ½مپھو–¹و³•مپ§ن؛’مپ„مپ«çµگهگˆمپ—(S-Bمپ¨P-Z)م€پDNAمپ®ن؛Œé‡چم‚‰مپ›م‚“و§‹é€ م‚’ç¶وŒپمپ—م€پRNAمپ«è»¢ه†™مپ™م‚‹مپ“مپ¨مپŒمپ§مپچم‚‹م€‚مپ“م‚Œم‚‰مپ®ن؛؛ه·¥ه،©هں؛مپ®هکهœ¨مپ¯م€پهœ°çگƒن¸ٹمپ§é€²هŒ–مپ—مپ¦مپچمپں4مپ¤مپ®ه¤©ç„¶مپ®و ¸é…¸ه،©هں؛مپ«مپ¯ç‰¹هˆ¥مپھم‚‚مپ®مپ¯ن½•م‚‚مپھمپ„مپ“مپ¨م‚’ç¤؛مپ™م‚‚مپ®مپ¨è€ƒمپˆم‚‰م‚Œم‚‹[70][71]م€‚ن¸€و–¹م€پDNAمپ¯RNAمپ¨ه¯†وژ¥مپھé–¢ن؟‚مپ«مپ‚م‚ٹم€پRNAمپ¯DNAمپ®è»¢ه†™ç”£ç‰©مپ¨مپ—مپ¦مپ مپ‘مپ§مپ¯مپھمپڈم€پç´°èƒه†…مپ§ه¤ڑمپڈمپ®ن»•ن؛‹م‚’مپ“مپھمپ™هˆ†هگو©ںو¢°مپ§م‚‚مپ‚م‚‹م€‚مپمپ®مپںم‚پمپ«مپ¯م€پRNAمپ¯éپ©هˆ‡مپھو§‹é€ مپ«وٹکم‚ٹ畳مپ¾م‚Œمپھمپ‘م‚Œمپ°مپھم‚‰مپھمپ„م€‚مپ™مپ¹مپ¦مپ®هڈ¯èƒ½مپھç«‹ن½“و§‹é€ م‚’ن½œم‚‹مپںم‚پمپ«مپ¯م€په¯¾ه؟œمپ™م‚‹RNAمپ«ه°‘مپھمپڈمپ¨م‚‚4مپ¤مپ®ه،©هں؛مپŒه؟…è¦پمپ§مپ‚م‚‹مپ“مپ¨مپŒç¤؛مپ•م‚Œمپ¦مپ„م‚‹[72]م€‚ن¸€و–¹م€پمپم‚Œن»¥ن¸ٹمپ®و•°م‚‚هڈ¯èƒ½مپ§مپ‚م‚‹مپŒم€پمپ“م‚Œمپ¯وœ€ه°ڈهٹھهٹ›مپ®è‡ھ然هژںçگ†مپ«هڈچمپ™م‚‹مپ“مپ¨مپ«مپھم‚‹م€‚ é…¸و€§ه؛¦DNAمپ®مƒھمƒ³é…¸هں؛مپ¯مƒھمƒ³é…¸مپ¨هگŒو§کمپ®é…¸و€§ç‰¹و€§م‚’ن¸ژمپˆم‚‹مپ“مپ¨مپ‹م‚‰م€په¼·é…¸مپ¨مپ؟مپھمپ™مپ“مپ¨مپŒمپ§مپچم‚‹م€‚DNAمپ¯م€پé€ڑه¸¸مپ®ç´°èƒه†…pHمپ§مپ¯ه®Œه…¨مپ«م‚¤م‚ھمƒ³هŒ–مپ—م€پ陽هگم‚’و”¾ه‡؛مپ—مپ¦مƒھمƒ³é…¸هں؛مپ¯è² é›»èچ·م‚’ه¸¯مپ³م‚‹م€‚مپ“م‚Œم‚‰مپ®è² é›»èچ·مپ¯م€پDNAم‚’هٹ و°´هˆ†è§£مپ—مپ†م‚‹و±‚و ¸ç‰©è³ھم‚’مپ¯مپمپ¤مپ‘مپ¦م€پهٹ و°´هˆ†è§£مپ«م‚ˆم‚‹هˆ†è§£مپ‹م‚‰DNAم‚’ن؟è·مپ™م‚‹[73]م€‚  ه·¨è¦–çڑ„ه¤–観細èƒمپ‹م‚‰وٹ½ه‡؛مپ•م‚Œمپں純粋مپھDNAمپ¯م€پ白مپ„糸çٹ¶مپ®ه‡é›†ه،ٹم‚’ه½¢وˆگمپ™م‚‹[74]م€‚ هŒ–ه¦ن؟®é£¾مپ¨DNAمƒ‘مƒƒم‚±مƒ¼م‚¸مƒ³م‚°مپ®ه¤‰هŒ–
م‚·مƒˆم‚·مƒ³مپŒمƒ،مƒپمƒ«هŒ–مپ•م‚Œمپں5-مƒ،مƒپمƒ«م‚·مƒˆم‚·مƒ³مپ¯م€پ脱م‚¢مƒںمƒژهŒ–مپ«م‚ˆم‚ٹمƒپمƒںمƒ³مپ«ه¤‰وڈ›مپ•م‚Œم‚‹
ه،©هں؛ن؟®é£¾مپ¨DNAمƒ‘مƒƒم‚±مƒ¼م‚¸مƒ³م‚°â†’詳細مپ¯م€ŒDNAمƒ،مƒپمƒ«هŒ–م€چمپٹم‚ˆمپ³م€Œم‚¯مƒمƒمƒپمƒ³مƒھمƒ¢مƒ‡مƒھمƒ³م‚°م€چم‚’هڈ‚ç…§
éپ؛ن¼هگمپ®ç™؛çڈ¾مپ¯م€پDNAمپŒوں“色ن½“مپ®ن¸مپ§م‚¯مƒمƒمƒپمƒ³مپ¨ه‘¼مپ°م‚Œم‚‹éڑژه±¤çڑ„مپھو§‹é€ مپ«مپ©مپ®م‚ˆمپ†مپ«مƒ‘مƒƒم‚±مƒ¼م‚¸مƒ³م‚°مپ•م‚Œمپ¦مپ„م‚‹مپ‹مپ«ه½±éں؟مپ•م‚Œم‚‹م€‚ه،©هں؛ن؟®é£¾مپ¯مƒ‘مƒƒم‚±مƒ¼م‚¸مƒ³م‚°مپ«é–¢ن¸ژمپ™م‚‹هڈ¯èƒ½و€§مپŒمپ‚م‚ٹم€پéپ؛ن¼هگç™؛çڈ¾مپŒن½ژمپ„مپ‹مپ¾مپ£مپںمپڈمپھمپ„é کهںںمپ¯é€ڑه¸¸م€پم‚·مƒˆم‚·مƒ³ه،©هں؛مپ®مƒ،مƒپمƒ«هŒ–مپŒé«کمƒ¬مƒ™مƒ«مپ§è¦‹م‚‰م‚Œم‚‹م€‚DNAمƒ‘مƒƒم‚±مƒ¼م‚¸مƒ³م‚°مپ¨مپمپ®éپ؛ن¼هگç™؛çڈ¾مپ¸مپ®ه½±éں؟مپ¯م€پم‚¯مƒمƒمƒپمƒ³و§‹é€ مپ«مپٹمپ„مپ¦DNAمپŒه·»مپچمپ¤مپ„مپ¦مپ„م‚‹مƒ’م‚¹مƒˆمƒ³م‚؟مƒ³مƒ‘م‚¯è³ھم‚³م‚¢مپ®ه…±وœ‰çµگهگˆن؟®é£¾م‚„م€پم‚¯مƒمƒمƒپمƒ³مƒ»مƒھمƒ¢مƒ‡مƒھمƒ³م‚°è¤‡هگˆن½“مپ«م‚ˆم‚‹مƒھمƒ¢مƒ‡مƒھمƒ³م‚°مپ§م‚‚èµ·مپ“م‚ٹمپ†م‚‹م€‚مپ•م‚‰مپ«م€پDNAمƒ،مƒپمƒ«هŒ–مپ¨مƒ’م‚¹مƒˆمƒ³ن؟®é£¾مپ®é–“مپ«مپ¯م‚¯مƒم‚¹مƒˆمƒ¼م‚¯مپŒمپ‚م‚‹مپںم‚پم€پم‚¯مƒمƒمƒپمƒ³مپ¨éپ؛ن¼هگç™؛çڈ¾مپ«هچ”èھ؟çڑ„مپ«ه½±éں؟م‚’ن¸ژمپˆم‚‹هڈ¯èƒ½و€§مپŒمپ‚م‚‹[75]م€‚ مپںمپ¨مپˆمپ°م€پم‚·مƒˆم‚·مƒ³مپ®مƒ،مƒپمƒ«هŒ–مپ¯5-مƒ،مƒپمƒ«م‚·مƒˆم‚·مƒ³م‚’ç”ںوˆگمپ—م€پمپ“م‚Œمپ¯Xوں“色ن½“مپ®ن¸چو´»و€§هŒ–مپ«é‡چè¦پمپ§مپ‚م‚‹[76]م€‚مƒ،مƒپمƒ«هŒ–مپ®ه¹³ه‡مƒ¬مƒ™مƒ«مپ¯ç”ں物مپ«م‚ˆمپ£مپ¦ç•°مپھم‚ٹم€پم‚«م‚¨مƒژمƒ©مƒ–مƒ‡م‚£مƒ†م‚£م‚¹مƒ»م‚¨مƒ¬م‚¬مƒ³م‚¹ï¼ˆCaenorhabditis elegans)مپ¨مپ„مپ†ç·ڑ虫مپ¯م‚·مƒˆم‚·مƒ³مپ®مƒ،مƒپمƒ«هŒ–م‚’و¬ مپڈمپŒم€پè„ٹو¤ژه‹•ç‰©مپ¯مƒ،مƒپمƒ«هŒ–مپ®مƒ¬مƒ™مƒ«مپŒé«کمپڈم€پDNAمپ®وœ€ه¤§1%مپŒ5-مƒ،مƒپمƒ«م‚·مƒˆم‚·مƒ³م‚’هگ«م‚€[77]م€‚5-مƒ،مƒپمƒ«م‚·مƒˆم‚·مƒ³مپ¯é‡چè¦پمپ§مپ‚م‚‹مپ«م‚‚مپ‹مپ‹م‚ڈم‚‰مپڑم€پ脱م‚¢مƒںمƒژهŒ–مپ—مپ¦مƒپمƒںمƒ³ه،©هں؛مپ«ه¤‰وڈ›مپ•م‚Œم‚‹مپ“مپ¨مپŒمپ‚م‚‹مپںم‚پم€پمƒ،مƒپمƒ«هŒ–م‚·مƒˆم‚·مƒ³مپ¯ç‰¹مپ«ه¤‰ç•°م‚’èµ·مپ“مپ—م‚„مپ™مپ„[78]م€‚مپمپ®ن»–مپ®ه،©هں؛ن؟®é£¾مپ¨مپ—مپ¦مپ¯م€پç´°èڈŒمپ«مپٹمپ‘م‚‹م‚¢مƒ‡مƒ‹مƒ³مپ®مƒ،مƒپمƒ«هŒ–م€پ脳مپ«مپٹمپ‘م‚‹5-مƒ’مƒ‰مƒم‚م‚·مƒ،مƒپمƒ«م‚·مƒˆم‚·مƒ³مپ®هکهœ¨[79]م€پمپٹم‚ˆمپ³م‚مƒچمƒˆمƒ—مƒ©م‚¹مƒˆé،مپ«مپٹمپ‘م‚‹ه،©هں؛Jم‚’ç”ںوˆگمپ™م‚‹مپںم‚پمپ®م‚¦مƒ©م‚·مƒ«مپ®م‚°مƒھم‚³م‚·مƒ«هŒ–مپھمپ©مپŒمپ‚م‚‹[80][81]م€‚ وگچه‚·â†’詳細مپ¯م€ŒDNAوگچه‚· (è‡ھ然ç™؛ç”ں)م€چم€پم€Œه¤‰ç•°م€چم€پمپٹم‚ˆمپ³م€Œè€پهŒ–مپ«مپٹمپ‘م‚‹DNAوگچه‚·çگ†è«–م€چم‚’هڈ‚ç…§
 DNAمپ¯م€پDNAé…چهˆ—م‚’ه¤‰هŒ–مپ•مپ›م‚‹مپ•مپ¾مپ–مپ¾مپھ種é،مپ®ه¤‰ç•°هژںمپ«م‚ˆمپ£مپ¦وگچه‚·م‚’هڈ—مپ‘م‚‹هڈ¯èƒ½و€§مپŒمپ‚م‚‹م€‚ه¤‰ç•°هژںمپ«مپ¯م€پé…¸هŒ–ه‰¤م‚„م‚¢مƒ«م‚مƒ«هŒ–ه‰¤مپھمپ©مپ®هŒ–ه¦ç‰©è³ھمپ®مپ»مپ‹م€پç´«ه¤–ç·ڑم‚„Xç·ڑمپھمپ©مپ®é«کم‚¨مƒچمƒ«م‚®مƒ¼é›»ç£پو”¾ه°„ç·ڑم‚‚هگ«مپ¾م‚Œم‚‹م€‚مپ©مپ®م‚ˆمپ†مپھDNAوگچه‚·مپŒç”ںمپکم‚‹مپ‹مپ¯ه¤‰ç•°هژںمپ®ç¨®é،مپ«م‚ˆمپ£مپ¦ç•°مپھم‚‹م€‚مپںمپ¨مپˆمپ°م€پç´«ه¤–ç·ڑمپ¯مƒ”مƒھمƒںم‚¸مƒ³ه،©هں؛é–“مپ®و¶و©‹مپ§مپ‚م‚‹مƒپمƒںمƒ³ن؛Œé‡ڈن½“م‚’ç”ںوˆگمپ™م‚‹مپ“مپ¨مپ«م‚ˆمپ£مپ¦م€پDNAمپ«وگچه‚·م‚’ن¸ژمپˆم‚‹هڈ¯èƒ½و€§مپŒمپ‚م‚‹[83]م€‚ن¸€و–¹م€پمƒ•مƒھمƒ¼مƒ©م‚¸م‚«مƒ«م‚„éپژé…¸هŒ–و°´ç´ مپ®م‚ˆمپ†مپھé…¸هŒ–ه‰¤مپ¯م€په،©هں؛ن؟®é£¾م€پ特مپ«م‚°م‚¢مƒژم‚·مƒ³مپ®ن؟®é£¾م‚„م€پن؛Œوœ¬éژ–هˆ‡و–مپھمپ©م€پمپ•مپ¾مپ–مپ¾مپھه½¢مپ®وگچه‚·م‚’ه¼•مپچèµ·مپ“مپ™[84]م€‚ه…¸ه‹çڑ„مپھمƒ’مƒˆç´°èƒمپ«مپ¯م€پé…¸هŒ–çڑ„وگچه‚·م‚’هڈ—مپ‘مپںه،©هں؛مپŒç´„15ن¸‡ه€‹و‰€مپ‚م‚‹[85]م€‚مپ“م‚Œم‚‰مپ®é…¸هŒ–çڑ„وگچه‚·مپ®مپ†مپ،وœ€م‚‚هچ±é™؛مپھمپ®مپ¯ن؟®ه¾©مپŒه›°é›£مپھن؛Œوœ¬éژ–هˆ‡و–مپ§مپ‚م‚ٹم€پ点ه¤‰ç•°م€پDNAé…چهˆ—مپ‹م‚‰مپ®وŒ؟ه…¥م‚„و¬ ه¤±م€پمپ‚م‚‹مپ„مپ¯وں“色ن½“転ه؛§م‚’ه¼•مپچèµ·مپ“مپ™هڈ¯èƒ½و€§مپŒمپ‚م‚‹[86]م€‚مپ“م‚Œم‚‰مپ®ه¤‰ç•°مپ¯ç™Œï¼ˆمپŒم‚“)م‚’ه¼•مپچèµ·مپ“مپ™هڈ¯èƒ½و€§مپŒمپ‚م‚‹م€‚DNAن؟®ه¾©و©ںو§‹مپ«مپ¯وœ¬è³ھçڑ„مپھé™گç•ŒمپŒمپ‚م‚‹مپںم‚پم€پن؛؛é–“مپŒé•·ç”ںمپچمپ™م‚Œمپ°م€پمپ„مپڑم‚Œمپ¯èھ°م‚‚癌م‚’ç™؛ç—‡مپ™م‚‹مپ“مپ¨مپ«مپھم‚‹[87][88]م€‚و´»و€§é…¸ç´ 種م‚„ç´°èƒو°´مپ®هٹ و°´هˆ†è§£و´»و€§مپھمپ©م‚’産ç”ںمپ™م‚‹و£ه¸¸مپھç´°èƒمƒ—مƒم‚»م‚¹مپ«èµ·ه› مپ™م‚‹م€پè‡ھ然ç™؛ç”ںçڑ„مپھDNAوگچه‚·م‚‚é »ç¹پمپ«èµ·مپ“م‚‹م€‚مپ“م‚Œم‚‰مپ®وگچه‚·مپ®ه¤§éƒ¨هˆ†مپ¯ن؟®ه¾©مپ•م‚Œم‚‹مپŒم€پمپ©مپ®ç´°èƒمپ«مپٹمپ„مپ¦م‚‚م€پن؟®ه¾©éپژ程مپ®ن½œç”¨مپ«م‚‚مپ‹مپ‹م‚ڈم‚‰مپڑم€پDNAوگچه‚·مپ®ن¸€éƒ¨مپŒو®‹م‚‹مپ“مپ¨مپŒمپ‚م‚‹م€‚مپ“م‚Œم‚‰مپ®و®‹هکDNAوگچه‚·مپ¯م€په“؛ن¹³é،مپ®وœ‰ç³¸هˆ†è£‚ه¾Œçµ„ç¹”مپ«مپٹمپ„مپ¦هٹ é½¢مپ¨مپ¨م‚‚مپ«è“„ç©چمپ™م‚‹م€‚مپ“مپ®è“„ç©چمپ¯è€پهŒ–مپ®é‡چè¦پمپھو ¹وœ¬هژںه› مپ§مپ‚م‚‹مپ¨è€ƒمپˆم‚‰م‚Œمپ¦مپ„م‚‹[89][90][91]م€‚ ه¤‰ç•°هژںمپ®ه¤ڑمپڈمپ¯éڑ£وژ¥مپ™م‚‹2مپ¤مپ®ه،©هں؛ه¯¾مپ®é–“مپ«ن¾µه…¥مپ—م€پمپ“م‚Œمپ¯م‚¤مƒ³م‚؟مƒ¼م‚«مƒ¬مƒ¼م‚·مƒ§مƒ³ï¼ˆè‹±: intercalation)مپ¨ه‘¼مپ°م‚Œم‚‹éپژ程مپ§مپ‚م‚‹م€‚مپ»مپ¨م‚“مپ©مپ®م‚¤مƒ³م‚؟مƒ¼م‚«مƒ¬مƒ¼م‚؟مƒ¼ï¼ˆن¾µه…¥ç‰©è³ھ)مپ¯èٹ³é¦™و—ڈمپ®ه¹³é¢هˆ†هگمپ§مپ‚م‚ٹم€پمپںمپ¨مپˆمپ°è‡هŒ–م‚¨مƒپم‚¸م‚¦مƒ م€پم‚¢م‚¯مƒھم‚¸مƒ³م€پمƒ€م‚¦مƒژمƒ«مƒ“م‚·مƒ³م€پمƒ‰م‚م‚½مƒ«مƒ“م‚·مƒ³مپھمپ©مپ§مپ‚م‚‹م€‚م‚¤مƒ³م‚؟مƒ¼م‚«مƒ¬مƒ¼م‚؟مƒ¼مپŒه،©هں؛ه¯¾مپ®é–“مپ«ن¾µه…¥مپ™م‚‹مپںم‚پمپ«مپ¯م€په،©هں؛مپŒé›¢م‚Œمپھمپ‘م‚Œمپ°مپھم‚‰مپڑم€پن؛Œé‡چم‚‰مپ›م‚“مپŒمپ»مپ©مپ‘م‚‹مپ“مپ¨مپ§DNAéژ–مپ«وھمپ؟مپŒç”ںمپکم‚‹م€‚مپ“م‚Œمپ¯è»¢ه†™مپ¨DNA複製مپ®ن¸،و–¹م‚’éک»ه®³مپ—م€پو¯’و€§مپ¨ه¤‰ç•°م‚’ه¼•مپچèµ·مپ“مپ™[92]م€‚مپمپ®çµگوœم€پDNAم‚¤مƒ³م‚؟مƒ¼م‚«مƒ¬مƒ¼م‚؟مƒ¼مپ¯ç™؛癌و€§م‚’ç”ںمپکم€پمپ¾مپںم‚µمƒھمƒ‰مƒم‚¤مƒ‰مپ®ه ´هگˆمپ¯ه‚¬ه¥‡ه½¢و€§م‚’ç”ںمپکم‚‹هڈ¯èƒ½و€§مپŒمپ‚م‚‹[93]م€‚مپ¾مپںم€پمƒ™مƒ³م‚¾[a]مƒ”مƒ¬مƒ³م‚¸م‚ھمƒ¼مƒ«م‚¨مƒم‚م‚·مƒ‰م‚„م‚¢مƒ•مƒ©مƒˆم‚م‚·مƒ³مپ®م‚ˆمپ†مپ«م€پDNAن»کهٹ ن½“م‚’ه½¢وˆگمپ—م€پ複製èھ¤م‚ٹم‚’ه¼•مپچèµ·مپ“مپ™م‚‚مپ®م‚‚مپ‚م‚‹[94]م€‚مپم‚Œمپ«م‚‚مپ‹مپ‹م‚ڈم‚‰مپڑم€پDNAمپ®è»¢ه†™م‚„複製م‚’éک»ه®³مپ™م‚‹èƒ½هٹ›مپŒمپ‚م‚‹مپںم‚پم€پن»–مپ®é،ن¼¼و¯’ç´ م‚‚م€پو€¥é€ںمپ«ه¢—و®–مپ™م‚‹ç™Œç´°èƒم‚’éک»ه®³مپ™م‚‹هŒ–ه¦ç™‚و³•مپ«ن½؟用مپ•م‚Œمپ¦مپ„م‚‹[95]م€‚ ç”ں物ه¦çڑ„و©ں能 DNAمپ¯é€ڑه¸¸م€پçœںو ¸ç”ں物مپ§مپ¯ç·ڑçٹ¶وں“色ن½“مپ¨مپ—مپ¦هکهœ¨مپ—م€پهژںو ¸ç”ں物مپ§مپ¯ç’°çٹ¶وں“色ن½“مپ¨مپ—مپ¦هکهœ¨مپ™م‚‹م€‚ç´°èƒه†…مپ®وں“色ن½“مپ®é›†هگˆمپŒم‚²مƒژمƒ م‚’و§‹وˆگمپ—م€پمƒ’مƒˆم‚²مƒژمƒ مپ§مپ¯46وœ¬مپ®وں“色ن½“مپ«ç´„30ه„„ه،©هں؛ه¯¾مپ®DNAمپŒé…چç½®مپ•م‚Œمپ¦مپ„م‚‹[96]م€‚DNAمپŒن¼éپ”مپ™م‚‹وƒ…ه ±مپ¯م€پéپ؛ن¼هگمپ¨ه‘¼مپ°م‚Œم‚‹DNAو–片مپ®é…چهˆ—مپ«هگ«مپ¾م‚Œمپ¦مپ„م‚‹م€‚éپ؛ن¼هگمپ«م‚ˆم‚‹éپ؛ن¼وƒ…ه ±مپ®ن¼éپ”مپ™مپھم‚ڈمپ،éپ؛ن¼مپ¯م€پ相補çڑ„مپھه،©هں؛ه¯¾ه½¢وˆگمپ«م‚ˆمپ£مپ¦éپ”وˆگمپ•م‚Œم‚‹م€‚مپںمپ¨مپˆمپ°م€پ転ه†™مپ«مپٹمپ„مپ¦ç´°èƒمپŒéپ؛ن¼هگمپ®وƒ…ه ±م‚’ن½؟用مپ™م‚‹éڑ›م€پDNAمپ¨و£مپ—مپ„RNAمƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰مپ¨مپ®é–“مپ«ه¼•هٹ›مپŒن½œç”¨مپ™م‚‹مپ“مپ¨مپ§م€پDNAé…چهˆ—مپŒç›¸è£œçڑ„مپھRNAé…چهˆ—مپ«è¤‡è£½مپ•م‚Œم‚‹م€‚é€ڑه¸¸م€پç؟»è¨³مپ¨ه‘¼مپ°م‚Œم‚‹éپژ程مپ§م€پمپ“مپ®RNAم‚³مƒ”مƒ¼مپ¯ن¸€è‡´مپ™م‚‹م‚؟مƒ³مƒ‘م‚¯è³ھé…چهˆ—م‚’ن½œم‚‹مپںم‚پمپ«ن½؟用مپ•م‚Œم‚‹مپŒم€پمپ“م‚Œم‚‚RNAمƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰é–“مپ®هگŒو§کمپھ相ن؛’ن½œç”¨مپ«ن¾هکمپ—مپ¦مپ„م‚‹م€‚مپ‚م‚‹مپ„مپ¯م€پç´°èƒمپ¯DNA複製مپ¨ه‘¼مپ°م‚Œم‚‹éپژ程مپ§م€پمپمپ®éپ؛ن¼وƒ…ه ±م‚’複製مپ™م‚‹مپ“مپ¨مپŒمپ§مپچم‚‹م€‚مپ“م‚Œم‚‰مپ®و©ں能مپ®è©³ç´°مپ«مپ¤مپ„مپ¦مپ¯ن»–مپ®è¨کن؛‹مپ§هڈ–م‚ٹن¸ٹمپ’مپ¦مپٹم‚ٹم€پمپ“مپ“مپ§مپ¯م‚²مƒژمƒ مپ®و©ں能م‚’ن»²ن»‹مپ™م‚‹DNAمپ¨ن»–مپ®هˆ†هگمپ¨مپ®ç›¸ن؛’ن½œç”¨مپ«ç„¦ç‚¹م‚’ه½“مپ¦م‚‹م€‚ éپ؛ن¼هگمپ¨م‚²مƒژمƒâ†’詳細مپ¯م€Œç´°èƒو ¸م€چم€پم€Œم‚¯مƒمƒمƒپمƒ³م€چم€پم€Œوں“色ن½“م€چم€پم€Œéپ؛ن¼هگم€چم€پمپٹم‚ˆمپ³م€Œمƒژمƒ³م‚³مƒ¼مƒ‡م‚£مƒ³م‚°DNAم€چم‚’هڈ‚ç…§
م‚²مƒژمƒ DNAمپ¯م€پDNAه‡ç¸®مپ¨ه‘¼مپ°م‚Œم‚‹éپژ程م‚’é€ڑمپکمپ¦م€پç´°èƒمپ®ه°ڈمپ•مپھن½“ç©چمپ«هڈژمپ¾م‚‹م‚ˆمپ†مپ«مپچمپ¤مپڈو•´ç„¶مپ¨è©°م‚پè¾¼مپ¾م‚Œمپ¦مپ„م‚‹م€‚çœںو ¸ç”ں物مپ®ه ´هگˆم€پDNAمپ¯ç´°èƒو ¸مپ«هکهœ¨مپ—م€پمƒںمƒˆم‚³مƒ³مƒ‰مƒھم‚¢م‚„葉緑ن½“مپ«م‚‚ه°‘é‡ڈمپŒهکهœ¨مپ™م‚‹م€‚هژںو ¸ç”ں物مپ§مپ¯م€پDNAمپ¯و ¸و§کن½“(مƒŒم‚¯مƒ¬م‚ھم‚¤مƒ‰ï¼‰مپ¨ه‘¼مپ°م‚Œم‚‹ç´°èƒè³ھه†…مپ®ن¸چè¦ڈه‰‡مپھه½¢م‚’مپ—مپںو§‹é€ ن½“مپ«ن؟وŒپمپ•م‚Œمپ¦مپ„م‚‹[97]م€‚م‚²مƒژمƒ مپ®éپ؛ن¼وƒ…ه ±مپ¯éپ؛ن¼هگه†…مپ«ن؟وŒپمپ•م‚Œمپ¦مپٹم‚ٹم€پç”ں物مپ«مپٹمپ‘م‚‹مپ“مپ®وƒ…ه ±مپ®ه®Œه…¨مپھ集هگˆم‚’مپمپ®éپ؛ن¼ه‹ï¼ˆè‹±: genotype)مپ¨ه‘¼مپ¶م€‚éپ؛ن¼هگمپ¯éپ؛ن¼مپ®هچکن½چمپ§مپ‚م‚ٹم€پç”ں物مپ®ç‰¹ه®ڑمپ®ه½¢è³ھمپ«ه½±éں؟م‚’ن¸ژمپˆم‚‹DNAمپ®é کهںںمپ§مپ‚م‚‹م€‚éپ؛ن¼هگمپ«مپ¯م€پ転ه†™هڈ¯èƒ½مپھم‚ھمƒ¼مƒ—مƒ³مƒھمƒ¼مƒ‡م‚£مƒ³م‚°مƒ•مƒ¬مƒ¼مƒ مپ¨م€پم‚ھمƒ¼مƒ—مƒ³مƒھمƒ¼مƒ‡م‚£مƒ³م‚°مƒ•مƒ¬مƒ¼مƒ مپ®è»¢ه†™م‚’هˆ¶ه¾،مپ™م‚‹مƒ—مƒمƒ¢مƒ¼م‚؟مƒ¼م‚„م‚¨مƒ³مƒڈمƒ³م‚µمƒ¼مپھمپ©مپ®هˆ¶ه¾،é…چهˆ—مپŒهگ«مپ¾م‚Œمپ¦مپ„م‚‹م€‚ ه¤ڑمپڈمپ®ç”ں物種مپ§مپ¯م€پم‚²مƒژمƒ é…چهˆ—ه…¨ن½“مپ®مپ”مپڈن¸€éƒ¨مپ®مپ؟م‚؟مƒ³مƒ‘م‚¯è³ھم‚’م‚³مƒ¼مƒ‰مپ—مپ¦مپ„م‚‹م€‚مپںمپ¨مپˆمپ°م€پمƒ’مƒˆم‚²مƒژمƒ مپ®مپ†مپ،م‚؟مƒ³مƒ‘م‚¯è³ھم‚’م‚³مƒ¼مƒ‰مپ™م‚‹م‚¨م‚¯م‚½مƒ³مپ¯م‚ڈمپڑمپ‹ç´„1.5ï¼…مپ—مپ‹مپھمپڈم€پمƒ’مƒˆDNAمپ®50%ن»¥ن¸ٹمپ¯éم‚³مƒ¼مƒ‰هڈچه¾©é…چهˆ—مپ§و§‹وˆگمپ•م‚Œمپ¦مپ„م‚‹[98]م€‚çœںو ¸ç”ں物مپ®م‚²مƒژمƒ مپ«éه¸¸مپ«ه¤ڑمپڈمپ®éم‚³مƒ¼مƒ‰DNAمپŒهکهœ¨مپ™م‚‹çگ†ç”±مپ¨م€پم‚²مƒژمƒ مپ®ه¤§مپچمپ•ï¼ˆCه€¤ï¼‰مپŒç”ں物種مپ«م‚ˆمپ£مپ¦è‘—مپ—مپڈç•°مپھم‚‹çگ†ç”±مپ¯م€پم€ŒCه€¤مپ®è¬ژم€چمپ¨مپ—مپ¦çں¥م‚‰م‚Œم‚‹é•·ه¹´مپ®é›£ه•ڈمپ§مپ‚م‚‹[99]م€‚مپ—مپ‹مپ—م€پم‚؟مƒ³مƒ‘م‚¯è³ھم‚’م‚³مƒ¼مƒ‰مپ—مپھمپ„DNAé…چهˆ—مپ®ن¸مپ«مپ¯م€پéپ؛ن¼هگç™؛çڈ¾مپ®èھ؟節مپ«é–¢ن¸ژمپ™م‚‹و©ں能çڑ„مپھéم‚³مƒ¼مƒ‰RNAهˆ†هگم‚’م‚³مƒ¼مƒ‰مپ—مپ¦مپ„م‚‹م‚‚مپ®م‚‚مپ‚م‚‹[100]م€‚  éم‚³مƒ¼مƒ‰DNAé…چهˆ—مپ®ن¸مپ«مپ¯وں“色ن½“مپ®و§‹é€ çڑ„ه½¹ه‰²م‚’وœمپںمپ™م‚‚مپ®مپŒمپ‚م‚‹م€‚مƒ†مƒمƒ،م‚¢مپ¨م‚»مƒ³مƒˆمƒمƒ،م‚¢مپ«مپ¯é€ڑه¸¸م€پمپ»مپ¨م‚“مپ©éپ؛ن¼هگمپŒهکهœ¨مپ—مپھمپ„مپŒم€پوں“色ن½“مپ®و©ں能مپ¨ه®‰ه®ڑو€§مپ«مپ¨مپ£مپ¦é‡چè¦پمپ§مپ‚م‚‹[64][102]م€‚مƒ’مƒˆمپ«ه¤ڑمپڈهکهœ¨مپ™م‚‹éم‚³مƒ¼مƒ‰DNAمپ¯هپ½éپ؛ن¼هگمپ§مپ‚م‚ٹم€په¤‰ç•°مپ«م‚ˆمپ£مپ¦و©ں能مپ—مپھمپڈمپھمپ£مپںéپ؛ن¼هگمپ®è¤‡è£½مپ§مپ‚م‚‹[103]م€‚مپ“م‚Œم‚‰مپ®é…چهˆ—مپ¯م€پéپ؛ن¼هگمپ®é‡چ複م‚„هˆ†ه²گمپ®éپژ程م‚’é€ڑمپکمپ¦م€پو–°مپ—مپ„éپ؛ن¼هگم‚’ç”ںمپ؟ه‡؛مپ™مپںم‚پمپ®éپ؛ن¼ç‰©è³ھمپ®هژںو–™مپ¨مپ—مپ¦ه½¹مپ«ç«‹مپ¤مپ“مپ¨م‚‚مپ‚م‚‹مپŒم€پé€ڑه¸¸مپ¯هچکمپھم‚‹هˆ†هگمپ®éپ؛物مپ§مپ‚م‚‹[104]م€‚ 転ه†™مپ¨ç؟»è¨³â†’詳細مپ¯م€Œéپ؛ن¼وƒ…ه ±م€چم€پم€Œè»¢ه†™ (ç”ں物ه¦)م€چم€پمپٹم‚ˆمپ³م€Œم‚؟مƒ³مƒ‘م‚¯è³ھç”ںهگˆوˆگم€چم‚’هڈ‚ç…§
éپ؛ن¼هگمپ¯éپ؛ن¼وƒ…ه ±م‚’هگ«م‚€DNAé…چهˆ—مپ§م€پç”ں物مپ®è،¨çڈ¾ه‹مپ«ه½±éں؟م‚’ن¸ژمپˆم‚‹مپ“مپ¨مپŒمپ‚م‚‹م€‚éپ؛ن¼هگه†…مپ§مپ¯م€پDNAéژ–مپ«و²؟مپ£مپںه،©هں؛é…چهˆ—مپŒمƒ،مƒƒم‚»مƒ³م‚¸مƒ£مƒ¼RNAé…چهˆ—م‚’è¦ڈه®ڑمپ—م€پمپم‚ŒمپŒ1مپ¤مپ‹è¤‡و•°مپ®م‚؟مƒ³مƒ‘م‚¯è³ھé…چهˆ—م‚’è¦ڈه®ڑمپ™م‚‹م€‚éپ؛ن¼هگمپ®مƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰é…چهˆ—مپ¨م‚؟مƒ³مƒ‘م‚¯è³ھمپ®م‚¢مƒںمƒژé…¸é…چهˆ—مپ¨مپ®é–¢ن؟‚مپ¯م€پéپ؛ن¼وڑ—هڈ·مپ¨ç·ڈ称مپ•م‚Œم‚‹ç؟»è¨³è¦ڈه‰‡مپ«م‚ˆمپ£مپ¦و±؛ه®ڑمپ•م‚Œم‚‹م€‚éپ؛ن¼وڑ—هڈ·مپ¯م€پم‚³مƒ‰مƒ³ï¼ˆcodon)مپ¨ه‘¼مپ°م‚Œم‚‹3و–‡ه—مپ®م€Œهچکèھم€چمپ‹م‚‰مپھم‚ٹ(ن¾‹ï¼ڑACTم€پCAGم€پTTT)م€پمƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰مپŒ3ه€‹é€£ç¶ڑمپ—مپںé…چهˆ—مپ«هں؛مپ¥مپ„مپ¦مپ„م‚‹م€‚ 転ه†™مپ®éڑ›م€پéپ؛ن¼هگمپ®م‚³مƒ‰مƒ³مپŒRNAمƒمƒھمƒ،مƒ©مƒ¼م‚¼مپ«م‚ˆمپ£مپ¦مƒ،مƒƒم‚»مƒ³م‚¸مƒ£مƒ¼RNAمپ«م‚³مƒ”مƒ¼مپ•م‚Œم‚‹م€‚و¬،مپ«م€پمپ“مپ®RNAم‚³مƒ”مƒ¼مپ¯مƒھمƒœم‚½مƒ¼مƒ مپ«م‚ˆمپ£مپ¦è§£èھمپ•م‚Œم€پمƒھمƒœم‚½مƒ¼مƒ مپ¯مƒ،مƒƒم‚»مƒ³م‚¸مƒ£مƒ¼RNAم‚’م‚¢مƒںمƒژé…¸م‚’éپ‹مپ¶مƒˆمƒ©مƒ³م‚¹مƒ•م‚،مƒ¼RNAمپ«ه،©هں؛ه¯¾هگˆمپ•مپ›م‚‹مپ“مپ¨مپ«م‚ˆمپ£مپ¦RNAé…چهˆ—م‚’èھمپ؟هڈ–م‚‹م€‚4種é،مپ®ه،©هں؛م‚’è،¨مپ™3و–‡ه—مپŒçµ„مپ؟هگˆم‚ڈمپ•مپ£مپ¦م€پ64é€ڑم‚ٹمپ®م‚³مƒ‰مƒ³مپ®هڈ¯èƒ½و€§مپŒهکهœ¨مپ™م‚‹ï¼ˆ43 é€ڑم‚ٹمپ®çµ„مپ؟هگˆم‚ڈمپ›ï¼‰م€‚مپ“م‚Œم‚‰مپ®م‚³مƒ‰مƒ³مپ¯20種é،مپ®و¨™و؛–م‚¢مƒںمƒژé…¸م‚’م‚³مƒ¼مƒ‰مپ—مپ¦مپٹم‚ٹم€پمپ»مپ¨م‚“مپ©مپ®م‚¢مƒںمƒژé…¸مپ¯è¤‡و•°مپ®م‚³مƒ‰مƒ³مپ«ه¯¾ه؟œن»کمپ‘م‚‰م‚Œم‚‹م€‚مپ¾مپںم€پم‚³مƒ¼مƒ‰é کهںںمپ®çµ‚م‚ڈم‚ٹم‚’ç¤؛مپ™3مپ¤مپ®م€Œçµ‚و¢م‚³مƒ‰مƒ³م€چ(مƒٹمƒ³م‚»مƒ³م‚¹م‚³مƒ‰مƒ³مپ¨م‚‚ه‘¼مپ°م‚Œم‚‹ï¼‰م‚‚مپ‚م‚‹م€‚مپ“م‚Œم‚‰مپ¯م€پTAGم€پTAAم€پTGAم‚³مƒ‰مƒ³مپ§مپ‚م‚‹ï¼ˆmRNAمپ§مپ¯UAGم€پUAAم€پUGA)م€‚  複製→詳細مپ¯م€ŒDNA複製م€چم‚’هڈ‚ç…§
ç´°èƒهˆ†è£‚مپ¯ç”ں物مپŒوˆگé•·مپ™م‚‹مپںم‚پمپ«ن¸چهڈ¯و¬ مپ§مپ‚م‚‹مپŒم€پç´°èƒمپŒهˆ†è£‚مپ™م‚‹éڑ›مپ«مپ¯م€پ2مپ¤مپ®ه¨کç´°èƒمپŒè¦ھمپ¨هگŒمپکéپ؛ن¼وƒ…ه ±م‚’وŒپمپ¤م‚ˆمپ†مپ«م€پم‚²مƒژمƒ ن¸مپ®DNAم‚’複製مپ—مپھمپ‘م‚Œمپ°مپھم‚‰مپھمپ„م€‚DNAمپ®ن؛Œوœ¬éژ–و§‹é€ مپ¯DNA複製مپ®هچکç´”مپھو©ںو§‹م‚’وڈگن¾›مپ™م‚‹م€‚مپ“مپ“مپ§مپ¯ن؛Œوœ¬éژ–مپŒهˆ†é›¢مپ•م‚Œم€پو¬،مپ«DNAمƒمƒھمƒ،مƒ©مƒ¼م‚¼مپ¨ه‘¼مپ°م‚Œم‚‹é…µç´ مپ«م‚ˆمپ£مپ¦مپم‚Œمپم‚Œمپ®éژ–مپ®ç›¸è£œçڑ„DNAé…چهˆ—مپŒه†چن½œوˆگمپ•م‚Œم‚‹م€‚مپ“مپ®é…µç´ مپ¯م€پ相補çڑ„ه،©هں؛ه¯¾مپ®ه½¢وˆگم‚’é€ڑمپکمپ¦و£مپ—مپ„ه،©هں؛م‚’見مپ¤مپ‘م€پمپم‚Œم‚’ه…ƒمپ®éژ–مپ«çµگهگˆمپ•مپ›م‚‹مپ“مپ¨مپ§ç›¸è£œéژ–م‚’ن½œوˆگمپ™م‚‹م€‚DNAمƒمƒھمƒ،مƒ©مƒ¼م‚¼مپ¯DNAéژ–م‚’5'مپ‹م‚‰3'مپ®و–¹هگ‘مپ«مپ—مپ‹ن¼¸é•·مپ§مپچمپھمپ„مپںم‚پم€پن؛Œé‡چم‚‰مپ›م‚“مپ®é€†ه¹³è،Œéژ–م‚’複製مپ™م‚‹مپںم‚پمپ«ç•°مپھم‚‹و©ںو§‹مپŒن½؟م‚ڈم‚Œم‚‹[105]م€‚مپ“مپ®م‚ˆمپ†مپ«مپ—مپ¦م€پهڈ¤مپ„éژ–مپ®ه،©هں؛مپŒو–°مپ—مپ„éژ–مپ®ه،©هں؛م‚’و±؛ه®ڑمپ—م€پç´°èƒمپ¯مپمپ®DNAمپ®ه®Œه…¨مپھ複製م‚’ه¾—م‚‹مپ“مپ¨مپŒمپ§مپچم‚‹م€‚ ç´°èƒه¤–و ¸é…¸è£¸مپ®ç´°èƒه¤–DNA(英: extracellular DNAم€پeDNA)مپ¯م€پمپمپ®مپ»مپ¨م‚“مپ©مپŒç´°èƒو»مپ®éڑ›مپ«و”¾ه‡؛مپ•م‚Œمپںم‚‚مپ®مپ§م€پç’°ه¢ƒن¸مپ«مپ»مپ¼éپچهœ¨مپ—مپ¦مپ„م‚‹م€‚هœںه£Œن¸مپ®و؟ƒه؛¦مپ¯2 خ¼g/Lمپ¨é«کمپڈم€پè‡ھ然مپ®و°´و€§ç’°ه¢ƒن¸مپ§مپ¯88 خ¼g/Lمپ«éپ”مپ™م‚‹مپ“مپ¨م‚‚مپ‚م‚‹[106]م€‚eDNAمپ®هƒچمپچمپ¨مپ—مپ¦م€پéپ؛ن¼هگمپ®و°´ه¹³ن¼و’مپ¸مپ®é–¢ن¸ژ[107]م€پو „é¤ٹç´ مپ®ن¾›çµ¦[108]م€پمپ‚م‚‹مپ„مپ¯م‚¤م‚ھمƒ³م‚„وٹ—ç”ں物è³ھم‚’هڈ–م‚ٹè¾¼م‚“مپ م‚ٹ用é‡ڈم‚’èھ؟و•´مپ™م‚‹مپںم‚پمپ®ç·©è،ه‰¤مپ¨مپ—مپ¦مپ®و©ں能مپھمپ©م€پمپ•مپ¾مپ–مپ¾مپھهڈ¯èƒ½و€§مپŒوڈگو،ˆمپ•م‚Œمپ¦مپ„م‚‹[109]م€‚eDNAمپ¯م€پمپ„مپڈمپ¤مپ‹مپ®ç´°èڈŒç¨®مپ®مƒگم‚¤م‚ھمƒ•م‚£مƒ«مƒ مپ«مپٹمپ„مپ¦م€پو©ں能çڑ„مپھç´°èƒه¤–مƒمƒˆمƒھمƒƒم‚¯م‚¹وˆگهˆ†مپ¨مپ—مپ¦و©ں能مپ™م‚‹م€‚eDNAمپ®هƒچمپچمپ«مپ¯م€پمƒگم‚¤م‚ھمƒ•م‚£مƒ«مƒ ه†…مپ®ç‰¹ه®ڑمپ®ç´°èƒه‹مپ®ن»کç€مپ¨هˆ†و•£م‚’هˆ¶ه¾،مپ™م‚‹èھچèکه› هگمپ¨مپ—مپ¦هƒچمپڈهڈ¯èƒ½و€§م‚„[110]م€پمƒگم‚¤م‚ھمƒ•م‚£مƒ«مƒ ه½¢وˆگمپ«ه¯„ن¸ژمپ™م‚‹هڈ¯èƒ½و€§[111]م€پمپ‚م‚‹مپ„مپ¯مƒگم‚¤م‚ھمƒ•م‚£مƒ«مƒ مپ®ç‰©çگ†çڑ„ه¼·ه؛¦مپ¨ç”ں物ه¦çڑ„م‚¹مƒˆمƒ¬م‚¹مپ«ه¯¾مپ™م‚‹وٹµوٹ—و€§مپ«ه¯„ن¸ژمپ™م‚‹هڈ¯èƒ½و€§مپŒمپ‚م‚‹[112]م€‚ ç„،ç´°èƒèƒژه…گDNAمپ¯و¯چن½“مپ®è،€و¶²ن¸مپ«هکهœ¨مپ—م€پمپمپ®ه،©هں؛é…چهˆ—م‚’و±؛ه®ڑمپ™م‚‹مپ“مپ¨مپ§ç™؛éپ”ن¸مپ®èƒژه…گمپ«é–¢مپ™م‚‹ه¤ڑمپڈمپ®وƒ…ه ±م‚’ه¾—م‚‹مپ“مپ¨مپŒمپ§مپچم‚‹[113]م€‚ ç’°ه¢ƒDNAمپ¨مپ—مپ¦çں¥م‚‰م‚Œم‚‹eDNAمپ¯م€پو°´ن¸م€په¤§و°—ن¸م€پ陸ن¸ٹمپ«مپٹمپ‘م‚‹ç”ں物種مپ®ه‹•مپچمپ¨هکهœ¨م‚’監視مپ—م€پمپمپ®هœ°هںںمپ®ç”ں物ه¤ڑو§کو€§م‚’è©•ن¾،مپ™م‚‹ç”ںو…‹ه¦مپ®èھ؟وں»مƒ„مƒ¼مƒ«مپ¨مپ—مپ¦م€پè‡ھ然科ه¦مپ®هˆ†é‡ژمپ§هˆ©ç”¨مپŒو‹،ه¤§مپ—مپ¦مپ„م‚‹[114][115]م€‚ ه¥½ن¸çگƒç´°èƒه¤–مƒˆمƒ©مƒƒمƒ—→詳細مپ¯م€Œه¥½ن¸çگƒç´°èƒه¤–مƒˆمƒ©مƒƒمƒ—م€چم‚’هڈ‚ç…§
ه¥½ن¸çگƒç´°èƒه¤–مƒˆمƒ©مƒƒمƒ—(英: neutrophil extracellular trapم€پNET)مپ¯م€پن¸»مپ«DNAمپ‹م‚‰و§‹وˆگمپ•م‚Œم‚‹ç´°èƒه¤–ç¹ٹç¶مپ®مƒچمƒƒمƒˆمƒ¯مƒ¼م‚¯مپ§مپ‚م‚ٹم€پ白è،€çگƒمپ®ن¸€ç¨®مپ§مپ‚م‚‹ه¥½ن¸çگƒمپŒه®؟ن¸»ç´°èƒمپ¸مپ®وگچه‚·م‚’وœ€ه°ڈé™گمپ«وٹ‘مپˆمپھمپŒم‚‰ç´°èƒه¤–مپ®ç—…هژںن½“م‚’و®؛و»…مپ™م‚‹مپ“مپ¨م‚’هڈ¯èƒ½مپ«مپ™م‚‹م€‚ م‚؟مƒ³مƒ‘م‚¯è³ھمپ¨مپ®ç›¸ن؛’ن½œç”¨DNAمپ®و©ں能مپ¯مپ™مپ¹مپ¦م‚؟مƒ³مƒ‘م‚¯è³ھمپ¨مپ®ç›¸ن؛’ن½œç”¨مپ«ن¾هکمپ—مپ¦مپ„م‚‹م€‚مپ“م‚Œم‚‰مپ®م‚؟مƒ³مƒ‘م‚¯è³ھ相ن؛’ن½œç”¨مپ¯é特異çڑ„مپ§مپ‚م‚‹مپ“مپ¨م‚‚مپ‚م‚Œمپ°م€پم‚؟مƒ³مƒ‘م‚¯è³ھمپŒهچکن¸€مپ®DNAé…چهˆ—مپ«ç‰¹ç•°çڑ„مپ«çµگهگˆمپ™م‚‹مپ“مپ¨م‚‚مپ‚م‚‹م€‚é…µç´ م‚‚DNAمپ«çµگهگˆمپ™م‚‹مپ“مپ¨مپŒمپ§مپچم€پمپمپ®ن¸مپ§م‚‚特مپ«é‡چè¦پمپھم‚‚مپ®مپ¯م€پ転ه†™مپ¨DNA複製مپ®éڑ›مپ«DNAه،©هں؛é…چهˆ—م‚’م‚³مƒ”مƒ¼مپ™م‚‹مƒمƒھمƒ،مƒ©مƒ¼م‚¼مپ§مپ‚م‚‹م€‚ DNAçµگهگˆم‚؟مƒ³مƒ‘م‚¯è³ھ→詳細مپ¯م€ŒDNAçµگهگˆم‚؟مƒ³مƒ‘م‚¯è³ھم€چم‚’هڈ‚ç…§
 DNAمپ¨çµگهگˆمپ™م‚‹و§‹é€ م‚؟مƒ³مƒ‘م‚¯è³ھمپ¯م€پé特異çڑ„DNA-م‚؟مƒ³مƒ‘م‚¯è³ھ相ن؛’ن½œç”¨مپ®ن¾‹مپ¨مپ—مپ¦م‚ˆمپڈçگ†è§£مپ•م‚Œمپ¦مپ„م‚‹م€‚وں“色ن½“ه†…مپ§DNAمپ¯و§‹é€ م‚؟مƒ³مƒ‘م‚¯è³ھمپ¨è¤‡هگˆن½“م‚’ه½¢وˆگمپ—مپ¦ن؟وŒپمپ•م‚Œمپ¦مپ„م‚‹م€‚مپ“م‚Œم‚‰مپ®م‚؟مƒ³مƒ‘م‚¯è³ھمپ¯DNAم‚’م‚¯مƒمƒمƒپمƒ³ï¼ˆوں“色è³ھ)مپ¨ه‘¼مپ°م‚Œم‚‹ç·»ه¯†مپھو§‹é€ مپ«çµ„ç¹”هŒ–مپ™م‚‹م€‚çœںو ¸ç”ں物مپ§مپ¯م€پمپ“مپ®و§‹é€ مپ¯مƒ’م‚¹مƒˆمƒ³مپ¨مپ„مپ†ه°ڈمپ•مپھه،©هں؛و€§م‚؟مƒ³مƒ‘م‚¯è³ھمپ®è¤‡هگˆن½“مپ«DNAمپŒçµگهگˆمپ—مپںم‚‚مپ®مپ§مپ‚م‚‹مپŒم€پهژںو ¸ç”ں物مپ§مپ¯è¤‡و•°ç¨®é،مپ®م‚؟مƒ³مƒ‘م‚¯è³ھمپŒé–¢ن¸ژمپ—مپ¦مپ„م‚‹[116][117]م€‚مƒ’م‚¹مƒˆمƒ³مپ¯مƒŒم‚¯مƒ¬م‚ھم‚½مƒ¼مƒ مپ¨ه‘¼مپ°م‚Œم‚‹ه††ç›¤çٹ¶مپ®è¤‡هگˆن½“م‚’ه½¢وˆگمپ—م€پمپمپ®è،¨é¢مپ«مپ¯ن؛Œوœ¬éژ–DNAمپŒ2ه‘¨ه®Œه…¨مپ«ه·»مپچمپ¤مپ„مپ¦مپ„م‚‹م€‚مپ“م‚Œم‚‰مپ®é特異çڑ„相ن؛’ن½œç”¨مپ¯م€پمƒ’م‚¹مƒˆمƒ³مپ®ه،©هں؛و€§و®‹هں؛مپŒDNAمپ®é…¸و€§ç³–-مƒھمƒ³é…¸éھ¨و ¼مپ¨م‚¤م‚ھمƒ³çµگهگˆم‚’ه½¢وˆگمپ™م‚‹مپ“مپ¨مپ«م‚ˆمپ£مپ¦ç”ںمپکم‚‹م‚‚مپ®مپ§م€پمپ—مپںمپŒمپ£مپ¦م€په،©هں؛é…چهˆ—مپ¨مپ¯مپ»مپ¨م‚“مپ©ç„،é–¢ن؟‚مپ§مپ‚م‚‹[118]م€‚مپ“م‚Œم‚‰مپ®ه،©هں؛و€§م‚¢مƒںمƒژé…¸و®‹هں؛مپ®هŒ–ه¦ن؟®é£¾مپ«مپ¯م€پمƒ،مƒپمƒ«هŒ–م€پمƒھمƒ³é…¸هŒ–م€پم‚¢م‚»مƒپمƒ«هŒ–مپھمپ©مپŒمپ‚م‚‹[119]م€‚مپ“م‚Œم‚‰مپ®هŒ–ه¦çڑ„ه¤‰هŒ–مپ¯DNAمپ¨مƒ’م‚¹مƒˆمƒ³é–“مپ®ç›¸ن؛’ن½œç”¨مپ®ه¼·ه؛¦م‚’ه¤‰هŒ–مپ•مپ›م€پDNAم‚’転ه†™ه› هگمپ«è؟‘مپ¥مپچم‚„مپ™مپڈمپ—مپںم‚ٹم€پمپ‚م‚‹مپ„مپ¯è؟‘مپ¥مپچمپ«مپڈمپڈمپ—م€پ転ه†™é€ںه؛¦م‚’ه¤‰هŒ–مپ•مپ›م‚‹[120]م€‚م‚¯مƒمƒمƒپمƒ³ه†…مپ®ن»–مپ®é特異çڑ„DNAçµگهگˆم‚؟مƒ³مƒ‘م‚¯è³ھمپ«مپ¯م€پو›²مپŒمپ£مپںDNAم‚„وھم‚“مپ DNAمپ«çµگهگˆمپ™م‚‹é«ک移ه‹•ه؛¦éƒ،م‚؟مƒ³مƒ‘م‚¯è³ھمپŒمپ‚م‚‹[121]م€‚مپ“م‚Œم‚‰مپ®م‚؟مƒ³مƒ‘م‚¯è³ھمپ¯م€پمƒŒم‚¯مƒ¬م‚ھم‚½مƒ¼مƒ مپ®é…چهˆ—م‚’و›²مپ’مپںم‚ٹم€پوں“色ن½“م‚’و§‹وˆگمپ™م‚‹ه¤§مپچمپھو§‹é€ ن½“م‚’組مپ؟ç«‹مپ¦م‚‹éڑ›مپ«é‡چè¦پمپ§مپ‚م‚‹[122]م€‚ DNAçµگهگˆم‚؟مƒ³مƒ‘م‚¯è³ھمپ®م‚‚مپ†ن¸€مپ¤مپ®م‚°مƒ«مƒ¼مƒ—مپ¨مپ—مپ¦م€پن¸€وœ¬éژ–DNAمپ¨ç‰¹ç•°çڑ„مپ«çµگهگˆمپ™م‚‹DNAçµگهگˆم‚؟مƒ³مƒ‘م‚¯è³ھمپŒمپ‚م‚‹م€‚مƒ’مƒˆمپ®ه ´هگˆم€پ複製م‚؟مƒ³مƒ‘م‚¯è³ھAمپŒمپ“مپ®ن¸€ç¾¤مپ®ن¸مپ§وœ€م‚‚م‚ˆمپڈçگ†è§£مپ•م‚Œمپ¦مپٹم‚ٹم€پDNA複製م€پ組وڈ›مپˆم€پDNAن؟®ه¾©مپھمپ©م€پن؛Œé‡چم‚‰مپ›م‚“مپŒهˆ†é›¢مپ™م‚‹مƒ—مƒم‚»م‚¹مپ«é–¢ن¸ژمپ—مپ¦مپ„م‚‹[123]م€‚مپ“م‚Œم‚‰مپ®çµگهگˆم‚؟مƒ³مƒ‘م‚¯è³ھمپ¯ن¸€وœ¬éژ–DNAم‚’ه®‰ه®ڑهŒ–مپ•مپ›م€پم‚¹مƒ†مƒ مƒ«مƒ¼مƒ—م‚’ه½¢وˆگمپ—مپںم‚ٹم€پمƒŒم‚¯مƒ¬م‚¢مƒ¼م‚¼مپ«م‚ˆم‚‹هˆ†è§£مپ‹م‚‰DNAم‚’ن؟è·مپ—مپ¦مپ„م‚‹مپ¨è€ƒمپˆم‚‰م‚Œمپ¦مپ„م‚‹م€‚  ه¯¾ç…§çڑ„مپ«م€پن»–مپ®م‚؟مƒ³مƒ‘م‚¯è³ھمپ¯ç‰¹ه®ڑمپ®DNAé…چهˆ—مپ«çµگهگˆمپ™م‚‹م‚ˆمپ†مپھ進هŒ–م‚’مپ—مپ¦مپچمپںم€‚وœ€م‚‚ç ”ç©¶مپŒé€²م‚“مپ§مپ„م‚‹مپ®مپ¯م€پ転ه†™م‚’هˆ¶ه¾،مپ™م‚‹م‚؟مƒ³مƒ‘م‚¯è³ھمپ§مپ‚م‚‹مپ•مپ¾مپ–مپ¾مپھ転ه†™ه› هگمپ§مپ‚م‚‹م€‚هگ„転ه†™ه› هگمپ¯مƒ—مƒمƒ¢مƒ¼م‚؟مƒ¼è؟‘مپڈمپ®ç‰¹ه®ڑمپ®DNAé…چهˆ—مپ«çµگهگˆمپ—م€پéپ؛ن¼هگمپ®è»¢ه†™م‚’و´»و€§هŒ–مپ¾مپںمپ¯éک»ه®³مپ™م‚‹م€‚転ه†™ه› هگمپ¯2مپ¤مپ®و–¹و³•مپ§مپ“م‚Œم‚’è،Œمپ†م€‚ن¸€مپ¤مپ¯م€پ転ه†™م‚’و‹…مپ†RNAمƒمƒھمƒ،مƒ©مƒ¼م‚¼مپ«ç›´وژ¥م€پمپ‚م‚‹مپ„مپ¯ن»–مپ®هھ’ن»‹م‚؟مƒ³مƒ‘م‚¯è³ھم‚’ن»‹مپ—مپ¦çµگهگˆمپ™م‚‹مپ“مپ¨مپ§مپ‚م‚‹م€‚مپ“م‚Œمپ«م‚ˆمپ£مپ¦م€پمƒمƒھمƒ،مƒ©مƒ¼م‚¼مپ¯مƒ—مƒمƒ¢مƒ¼م‚؟مƒ¼مپ«ن½چç½®مپ—م€پ転ه†™م‚’é–‹ه§‹مپ™م‚‹مپ“مپ¨مپŒمپ§مپچم‚‹[125]م€‚مپ‚م‚‹مپ„مپ¯م€پ転ه†™ه› هگمپ¯مƒ—مƒمƒ¢مƒ¼م‚؟مƒ¼مپ®مƒ’م‚¹مƒˆمƒ³م‚’ن؟®é£¾مپ™م‚‹é…µç´ مپ¨çµگهگˆمپ™م‚‹مپ“مپ¨مپŒمپ§مپچم‚‹م€‚مپ“م‚Œمپ«م‚ˆمپ£مپ¦DNA鋳ه‹مپ«ه¯¾مپ™م‚‹مƒمƒھمƒ،مƒ©مƒ¼م‚¼مپ®è؟‘مپ¥مپچم‚„مپ™مپ•م‚’ه¤‰هŒ–مپ•مپ›م‚‹[126]م€‚ مپ“م‚Œم‚‰مپ®DNAو¨™çڑ„مپ¯ç”ں物مپ®م‚²مƒژمƒ ه…¨ن½“مپ«هکهœ¨مپ™م‚‹هڈ¯èƒ½و€§مپŒمپ‚م‚‹مپںم‚پم€پن¸€ç¨®é،مپ®è»¢ه†™ه› هگمپ®و´»و€§مپŒه¤‰هŒ–مپ™م‚‹مپ¨م€پن½•هچƒم‚‚مپ®éپ؛ن¼هگمپ«ه½±éں؟م‚’هڈٹمپ¼مپ™هڈ¯èƒ½و€§مپŒمپ‚م‚‹[127]م€‚مپمپ®çµگوœم€پمپ“م‚Œم‚‰مپ®م‚؟مƒ³مƒ‘م‚¯è³ھمپ¯مپ—مپ°مپ—مپ°م€پç’°ه¢ƒه¤‰هŒ–مپ¸مپ®ه؟œç”م‚„ç´°èƒمپ®هˆ†هŒ–مƒ»ç™؛éپ”م‚’هˆ¶ه¾،مپ™م‚‹م‚·م‚°مƒٹمƒ«ن¼éپ”مƒ—مƒم‚»م‚¹مپ®و¨™çڑ„مپ¨مپھم‚‹م€‚مپ“م‚Œم‚‰مپ®è»¢ه†™ه› هگمپ®DNAمپ¨مپ®ç›¸ن؛’ن½œç”¨مپ®ç‰¹ç•°و€§مپ¯م€پم‚؟مƒ³مƒ‘م‚¯è³ھمپŒDNAه،©هں؛مپ®ç«¯مپ¨ن½•ه؛¦م‚‚وژ¥è§¦مپ—مپ¦م€پDNAé…چهˆ—م‚’م€Œèھمپ؟هڈ–م‚‹م€چمپ“مپ¨م‚’هڈ¯èƒ½مپ«مپ™م‚‹مپ“مپ¨مپ§ç”ںمپکم‚‹م€‚مپ“م‚Œم‚‰مپ®ه،©هں؛相ن؛’ن½œç”¨مپ®مپ»مپ¨م‚“مپ©مپ¯ه،©هں؛مپŒوœ€م‚‚وژ¥è؟‘مپ—م‚„مپ™مپ„ن¸»و؛مپ§èµ·مپ“م‚‹[25]م€‚  DNAن؟®é£¾é…µç´مƒŒم‚¯مƒ¬م‚¢مƒ¼م‚¼مپ¨مƒھم‚¬مƒ¼م‚¼مƒŒم‚¯مƒ¬م‚¢مƒ¼م‚¼مپ¯م€پمƒ›م‚¹مƒ›م‚¸م‚¨م‚¹مƒ†مƒ«çµگهگˆمپ®هٹ و°´هˆ†è§£م‚’触هھ’مپ™م‚‹مپ“مپ¨مپ«م‚ˆمپ£مپ¦DNAéژ–م‚’هˆ‡و–مپ™م‚‹é…µç´ مپ§مپ‚م‚‹م€‚DNAéژ–مپ®وœ«ç«¯مپ‹م‚‰مƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰م‚’هٹ و°´هˆ†è§£مپ™م‚‹مƒŒم‚¯مƒ¬م‚¢مƒ¼م‚¼مپ¯م‚¨م‚م‚½مƒŒم‚¯مƒ¬م‚¢مƒ¼م‚¼مپ¨ه‘¼مپ°م‚Œم€پن¸€و–¹م‚¨مƒ³مƒ‰مƒŒم‚¯مƒ¬م‚¢مƒ¼م‚¼مپ¯éژ–ه†…مپ§هˆ‡و–مپ™م‚‹م€‚هˆ†هگç”ں物ه¦مپ§وœ€م‚‚م‚ˆمپڈن½؟用مپ•م‚Œم‚‹مƒŒم‚¯مƒ¬م‚¢مƒ¼م‚¼مپ¯م€پ特異çڑ„é…چهˆ—مپ§DNAم‚’هˆ‡و–مپ™م‚‹هˆ¶é™گم‚¨مƒ³مƒ‰مƒŒم‚¯مƒ¬م‚¢مƒ¼م‚¼مپ§مپ‚م‚‹م€‚مپںمپ¨مپˆمپ°م€پن¸ٹه›³مپ«ç¤؛مپ—مپںEcoRVé…µç´ مپ¯م€پDNAéژ–مپ®6ه،©هں؛é…چهˆ— 5′-GATATC-3′ م‚’èھچèکمپ—م€پو°´ه¹³ç·ڑمپ§هˆ‡و–مپ™م‚‹م€‚è‡ھ然界مپ§مپ“م‚Œم‚‰مپ®é…µç´ مپ¯م€پهˆ¶é™گن؟®é£¾ç³»مپ®ن¸€éƒ¨مپ¨مپ—مپ¦ç´°èڈŒمپ®ç´°èƒه†…مپ«ن¾µه…¥مپ—مپںمƒ•م‚،مƒ¼م‚¸DNAم‚’و¶ˆهŒ–مپ™م‚‹مپ“مپ¨مپ«م‚ˆم‚ٹم€پç´°èڈŒم‚’مƒ•م‚،مƒ¼م‚¸و„ںوں“مپ‹م‚‰ن؟è·مپ—مپ¦مپ„م‚‹[129]م€‚وٹ€è،“هˆ†é‡ژمپ§مپ¯م€پمپ“م‚Œم‚‰مپ®é…چهˆ—特異çڑ„مƒŒم‚¯مƒ¬م‚¢مƒ¼م‚¼مپ¯هˆ†هگم‚¯مƒمƒ¼مƒ‹مƒ³م‚°م‚„DNAمƒ—مƒمƒ•م‚،م‚¤مƒھمƒ³م‚°مپ«ن½؟用مپ•م‚Œمپ¦مپ„م‚‹م€‚ DNAمƒھم‚¬مƒ¼م‚¼مپ¨ه‘¼مپ°م‚Œم‚‹é…µç´ مپ¯م€پهˆ‡و–مپ¾مپںمپ¯ç ´وگچمپ—مپںDNAéژ–م‚’ه†چçµگهگˆمپ•مپ›م‚‹مپ“مپ¨مپŒمپ§مپچم‚‹[130]م€‚مƒھم‚¬مƒ¼م‚¼مپ¯م€پمƒ©م‚®مƒ³م‚°éژ–DNA複製مپ«مپٹمپ„مپ¦ç‰¹مپ«é‡چè¦پمپ§م€پ複製مƒ•م‚©مƒ¼م‚¯مپ§ن½œم‚‰م‚Œمپںçںمپ„DNAم‚»م‚°مƒ،مƒ³مƒˆم‚’DNA鋳ه‹مپ®ه®Œه…¨مپھم‚³مƒ”مƒ¼مپ«çµگهگˆمپ™م‚‹هƒچمپچم‚’مپ™م‚‹م€‚مپ“م‚Œم‚‰مپ¯مپ¾مپںDNAن؟®ه¾©م‚„éپ؛ن¼çڑ„組وڈ›مپˆمپ«م‚‚ن½؟用مپ•م‚Œم‚‹[130]م€‚ مƒˆمƒم‚¤م‚½مƒ،مƒ©مƒ¼م‚¼مپ¨مƒکمƒھم‚«مƒ¼م‚¼مƒˆمƒم‚¤م‚½مƒ،مƒ©مƒ¼م‚¼مپ¯مƒŒم‚¯مƒ¬م‚¢مƒ¼م‚¼مپ¨مƒھم‚¬مƒ¼م‚¼مپ®ن¸،و–¹مپ®و´»و€§م‚’وŒپمپ¤é…µç´ مپ§مپ‚م‚‹م€‚مپ“م‚Œم‚‰مپ®م‚؟مƒ³مƒ‘م‚¯è³ھمپ¯DNAم‚¹مƒ¼مƒ‘مƒ¼م‚³م‚¤مƒ«مپ®é‡ڈم‚’ه¤‰هŒ–مپ•مپ›م‚‹م€‚مپ“م‚Œم‚‰مپ®é…µç´ مپ®ن¸مپ«مپ¯م€پDNAم‚‰مپ›م‚“م‚’هˆ‡و–مپ—م€پمپمپ®ن¸€éƒ¨هˆ†م‚’ه›è»¢مپ•مپ›م‚‹مپ“مپ¨مپ§م‚¹مƒ¼مƒ‘مƒ¼م‚³م‚¤مƒ«مپ®مپ²مپڑمپ؟م‚’ن½ژو¸›مپ•مپ›م€پمپمپ®ه¾ŒDNAمپ®هˆ‡و–部م‚’ه°پç€مپ™م‚‹م‚‚مپ®م‚‚مپ‚م‚‹[44]م€‚هˆ¥مپ®ç¨®é،مپ®é…µç´ مپ¯م€پDNAم‚‰مپ›م‚“م‚’هˆ‡و–مپ—م€پمپمپ®هˆ‡و–部هˆ†مپ«2وœ¬ç›®مپ®DNAم‚’é€ڑéپژمپ•مپ›مپ¦مپ‹م‚‰م€پم‚‰مپ›م‚“م‚’ه†چçµگهگˆمپ™م‚‹مپ“مپ¨مپŒمپ§مپچم‚‹[131]م€‚مپ“مپ®م‚ˆمپ†مپ«مƒˆمƒم‚¤م‚½مƒ،مƒ©مƒ¼م‚¼مپ¯م€پDNA複製م‚„転ه†™مپھمپ©م€پDNAمپŒé–¢ن¸ژمپ™م‚‹ه¤ڑمپڈمپ®éپژ程مپ«ه؟…è¦پمپھé…µç´ مپ§مپ‚م‚‹[45]م€‚ مƒکمƒھم‚«مƒ¼م‚¼مپ¯هˆ†هگمƒ¢مƒ¼م‚؟مƒ¼مپ¨مپ—مپ¦هƒچمپڈم‚؟مƒ³مƒ‘م‚¯è³ھمپ§مپ‚م‚‹م€‚مپ“م‚Œم‚‰مپ¯م€پمƒŒم‚¯مƒ¬م‚ھم‚·مƒ‰ن¸‰مƒھمƒ³é…¸م€پن¸»مپ«م‚¢مƒ‡مƒژم‚·مƒ³ن¸‰مƒھمƒ³é…¸ï¼ˆATP)مپ®هŒ–ه¦م‚¨مƒچمƒ«م‚®مƒ¼م‚’هˆ©ç”¨مپ—مپ¦م€په،©هں؛é–“مپ®و°´ç´ çµگهگˆم‚’هˆ‡و–مپ—م€پDNAن؛Œé‡چم‚‰مپ›م‚“م‚’مپ»مپ©مپ„مپ¦ن¸€وœ¬éژ–مپ«مپ™م‚‹[132]م€‚مپ“م‚Œم‚‰مپ®é…µç´ مپ¯م€پé…µç´ مپŒDNAه،©هں؛مپ«è؟‘وژ¥مپ™م‚‹ه؟…è¦پمپŒمپ‚م‚‹مپ»مپ¨م‚“مپ©مپ®éپژ程مپ«مپ¨مپ£مپ¦ن¸چهڈ¯و¬ مپ§مپ‚م‚‹م€‚ مƒمƒھمƒ،مƒ©مƒ¼م‚¼مƒمƒھمƒ،مƒ©مƒ¼م‚¼مپ¯مƒŒم‚¯مƒ¬م‚ھم‚·مƒ‰ن¸‰مƒھمƒ³é…¸مپ‹م‚‰مƒمƒھمƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰éژ–م‚’هگˆوˆگمپ™م‚‹é…µç´ مپ§مپ‚م‚‹م€‚مپمپ®ç”ںوˆگ物مپ®é…چهˆ—مپ¯م€پ鋳ه‹ï¼ˆè‹±: template)مپ¨ه‘¼مپ°م‚Œم‚‹و—¢هکمپ®مƒمƒھمƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰éژ–مپ«هں؛مپ¥مپ„مپ¦ن½œم‚‰م‚Œم‚‹م€‚مپ“م‚Œم‚‰مپ®é…µç´ مپ¯م€پن¼¸é•·مپ™م‚‹مƒمƒھمƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰éژ–وœ«ç«¯مپ®3'مƒ’مƒ‰مƒم‚م‚·هں؛مپ«ç¹°م‚ٹè؟”مپ—مƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰م‚’ن»کهٹ مپ™م‚‹و©ں能م‚’وŒپمپ¤م€‚çµگوœمپ¨مپ—مپ¦مپ™مپ¹مپ¦مپ®مƒمƒھمƒ،مƒ©مƒ¼م‚¼مپ¯5'مپ‹م‚‰3'مپ®و–¹هگ‘مپ«هƒچمپڈ[133]م€‚مپ“م‚Œم‚‰مپ®é…µç´ مپ®و´»و€§éƒ¨ن½چمپ§مپ¯م€په…¥مپ£مپ¦مپچمپںمƒŒم‚¯مƒ¬م‚ھم‚·مƒ‰ن¸‰مƒھمƒ³é…¸مپŒé‹³ه‹مپ¨ه،©هں؛ه¯¾م‚’ه½¢وˆگمپ™م‚‹م€‚مپ“م‚Œمپ«م‚ˆم‚ٹم€پمƒمƒھمƒ،مƒ©مƒ¼م‚¼مپ¯é‹³ه‹مپ®ç›¸è£œéژ–م‚’و£ç¢؛مپ«هگˆوˆگمپ™م‚‹مپ“مپ¨مپŒمپ§مپچم‚‹م€‚مƒمƒھمƒ،مƒ©مƒ¼م‚¼مپ¯م€پن½؟用مپ™م‚‹é‹³ه‹مپ®ç¨®é،مپ«م‚ˆمپ£مپ¦هˆ†é،مپ•م‚Œم‚‹م€‚ DNA複製مپ¯م€پDNAن¾هکو€§DNAمƒمƒھمƒ،مƒ©مƒ¼م‚¼مپŒDNAمƒمƒھمƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰éژ–مپ®م‚³مƒ”مƒ¼م‚’ن½œم‚‹م€‚ç”ں物ه¦çڑ„وƒ…ه ±م‚’ن؟هکمپ™م‚‹مپںم‚پمپ«مپ¯م€پهگ„م‚³مƒ”مƒ¼مپ®ه،©هں؛é…چهˆ—مپŒé‹³ه‹éژ–مپ®ه،©هں؛é…چهˆ—مپ¨و£ç¢؛مپ«ç›¸è£œçڑ„مپ§مپ‚م‚‹مپ“مپ¨مپŒن¸چهڈ¯و¬ مپ§مپ‚م‚‹م€‚ه¤ڑمپڈمپ®DNAمƒمƒھمƒ،مƒ©مƒ¼م‚¼مپ¯و ،و£و´»و€§م‚’وŒپمپ£مپ¦مپ„م‚‹م€‚مپ“م‚Œمپ«م‚ˆم‚ٹمƒمƒھمƒ،مƒ©مƒ¼م‚¼مپ¯م€پمƒںم‚¹مƒمƒƒمƒپمپ—مپںمƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰é–“مپ§مپ®ه،©هں؛ه¯¾ه½¢وˆگمپ®و¬ ه¦‚مپ«م‚ˆمپ£مپ¦م€پهگˆوˆگهڈچه؟œمپ®éڑ›مپ«مپ¨مپچمپٹم‚ٹèµ·مپ“م‚‹èھ¤م‚ٹم‚’و¤œه‡؛مپ™م‚‹مپ“مپ¨مپŒمپ§مپچم‚‹م€‚مƒںم‚¹مƒمƒƒمƒپمپŒو¤œه‡؛مپ•م‚Œم‚‹مپ¨م€پ3'→5'م‚¨م‚م‚½مƒŒم‚¯مƒ¬م‚¢مƒ¼م‚¼و´»و€§مپŒو´»و€§هŒ–مپ•م‚Œم€پèھ¤مپ£مپںه،©هں؛مپŒé™¤هژ»مپ•م‚Œم‚‹[134]م€‚مپ»مپ¨م‚“مپ©مپ®ç”ں物مپ§DNAمƒمƒھمƒ،مƒ©مƒ¼م‚¼مپ¯م€پDNAم‚¯مƒ©مƒ³مƒ—م‚„مƒکمƒھم‚«مƒ¼م‚¼مپھمپ©مپ®è¤‡و•°مپ®م‚¢م‚¯م‚»م‚µمƒھمƒ¼مƒ»م‚µمƒ–مƒ¦مƒ‹مƒƒمƒˆم‚’هگ«م‚€م€پمƒ¬مƒ—مƒھم‚½مƒ¼مƒ مپ¨ه‘¼مپ°م‚Œم‚‹ه¤§مپچمپھ複هگˆن½“مپ®ن¸مپ§و©ں能مپ™م‚‹[135]م€‚ RNAن¾هکو€§DNAمƒمƒھمƒ،مƒ©مƒ¼م‚¼مپ¯م€پRNAéژ–مپ®ه،©هں؛é…چهˆ—م‚’DNAمپ«م‚³مƒ”مƒ¼مپ™م‚‹ç‰¹و®ٹمپھمƒمƒھمƒ،مƒ©مƒ¼م‚¼مپ§مپ‚م‚‹م€‚مپ“م‚Œم‚‰مپ«مپ¯م€پمƒ¬مƒˆمƒم‚¦م‚¤مƒ«م‚¹مپ«م‚ˆم‚‹ç´°èƒو„ںوں“مپ«é–¢ن¸ژمپ™م‚‹م‚¦م‚¤مƒ«م‚¹و€§é…µç´ مپ§مپ‚م‚‹é€†è»¢ه†™é…µç´ م‚„م€پمƒ†مƒمƒ،م‚¢مپ®è¤‡è£½مپ«ه؟…è¦پمپھمƒ†مƒمƒ،مƒ©مƒ¼م‚¼مپŒهگ«مپ¾م‚Œم‚‹[63][136]م€‚مپںمپ¨مپˆمپ°م€پHIV逆転ه†™é…µç´ مپ¯م€پم‚¨م‚¤م‚؛م‚¦م‚¤مƒ«م‚¹مپ®è¤‡è£½مپ«é–¢ن¸ژمپ™م‚‹é…µç´ مپ§مپ‚م‚‹[136]م€‚مƒ†مƒمƒ،مƒ©مƒ¼م‚¼مپ¯م€پمپمپ®و§‹é€ مپ®ن¸€éƒ¨مپ¨مپ—مپ¦è‡ھè؛«مپ®RNA鋳ه‹م‚’هگ«م‚€مپ¨مپ„مپ†çڈچمپ—مپ„مƒمƒھمƒ،مƒ©مƒ¼م‚¼مپ§مپ‚م‚‹م€‚مپ“م‚Œمپ¯وں“色ن½“مپ®وœ«ç«¯مپ«مƒ†مƒمƒ،م‚¢م‚’هگˆوˆگمپ™م‚‹م€‚مƒ†مƒمƒ،م‚¢مپ¯éڑ£وژ¥مپ™م‚‹وں“色ن½“وœ«ç«¯مپŒèچهگˆمپ™م‚‹مپ®م‚’éک²مپژم€پوں“色ن½“وœ«ç«¯م‚’وگچه‚·مپ‹م‚‰ن؟è·مپ™م‚‹[64]م€‚ 転ه†™مپ¯م€پDNAéژ–مپ®é…چهˆ—م‚’RNAمپ«م‚³مƒ”مƒ¼مپ™م‚‹DNAن¾هکو€§RNAمƒمƒھمƒ،مƒ©مƒ¼م‚¼مپ«م‚ˆمپ£مپ¦è،Œم‚ڈم‚Œم‚‹م€‚éپ؛ن¼هگمپ®è»¢ه†™م‚’é–‹ه§‹مپ™م‚‹مپںم‚پمپ«م€پRNAمƒمƒھمƒ،مƒ©مƒ¼م‚¼مپ¯مƒ—مƒمƒ¢مƒ¼م‚؟مƒ¼مپ¨ه‘¼مپ°م‚Œم‚‹DNAé…چهˆ—مپ«çµگهگˆمپ—م€پDNAéژ–م‚’هˆ†é›¢مپ™م‚‹م€‚مپمپ®ه¾Œم€پم‚؟مƒ¼مƒںمƒچمƒ¼م‚؟مƒ¼مپ¨ه‘¼مپ°م‚Œم‚‹DNAمپ®é کهںںمپ«هˆ°éپ”مپ™م‚‹مپ¾مپ§م€پéپ؛ن¼هگé…چهˆ—م‚’مƒ،مƒƒم‚»مƒ³م‚¸مƒ£مƒ¼RNA転ه†™ç‰©مپ«م‚³مƒ”مƒ¼مپ—م€پمپمپ“مپ§هپœو¢مپ—مپ¦DNAمپ‹م‚‰هˆ†é›¢مپ™م‚‹م€‚مƒ’مƒˆمپ®DNAن¾هکو€§DNAمƒمƒھمƒ،مƒ©مƒ¼م‚¼مپ¨هگŒو§کمپ«م€پمƒ’مƒˆم‚²مƒژمƒ مپ®مپ»مپ¨م‚“مپ©مپ®éپ؛ن¼هگم‚’転ه†™مپ™م‚‹é…µç´ مپ§مپ‚م‚‹RNAمƒمƒھمƒ،مƒ©مƒ¼م‚¼IIمپ¯م€پمپ„مپڈمپ¤مپ‹èھ؟節م‚µمƒ–مƒ¦مƒ‹مƒƒمƒˆمپ¨م‚¢م‚¯م‚»م‚µمƒھمƒ¼م‚µمƒ–مƒ¦مƒ‹مƒƒمƒˆم‚’وŒپمپ¤ه¤§مپچمپھم‚؟مƒ³مƒ‘م‚¯è³ھ複هگˆن½“مپ®ن¸€éƒ¨مپ¨مپ—مپ¦هƒچمپ„مپ¦مپ„م‚‹[137]م€‚ éپ؛ن¼هگ組وڈ›مپˆ
→詳細مپ¯م€Œéپ؛ن¼çڑ„組وڈ›مپˆم€چم‚’هڈ‚ç…§
 DNAم‚‰مپ›م‚“مپ¯é€ڑه¸¸م€پن»–مپ®DNAم‚»م‚°مƒ،مƒ³مƒˆمپ¨ç›¸ن؛’ن½œç”¨مپ™م‚‹مپ“مپ¨مپ¯مپھمپڈم€پمƒ’مƒˆمپ®ç´°èƒمپ§مپ¯م€پç•°مپھم‚‹وں“色ن½“مپ¯وں“色ن½“مƒ†مƒھمƒˆمƒھمƒ¼ï¼ˆè‹±: chromosome territories)مپ¨ه‘¼مپ°م‚Œم‚‹و ¸ه†…مپ®هˆ¥م€…مپ®é کهںںم‚’هچ م‚پم‚‹مپ“مپ¨مپ•مپˆمپ‚م‚‹[139]م€‚مپ“مپ®م‚ˆمپ†مپ«ç•°مپھم‚‹وں“色ن½“مپŒç‰©çگ†çڑ„مپ«هˆ†é›¢مپ—مپ¦مپ„م‚‹مپ“مپ¨مپ¯م€پDNAمپŒه®‰ه®ڑمپ—مپںوƒ…ه ±ن؟ç®،ه ´و‰€مپ¨مپ—مپ¦و©ں能مپ™م‚‹مپںم‚پمپ«é‡چè¦پمپ§مپ‚م‚‹م€‚مپھمپœمپھم‚‰م€پوں“色ن½“مپŒç›¸ن؛’ن½œç”¨مپ™م‚‹و•°ه°‘مپھمپ„و©ںن¼ڑمپ®مپ²مپ¨مپ¤مپŒم€پوœ‰و€§ç”ںو®–مپ®éڑ›مپ«èµ·مپ“م‚‹وں“色ن½“ن؛¤ه·®ï¼ˆè‹±: chromosomal crossover)مپ§مپ‚م‚ٹم€پمپمپ®éڑ›مپ«éپ؛ن¼çڑ„組وڈ›مپˆمپŒèµ·مپ“م‚‹مپ‹م‚‰مپ§مپ‚م‚‹م€‚وں“色ن½“ن؛¤ه·®مپ¨مپ¯م€پDNAمپ®2وœ¬مپ®م‚‰مپ›م‚“مپŒهˆ‡و–مپ•م‚Œم€پن¸€éƒ¨مپŒه…¥م‚Œو›؟م‚ڈم‚ٹم€په†چمپ³çµگهگˆمپ™م‚‹مپ“مپ¨مپ§مپ‚م‚‹م€‚ 組وڈ›مپˆمپ¯م€پوں“色ن½“مپŒéپ؛ن¼وƒ…ه ±م‚’ن؛¤وڈ›مپ—مپ¦éپ؛ن¼هگمپ®و–°مپ—مپ„組مپ؟هگˆم‚ڈمپ›م‚’ن½œم‚ٹه‡؛مپ™مپ“مپ¨م‚’هڈ¯èƒ½مپ«مپ—م€پمپ“م‚Œمپ«م‚ˆم‚ٹè‡ھ然éپ¸وٹمپ®هٹ¹çژ‡م‚’é«کم‚پم€پو–°مپ—مپ„م‚؟مƒ³مƒ‘م‚¯è³ھمپ®و€¥é€ںمپھ進هŒ–مپ«مپٹمپ„مپ¦é‡چè¦پمپ§مپ‚م‚‹[140]م€‚éپ؛ن¼çڑ„組وڈ›مپˆمپ¯DNAن؟®ه¾©م€پ特مپ«ن؛Œوœ¬éژ–هˆ‡و–مپ«ه¯¾مپ™م‚‹ç´°èƒمپ®هڈچه؟œمپ«م‚‚é–¢ن¸ژمپ—مپ¦مپ„م‚‹هڈ¯èƒ½و€§مپŒمپ‚م‚‹[141]م€‚ وں“色ن½“ن؛¤ه·®مپ®وœ€م‚‚ن¸€èˆ¬çڑ„مپھه½¢و…‹مپ¯ç›¸هگŒçµ„وڈ›مپˆمپ§م€پé–¢ن¸ژمپ™م‚‹2مپ¤مپ®وں“色ن½“مپ®é…چهˆ—مپ¯éه¸¸مپ«م‚ˆمپڈن¼¼مپ¦مپ„م‚‹م€‚é相هگŒçµ„وڈ›مپˆمپ¯م€پوں“色ن½“転ه؛§م‚„éپ؛ن¼çڑ„ç•°ه¸¸م‚’ç”ںمپکمپ•مپ›م‚‹مپںم‚پم€پç´°èƒمپ«وگچه‚·م‚’ن¸ژمپˆم‚‹هڈ¯èƒ½و€§مپŒمپ‚م‚‹م€‚組وڈ›مپˆهڈچه؟œمپ¯م€پRAD51مپ®م‚ˆمپ†مپھمƒھم‚³مƒ³مƒ“مƒٹمƒ¼م‚¼مپ¨مپ—مپ¦çں¥م‚‰م‚Œم‚‹é…µç´ مپ«م‚ˆمپ£مپ¦è§¦هھ’مپ•م‚Œم‚‹[142]م€‚組وڈ›مپˆمپ®وœ€هˆمپ®و®µéڑژمپ¯م€پم‚¨مƒ³مƒ‰مƒŒم‚¯مƒ¬م‚¢مƒ¼م‚¼مپ‹DNAمپ®وگچه‚·مپ«م‚ˆمپ£مپ¦ه¼•مپچèµ·مپ“مپ•م‚Œم‚‹ن؛Œوœ¬éژ–هˆ‡و–مپ§مپ‚م‚‹[143]م€‚مپمپ®ه¾Œم€پمƒھم‚³مƒ³مƒ“مƒٹمƒ¼م‚¼مپ«م‚ˆمپ£مپ¦éƒ¨هˆ†çڑ„مپ«è§¦هھ’مپ•م‚Œم‚‹ن¸€é€£مپ®و®µéڑژمپ«م‚ˆمپ£مپ¦م€پ2مپ¤مپ®م‚‰مپ›م‚“مپ¯ه°‘مپھمپڈمپ¨م‚‚1مپ¤مپ®مƒ›مƒھمƒ‡م‚¤م‚¸مƒ£مƒ³م‚¯م‚·مƒ§مƒ³مپ«م‚ˆمپ£مپ¦çµگهگˆمپ•م‚Œم€پو¬،مپ«م€پهگ„م‚‰مپ›م‚“ن¸مپ®ن¸€وœ¬éژ–م‚»م‚°مƒ،مƒ³مƒˆمپŒن»–و–¹مپ®م‚‰مپ›م‚“مپ®ç›¸è£œéژ–مپ¨ن؛Œوœ¬éژ–م‚’ه½¢وˆگمپ™م‚‹م€‚مƒ›مƒھمƒ‡م‚¤م‚¸مƒ£مƒ³م‚¯م‚·مƒ§مƒ³مپ¯ه››é¢ن½“مپ®وژ¥هگˆو§‹é€ مپ§م€پوں“色ن½“ه¯¾مپ«و²؟مپ£مپ¦ç§»ه‹•مپ™م‚‹مپ“مپ¨مپŒمپ§مپچم€پن¸€و–¹مپ®éژ–م‚’م‚‚مپ†ن¸€و–¹مپ®éژ–مپ¨ن؛¤وڈ›مپ™م‚‹مپ“مپ¨مپŒمپ§مپچم‚‹م€‚組وڈ›مپˆهڈچه؟œمپ¯م€پçµگهگˆéƒ¨مپ®هˆ‡و–مپ¨éپٹ離مپ—مپںDNAمپ®ه†چçµگهگˆمپ«م‚ˆمپ£مپ¦هپœو¢مپ™م‚‹[144]م€‚組وڈ›مپˆمپ®éڑ›مپ«هگŒمپکو–¹هگ‘و€§ï¼ˆو¥µو€§ï¼‰مپ®éژ–مپ مپ‘مپŒDNAم‚’ن؛¤وڈ›مپ™م‚‹م€‚هˆ‡و–مپ«مپ¯و±è¥؟هˆ‡و–(east-west cleavage)مپ¨هچ—هŒ—هˆ‡و–(north–south cleavage)مپ®2種é،مپŒمپ‚م‚‹م€‚هچ—هŒ—هˆ‡و–مپ¯DNAمپ®ن¸،éژ–م‚’هˆ‡و–مپ™م‚‹مپŒم€پو±è¥؟هˆ‡و–مپ¯DNAمپ®ç‰‡éژ–م‚’مپمپ®مپ¾مپ¾و®‹مپ™م€‚組وڈ›مپˆمپ®éڑ›مپ«مƒ›مƒھمƒ‡م‚¤م‚¸مƒ£مƒ³م‚¯م‚·مƒ§مƒ³مپŒه½¢وˆگمپ•م‚Œم‚‹مپ“مپ¨مپ§م€پéپ؛ن¼çڑ„ه¤ڑو§کو€§م€پوں“色ن½“ن¸ٹمپ§مپ®éپ؛ن¼هگمپ®ن؛¤وڈ›م€پمپٹم‚ˆمپ³é‡ژç”ںه‹م‚¦م‚¤مƒ«م‚¹م‚²مƒژمƒ مپ®ç™؛çڈ¾مپŒهڈ¯èƒ½مپ«مپھم‚‹م€‚ 進هŒ–→詳細مپ¯م€ŒRNAمƒ¯مƒ¼مƒ«مƒ‰ن»®èھ¬م€چم‚’هڈ‚ç…§
DNAمپ«مپ¯مپ‚م‚‰م‚†م‚‹ç”ںه‘½ن½“مپŒو©ں能مپ—م€پوˆگé•·مپ—م€پç”ںو®–مپ™م‚‹مپںم‚پمپ®éپ؛ن¼وƒ…ه ±مپŒهگ«مپ¾م‚Œمپ¦مپ„م‚‹م€‚مپ—مپ‹مپ—40ه„„ه¹´مپ®ç”ںه‘½مپ®و´هڈ²مپ®ن¸مپ§م€پDNAمپŒمپ„مپ¤مپ‹م‚‰مپ“مپ®و©ں能م‚’وœمپںمپ—مپ¦مپچمپںمپ‹مپ¯ن¸چوکژمپ§مپ‚م‚‹م€‚وœ€م‚‚هˆوœںمپ®ç”ںه‘½ن½“مپ¯RNAم‚’éپ؛ن¼ç‰©è³ھمپ¨مپ—مپ¦ن½؟مپ£مپ¦مپ„مپںمپ®مپ§مپ¯مپھمپ„مپ‹مپ¨مپ„مپ†وڈگو،ˆم‚‚مپ‚م‚‹[145][146]م€‚RNAمپ¯éپ؛ن¼وƒ…ه ±مپ®ن¼éپ”مپ¨مƒھمƒœم‚¶م‚¤مƒ مپ®ن¸€éƒ¨مپ¨مپ—مپ¦مپ®è§¦هھ’ن½œç”¨مپ®ن¸،و–¹م‚’è،Œمپ†مپ“مپ¨مپŒمپ§مپچم‚‹مپںم‚پم€پهˆوœںمپ®ç´°èƒن»£è¬مپ«مپٹمپ„مپ¦ن¸ه؟ƒçڑ„مپھه½¹ه‰²م‚’وœمپںمپ—مپ¦مپ„مپںهڈ¯èƒ½و€§مپŒمپ‚م‚‹[147]م€‚و ¸é…¸مپŒè§¦هھ’ن½œç”¨مپ¨éپ؛ن¼ه¦مپ®ن¸،و–¹مپ«ن½؟م‚ڈم‚Œمپ¦مپ„مپںمپ¨مپ™م‚‹م€پمپ“مپ®هڈ¤ن»£مپ®RNAمƒ¯مƒ¼مƒ«مƒ‰مپ¯م€پ4ه،©هں؛مپ«هں؛مپ¥مپڈçڈ¾هœ¨مپ®éپ؛ن¼وڑ—هڈ·مپ®é€²هŒ–مپ«ه½±éں؟م‚’ن¸ژمپˆمپںمپ‹م‚‚مپ—م‚Œمپھمپ„م€‚مپ“مپ®م‚ˆمپ†مپھç”ں物مپ«مپٹمپ‘م‚‹ç•°مپھم‚‹ه،©هں؛مپ®و•°مپ¯م€په°‘مپھمپ„ه،©هں؛و•°مپ«م‚ˆم‚‹è¤‡è£½ç²¾ه؛¦مپ®هگ‘ن¸ٹمپ¨م€په¤ڑو•°مپ®ه،©هں؛مپ«م‚ˆم‚‹مƒھمƒœم‚¶م‚¤مƒ مپ®è§¦هھ’هٹ¹çژ‡مپ®هگ‘ن¸ٹمپ¨مپ®é‡£م‚ٹهگˆمپ„é–¢ن؟‚مپ«م‚ˆمپ£مپ¦مپچمپ¾مپ£مپںهڈ¯èƒ½و€§م‚‚مپ‚م‚‹[148]م€‚مپ—مپ‹مپ—DNAمپ¯ç’°ه¢ƒن¸مپ§100ن¸‡ه¹´وœھو؛€مپ—مپ‹هکهœ¨مپ§مپچمپڑم€پو؛¶و¶²ن¸مپ§م‚†مپ£مپڈم‚ٹمپ¨çںمپ„و–片مپ«هˆ†è§£مپ•م‚Œم‚‹مپںم‚پم€پمپ»مپ¨م‚“مپ©مپ®هŒ–çں³مپ‹م‚‰DNAم‚’ه›هڈژمپ™م‚‹مپ“مپ¨مپ¯ن¸چهڈ¯èƒ½مپ§م€پهڈ¤ن»£مپ®éپ؛ن¼هگç³»مپ®ç›´وژ¥çڑ„مپھ証و‹ مپ¯مپھمپ„[149]م€‚م‚ˆم‚ٹهڈ¤مپ„DNAمپŒهکهœ¨مپ™م‚‹مپ¨مپ„مپ†ن¸»ه¼µم‚‚مپھمپ•م‚Œمپ¦مپٹم‚ٹم€پ特مپ«2ه„„5هچƒن¸‡ه¹´ه‰چمپ®ه،©مپ®çµگو™¶مپ‹م‚‰ç”ںهکهڈ¯èƒ½مپھç´°èڈŒمپŒهˆ†é›¢مپ•م‚Œمپںمپ¨مپ„مپ†ه ±ه‘ٹمپŒمپ‚م‚‹مپŒ[150]م€پمپ“م‚Œم‚‰مپ®ن¸»ه¼µمپ«مپ¯è³›هگ¦مپŒمپ‚م‚‹[151][152]م€‚ DNAمپ®و§‹وˆگè¦پç´ ï¼ˆم‚¢مƒ‡مƒ‹مƒ³م€پم‚°م‚¢مƒ‹مƒ³م€پمپٹم‚ˆمپ³é–¢é€£مپ™م‚‹وœ‰و©ںهˆ†هگ)مپ¯م€پهœ°çگƒه¤–مپ®ه®‡ه®™ç©؛é–“مپ§ه½¢وˆگمپ•م‚Œمپںهڈ¯èƒ½و€§م‚‚مپ‚م‚‹[153][154][155]م€‚م‚¦مƒ©م‚·مƒ«م€پم‚·مƒˆم‚·مƒ³م€پمƒپمƒںمƒ³م‚’هگ«م‚€م€پç”ںه‘½مپ®è¤‡é›‘مپھDNAم‚„RNAمپ®وœ‰و©ںهŒ–هگˆç‰©م‚‚مپ¾مپںم€پéڑ•çں³مپ‹م‚‰ç™؛見مپ•م‚Œمپںمƒ”مƒھمƒںم‚¸مƒ³مپ®م‚ˆمپ†مپھهŒ–ه¦ç‰©è³ھم‚’ه‡؛ç™؛点مپ¨مپ—مپ¦م€په®‡ه®™ç©؛é–“مپ®و¨،ه€£مپ—مپںو،ن»¶ن¸‹مپ®ه®ں験ه®¤مپ§هگˆوˆگمپ•م‚Œمپ¦مپ„م‚‹م€‚مƒ”مƒھمƒںم‚¸مƒ³مپ¯م€په®‡ه®™مپ§ç™؛見مپ•م‚Œمپںوœ€م‚‚ç‚ç´ م‚’ه¤ڑمپڈهگ«م‚€هŒ–ه¦ç‰©è³ھمپ§مپ‚م‚‹ه¤ڑç’°èٹ³é¦™و—ڈç‚هŒ–و°´ç´ (PAH)مپ¨هگŒو§کم€پ赤色ه·¨وکںم‚„وکںé–“ه®‡ه®™ه،µم‚„م‚¬م‚¹é›²مپ§ه½¢وˆگمپ•م‚Œمپںهڈ¯èƒ½و€§مپŒمپ‚م‚‹[156]م€‚ 2021ه¹´2وœˆم€پ科ه¦è€…مپںمپ،مپ¯هˆم‚پمپ¦م€پ100ن¸‡ه¹´ن»¥ن¸ٹه‰چمپ®مƒمƒ³مƒ¢م‚¹è±،مپ®éپ؛ن½“مپ‹م‚‰DNAé…چهˆ—م‚’و±؛ه®ڑمپ—مپںمپ“مپ¨م‚’ه ±ه‘ٹمپ—مپںم€‚مپ“م‚Œمپ¾مپ§مپ«ه،©هں؛é…چهˆ—مپŒو±؛ه®ڑمپ•م‚Œمپںوœ€هڈ¤مپ®DNAمپ§مپ‚م‚‹[157][158]م€‚ وٹ€è،“مپ«مپٹمپ‘م‚‹ç”¨é€”éپ؛ن¼هگه·¥ه¦مƒ•م‚§مƒژمƒ¼مƒ«مƒ»م‚¯مƒمƒمƒ›مƒ«مƒ وٹ½ه‡؛و³•مپ®م‚ˆمپ†مپ«م€پç”ں物مپ‹م‚‰DNAم‚’精製مپ™م‚‹و–¹و³•م‚„م€پهˆ¶é™گو¶ˆهŒ–م‚„مƒمƒھمƒ،مƒ©مƒ¼م‚¼é€£éژ–هڈچه؟œمپ®م‚ˆمپ†مپ«ه®ں験ه®¤مپ§DNAم‚’و“چن½œمپ™م‚‹و–¹و³•مپŒé–‹ç™؛مپ•م‚Œمپںم€‚çڈ¾ن»£مپ®ç”ں物ه¦م‚„ç”ںهŒ–ه¦مپ§مپ¯م€پ組وڈ›مپˆDNAمپ®هˆ†é‡ژمپ§مپ“م‚Œم‚‰مپ®وٹ€è،“م‚’و´»ç”¨مپ—مپ¦مپ„م‚‹م€‚組وڈ›مپˆDNAمپ¨مپ¯م€پن»–مپ®DNAé…چهˆ—مپ‹م‚‰çµ„مپ؟ç«‹مپ¦م‚‰م‚Œمپںن؛؛ه·¥مپ®DNAé…چهˆ—مپ§مپ‚م‚‹م€‚مپ“م‚Œم‚‰مپ¯م‚¦م‚¤مƒ«م‚¹مƒ™م‚¯م‚؟مƒ¼م‚’هˆ©ç”¨مپ—مپ¦م€پمƒ—مƒ©م‚¹مƒںمƒ‰مپ‚م‚‹مپ„مپ¯ن»–مپ®éپ©هˆ‡مپھه‹ه¼ڈمپ§م€پç”ں物مپ«ه½¢è³ھ転وڈ›مپ™م‚‹مپ“مپ¨مپŒمپ§مپچم‚‹[159]م€‚ç”ں産مپ•م‚Œمپںéپ؛ن¼هگ組وڈ›مپˆç”ں物مپ¯م€پ組وڈ›مپˆم‚؟مƒ³مƒ‘م‚¯è³ھمپ®م‚ˆمپ†مپھ製ه“پم‚’è£½é€ مپ—مپںم‚ٹم€پهŒ»ه¦ç ”究مپ§ن½؟用مپ—مپںم‚ٹ[160]م€پè¾²و¥مپ§ç¹پو®–مپ—مپںم‚ٹمپ™م‚‹[161][162]م€‚ DNAمƒ—مƒمƒ•م‚،م‚¤مƒھمƒ³م‚°â†’詳細مپ¯م€ŒDNAمƒ—مƒمƒ•م‚،م‚¤مƒھمƒ³م‚°م€چم‚’هڈ‚ç…§
و³•ç§‘ه¦è€…مپ¯م€پçٹ¯ç½ھçڈ¾ه ´مپ§ç™؛見مپ•م‚Œمپںè،€و¶²م€پç²¾و¶²م€پçڑ®è†ڑم€په”¾و¶²م€پمپ¾مپںمپ¯و¯›é«ھمپ«هگ«مپ¾م‚Œم‚‹DNAم‚’هˆ©ç”¨مپ—مپ¦م€پهٹ ه®³è€…مپھمپ©مپ®ه€‹ن؛؛مپ¨ن¸€è‡´مپ™م‚‹DNAم‚’特ه®ڑمپ™م‚‹مپ“مپ¨مپŒمپ§مپچم‚‹[163]م€‚مپ“مپ®و‰‹و³•مپ¯و£ه¼ڈمپ«مپ¯DNAمƒ—مƒمƒ•م‚،م‚¤مƒھمƒ³م‚°ï¼ˆè‹±: DNA profiling)مپ¨ه‘¼مپ°م‚Œم€پDNAوŒ‡ç´‹و³•ï¼ˆè‹±: DNA fingerprinting)مپ¨م‚‚ه‘¼مپ°م‚Œم‚‹م€‚DNAمƒ—مƒمƒ•م‚،م‚¤مƒھمƒ³م‚°مپ§مپ¯م€پم‚·مƒ§مƒ¼مƒˆم‚؟مƒ³مƒ‡مƒ مƒھمƒ”مƒ¼مƒˆï¼ˆç¸¦هˆ—ه‹هڈچه¾©é…چهˆ—)م‚„مƒںمƒ‹م‚µمƒ†مƒ©م‚¤مƒˆمپھمپ©م€پهڈچه¾©DNAمپ®هڈ¯ه¤‰éƒ¨هˆ†مپ®é•·مپ•م‚’ه€‹ن؛؛é–“مپ§و¯”較مپ™م‚‹م€‚مپ“مپ®و–¹و³•مپ¯é€ڑه¸¸م€پن¸€è‡´مپ™م‚‹DNAم‚’هگŒه®ڑمپ™م‚‹مپںم‚پمپ®éه¸¸مپ«ن؟،é ¼و€§مپ®é«کمپ„وٹ€è،“مپ§مپ‚م‚‹[164]م€‚مپںمپ مپ—م€پçڈ¾ه ´مپŒè¤‡و•°هگچمپ®DNAمپ§و±ڑوں“مپ•م‚Œمپ¦مپ„م‚‹ه ´هگˆم€پهگŒه®ڑمپŒè¤‡é›‘مپ«مپھم‚‹مپ“مپ¨مپŒمپ‚م‚‹[165]م€‚DNAمƒ—مƒمƒ•م‚،م‚¤مƒھمƒ³م‚°مپ¯1984ه¹´مپ«م‚¤م‚®مƒھم‚¹مپ®éپ؛ن¼ه¦è€…م‚¢مƒ¬مƒƒم‚¯مƒ»م‚¸م‚§مƒ•مƒھمƒ¼م‚؛مپ«م‚ˆمپ£مپ¦é–‹ç™؛مپ•م‚Œ[166]م€پ1988ه¹´مپ®م‚¨مƒ³مƒ€مƒ¼مƒ“مƒ¼و®؛ن؛؛ن؛‹ن»¶مپ§م‚³مƒھمƒ³مƒ»مƒ”مƒƒمƒپمƒ•م‚©مƒ¼م‚¯م‚’وœ‰ç½ھمپ«مپ™م‚‹مپںم‚پمپ«و³•ç§‘ه¦مپ§هˆم‚پمپ¦ن½؟用مپ•م‚Œمپں[167]م€‚ و³•ç§‘ه¦مپŒç™؛éپ”مپ—م€پè،€و¶²م€پçڑ®è†ڑم€په”¾و¶²م€پو¯›é«ھمپھمپ©مپ®ه¾®é‡ڈم‚µمƒ³مƒ—مƒ«مپ§éپ؛ن¼هگç…§هگˆمپŒمپ§مپچم‚‹م‚ˆمپ†مپ«مپھمپ£مپںمپ“مپ¨مپ§م€په¤ڑمپڈمپ®ن؛‹ن»¶مپŒه†چèھ؟وں»مپ•م‚Œم‚‹م‚ˆمپ†مپ«مپھمپ£مپںم€‚ه½“هˆمپ®èھ؟وں»و™‚مپ«مپ¯ç§‘ه¦çڑ„مپ«ن¸چهڈ¯èƒ½مپ§مپ‚مپ£مپں証و‹ م‚‚م€پçڈ¾هœ¨مپ§مپ¯ç™؛見مپ•م‚Œم‚‹مپ“مپ¨مپŒمپ‚م‚‹م€‚ن¸€éƒ¨مپ®هœ°هںںمپ«مپٹمپ„مپ¦ن؛Œé‡چمپ®هچ±é™؛مپ®هژںه‰‡ï¼ˆè‹±: double jeopardy law)مپŒو’¤ه»ƒمپ•م‚Œمپںمپ“مپ¨م‚‚مپ‚مپ„مپ¾مپ£مپ¦م€پمپ“م‚Œمپ¾مپ§مپ®è£پهˆ¤مپ§é™ھه¯©م‚’ç´چه¾—مپ•مپ›م‚‹مپ«هچپهˆ†مپھ証و‹ مپŒه¾—م‚‰م‚Œمپھمپ‹مپ£مپںن؛‹ن»¶مپ§م‚‚ه†چه¯©مپŒهڈ¯èƒ½مپ«مپھم‚‹مپ“مپ¨مپŒمپ‚م‚‹م€‚é‡چه¤§çٹ¯ç½ھمپ§èµ·è¨´مپ•م‚Œمپںن؛؛م€…مپ¯م€پç…§هگˆç›®çڑ„مپ§DNAم‚µمƒ³مƒ—مƒ«مپ®وڈگه‡؛م‚’و±‚م‚پم‚‰م‚Œم‚‹مپ“مپ¨مپŒمپ‚م‚‹م€‚و³•ç§‘ه¦çڑ„مپ«ه¾—م‚‰م‚ŒمپںDNAç…§هگˆمپ«ه¯¾مپ™م‚‹وœ€م‚‚وکژ白مپھوٹ—ه¼پمپ¯م€پ証و‹ مپ®ç›¸ن؛’و±ڑوں“مپŒèµ·مپ“مپ£مپںمپ¨ن¸»ه¼µمپ™م‚‹مپ“مپ¨مپ§مپ‚م‚‹م€‚مپ“مپ®مپںم‚پم€پé‡چه¤§çٹ¯ç½ھمپ®و–°ن؛‹ن¾‹مپ«ه¯¾مپ—م€پç´°ه؟ƒمپ®و³¨و„ڈم‚’و‰•مپ£مپںهژ³و ¼مپھهڈ–م‚ٹو‰±مپ„و‰‹é †مپŒه°ژه…¥مپ•م‚Œم‚‹م‚ˆمپ†مپ«مپھمپ£مپںم€‚ DNAمƒ—مƒمƒ•م‚،م‚¤مƒھمƒ³م‚°مپ¯مپ¾مپںم€پ集ه›£و»ه‚·ن؛‹ن»¶مپ®çٹ 牲者[168]م€پé‡چه¤§ن؛‹و•…مپ®éپ؛ن½“م‚„مپمپ®ن¸€éƒ¨م€پ集ه›£وˆ¦و²،者ه¢“هœ°مپ«مپٹمپ‘م‚‹çٹ 牲者ه€‹ن؛؛مپ®è؛«ه…ƒم‚’م€په®¶و—ڈمپ¨مپ®ç…§هگˆمپ«م‚ˆمپ£مپ¦ç¢؛èھچمپ™م‚‹مپںم‚پمپ«م‚‚ن½؟用مپ•م‚Œم€پوˆگهٹںم‚’هڈژم‚پمپ¦مپ„م‚‹م€‚ DNAمƒ—مƒمƒ•م‚،م‚¤مƒھمƒ³م‚°مپ¯م€پèھ°مپ‹مپŒهگن¾›مپ®ç”ںمپ؟مپ®è¦ھمپ¾مپںمپ¯ç¥–父و¯چمپ§مپ‚م‚‹مپ‹مپ©مپ†مپ‹م‚’هˆ¤ه®ڑمپ™م‚‹مپںم‚پمپ®DNAè¦ھهگé‘‘ه®ڑمپ«م‚‚ن½؟用مپ•م‚Œم€پè¦ھمپ¨مپ•م‚Œم‚‹ن؛؛物مپŒهگن¾›مپ¨ç”ں物ه¦çڑ„مپ«è،€ç¸پé–¢ن؟‚مپŒمپ‚م‚‹ه ´هگˆم€پè¦ھمپ§مپ‚م‚‹ç¢؛çژ‡مپ¯é€ڑه¸¸99.99%مپ§مپ‚م‚‹م€‚é€ڑه¸¸مپ®DNAé…چهˆ—و±؛ه®ڑو³•مپ¯ه‡؛ç”ںه¾Œمپ«è،Œم‚ڈم‚Œم‚‹مپŒم€پو¯چè¦ھمپŒمپ¾مپ ه¦ٹه¨ مپ—مپ¦مپ„م‚‹é–“مپ«è¦ھهگé–¢ن؟‚م‚’و¤œوں»مپ™م‚‹و–°مپ—مپ„و–¹و³•مپŒمپ‚م‚‹[169]م€‚ DNAé…µç´ مپ¾مپںمپ¯è§¦هھ’DNA→詳細مپ¯م€Œمƒ‡م‚ھم‚م‚·مƒھمƒœم‚¶م‚¤مƒ م€چم‚’هڈ‚ç…§
مƒ‡م‚ھم‚م‚·مƒھمƒœم‚¶م‚¤مƒ (英: deoxyribozyme)مپ¯م€پDNAé…µç´ ï¼ˆDNAzymes)مپ¾مپںمپ¯è§¦هھ’DNA(catalytic DNA)مپ¨م‚‚ه‘¼مپ°م‚Œم€پ1994ه¹´مپ«هˆم‚پمپ¦ç™؛見مپ•م‚Œمپں[170]م€‚مپ“م‚Œم‚‰مپ®ه¤§éƒ¨هˆ†مپ¯م€پin vitroéپ¸وٹو³•مپ¾مپںمپ¯è©¦é¨“ç®،ه†…進هŒ–و³•ï¼ˆè‹±: Systematic Evolution of Ligands by Exponential Enrichmentم€پSELEX)مپ¨ه‘¼مپ°م‚Œم‚‹çµ„مپ؟هگˆم‚ڈمپ›م‚¢مƒ—مƒمƒ¼مƒپم‚’ن½؟用مپ—مپ¦م€پمƒ©مƒ³مƒ€مƒ مپھDNAé…چهˆ—مپ®ه¤§è¦ڈو¨،مƒ—مƒ¼مƒ«مپ‹م‚‰هچک離مپ•م‚Œمپںن¸€وœ¬éژ–DNAé…چهˆ—مپ§مپ‚م‚‹م€‚DNAé…µç´ مپ¯م€پRNA-DNAهˆ‡و–م€پRNA-DNAمƒ©م‚¤م‚²مƒ¼م‚·مƒ§مƒ³م€پم‚¢مƒںمƒژé…¸مپ®مƒھمƒ³é…¸هŒ–-脱مƒھمƒ³é…¸هŒ–م€پç‚ç´ -ç‚ç´ çµگهگˆه½¢وˆگمپھمپ©م€پمپ•مپ¾مپ–مپ¾مپھهŒ–ه¦هڈچه؟œم‚’触هھ’مپ™م‚‹م€‚DNAé…µç´ مپ¯م€پ触هھ’هڈچه؟œمپ®هŒ–ه¦هڈچه؟œé€ںه؛¦م‚’م€پç„،触هھ’هڈچه؟œمپ®وœ€ه¤§1هچƒه„„ه€چمپ«هگ‘ن¸ٹمپ•مپ›م‚‹مپ“مپ¨مپŒمپ§مپچم‚‹[171]م€‚DNAé…µç´ مپ®ن¸مپ§م‚‚مپ£مپ¨م‚‚ه؛ƒمپڈç ”ç©¶مپ•م‚Œمپ¦مپ„م‚‹مپ®مپ¯RNAهˆ‡و–ه‹مپ§م€پمپ•مپ¾مپ–مپ¾مپھ金ه±م‚¤م‚ھمƒ³مپ®و¤œه‡؛م‚„و²»ç™‚è–¬مپ®è¨è¨ˆمپ«ن½؟用مپ•م‚Œمپ¦مپ„م‚‹م€‚GR-5 DNAé…µç´ ï¼ˆé‰›ç‰¹ç•°çڑ„)[170]م€پCA1-3 DNAé…µç´ ï¼ˆéٹ…特異çڑ„)[172]م€پ39E DNAé…µç´ ï¼ˆم‚¦مƒ©مƒ‹مƒ«ç‰¹ç•°çڑ„)م€پNaA43 DNAé…µç´ ï¼ˆمƒٹمƒˆمƒھم‚¦مƒ 特異çڑ„)[173]مپھمپ©م€پمپ„مپڈمپ¤مپ‹مپ®é‡‘ه±ç‰¹ç•°çڑ„DNAé…µç´ مپŒه ±ه‘ٹمپ•م‚Œمپ¦مپ„م‚‹م€‚NaA43 DNAé…µç´ مپ¯م€پمƒٹمƒˆمƒھم‚¦مƒ مپ«ه¯¾مپ—مپ¦ن»–مپ®é‡‘ه±م‚¤م‚ھمƒ³م‚ˆم‚ٹم‚‚10,000ه€چن»¥ن¸ٹéپ¸وٹçڑ„مپ§مپ‚م‚‹مپ¨ه ±ه‘ٹمپ•م‚Œمپ¦مپٹم‚ٹم€پç´°èƒه†…مپ§مƒھم‚¢مƒ«م‚؟م‚¤مƒ مپ®مƒٹمƒˆمƒھم‚¦مƒ م‚»مƒ³م‚µمƒ¼م‚’ن½œوˆگمپ™م‚‹مپںم‚پمپ«ن½؟用مپ•م‚Œمپںم€‚ مƒگم‚¤م‚ھم‚¤مƒ³مƒ•م‚©مƒمƒ†م‚£م‚¯م‚¹â†’詳細مپ¯م€Œمƒگم‚¤م‚ھم‚¤مƒ³مƒ•م‚©مƒمƒ†م‚£م‚¯م‚¹م€چم‚’هڈ‚ç…§
مƒگم‚¤م‚ھم‚¤مƒ³مƒ•م‚©مƒمƒ†م‚£م‚¯م‚¹مپ¯م€پDNAو ¸é…¸é…چهˆ—مƒ‡مƒ¼م‚؟م‚’هگ«م‚€ç”ں物ه¦çڑ„مƒ‡مƒ¼م‚؟مپ®ن؟هکم€پمƒ‡مƒ¼م‚؟مƒم‚¤مƒ‹مƒ³م‚°م€پو¤œç´¢م€پو“چن½œمپ®مپںم‚پمپ®وٹ€è،“é–‹ç™؛م‚’هگ«م‚€ه¦ه•ڈهˆ†é‡ژمپ§مپ‚م‚‹م€‚مپ“م‚Œم‚‰مپ®وٹ€è،“مپ¯م€پم‚³مƒ³مƒ”مƒ¥مƒ¼م‚؟م‚µم‚¤م‚¨مƒ³م‚¹م€پ特مپ«و–‡ه—هˆ—و¤œç´¢م‚¢مƒ«م‚´مƒھم‚؛مƒ م€پو©ںو¢°ه¦ç؟’م€پمƒ‡مƒ¼م‚؟مƒ™مƒ¼م‚¹çگ†è«–مپ«ه؛ƒمپڈه؟œç”¨مپ•م‚Œم‚‹م‚ˆمپ†مپ«مپھمپ£مپں[174]م€‚و–‡ه—هˆ—و¤œç´¢مپ¾مپںمپ¯مƒمƒƒمƒپمƒ³م‚°م‚¢مƒ«م‚´مƒھم‚؛مƒ مپ¯م€پم‚ˆم‚ٹه¤§مپچمپھو–‡ه—هˆ—مپ®ن¸مپ«مپ‚م‚‹و–‡ه—هˆ—مپ®ه‡؛çڈ¾م‚’و¤œه‡؛مپ™م‚‹و‰‹و³•مپ§م€پمƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰مپ®ç‰¹ç•°çڑ„é…چهˆ—م‚’و¤œç´¢مپ™م‚‹مپںم‚پمپ«é–‹ç™؛مپ•م‚Œمپں[175]م€‚DNAé…چهˆ—م‚’ن»–مپ®DNAé…چهˆ—مپ¨و•´هˆ—مپ•مپ›م‚‹مپ“مپ¨مپ§م€پ相هگŒé…چهˆ—م‚’هگŒه®ڑمپ—م€پمپم‚Œم‚‰م‚’هŒ؛هˆ¥مپ™م‚‹ç‰¹ç•°çڑ„ه¤‰ç•°م‚’çھپمپچو¢م‚پم‚‹مپ“مپ¨مپŒمپ§مپچم‚‹م€‚مپ“م‚Œم‚‰مپ®وٹ€è،“م€پ特مپ«ه¤ڑé‡چé…چهˆ—م‚¢مƒ©م‚¤مƒ³مƒ،مƒ³مƒˆمپ¯م€پ系統çڑ„é–¢ن؟‚م‚„م‚؟مƒ³مƒ‘م‚¯è³ھو©ں能م‚’ç ”ç©¶مپ™م‚‹éڑ›مپ«ن½؟用مپ•م‚Œم‚‹[176]م€‚مƒ’مƒˆم‚²مƒژمƒ مƒ—مƒم‚¸م‚§م‚¯مƒˆمپ§ن½œوˆگمپ•م‚Œمپںم‚ˆمپ†مپھه…¨م‚²مƒژمƒ DNAé…چهˆ—مپ®ه¤§è¦ڈو¨،مپھمƒ‡مƒ¼م‚؟م‚»مƒƒمƒˆمپ¯م€پهگ„وں“色ن½“ن¸ٹمپ®éپ؛ن¼هگم‚„èھ؟節م‚¨مƒ¬مƒ،مƒ³مƒˆمپ®ن½چç½®م‚’特ه®ڑمپ™م‚‹م‚¢مƒژمƒ†مƒ¼م‚·مƒ§مƒ³مپŒمپھمپڈمپ¦مپ¯هˆ©ç”¨مپŒه›°é›£مپ§مپ‚م‚‹م€‚م‚؟مƒ³مƒ‘م‚¯è³ھم‚„RNAم‚’م‚³مƒ¼مƒ‰مپ™م‚‹éپ؛ن¼هگمپ«é–¢é€£مپ™م‚‹ç‰¹ه¾´çڑ„مپھمƒ‘م‚؟مƒ¼مƒ³م‚’وŒپمپ¤DNAé…چهˆ—é کهںںمپ¯م€پéپ؛ن¼هگوژ¢ç´¢م‚¢مƒ«م‚´مƒھم‚؛مƒ مپ«م‚ˆمپ£مپ¦هگŒه®ڑمپ™م‚‹مپ“مپ¨مپŒمپ§مپچم€پمپ“م‚Œمپ«م‚ˆم‚ٹç ”ç©¶è€…مپ¯م€پ特ه®ڑمپ®éپ؛ن¼هگ産物مپŒه®ں験çڑ„مپ«هچک離مپ•م‚Œم‚‹ه‰چمپ§مپ‚مپ£مپ¦م‚‚م€پç”ں物ه†…مپ§مپ®هکهœ¨مپ¨هڈ¯èƒ½و€§مپ®مپ‚م‚‹و©ں能م‚’ن؛ˆو¸¬مپ™م‚‹مپ“مپ¨مپŒمپ§مپچم‚‹[177]م€‚مپ¾مپںم€پم‚²مƒژمƒ ه…¨ن½“م‚’و¯”較مپ™م‚‹مپ“مپ¨مپ§م€پç”ں物مپ®é€²هŒ–مپ®و´هڈ²مپ«ç„¦ç‚¹م‚’ه½“مپ¦مپںم‚ٹم€پ複雑مپھ進هŒ–مپ®éپژ程م‚’ç ”ç©¶مپ™م‚‹مپ“مپ¨م‚‚مپ§مپچم‚‹م€‚ DNAمƒٹمƒژمƒ†م‚¯مƒژمƒم‚¸مƒ¼ →詳細مپ¯م€ŒDNAمƒٹمƒژمƒ†م‚¯مƒژمƒم‚¸مƒ¼م€چم‚’هڈ‚ç…§
DNAمƒٹمƒژمƒ†م‚¯مƒژمƒم‚¸مƒ¼مپ¯م€پDNAم‚„ن»–مپ®و ¸é…¸مپ«ç‰¹وœ‰مپ®هˆ†هگèھچèک特و€§م‚’هˆ©ç”¨مپ—مپ¦م€پوœ‰ç”¨مپھ特و€§م‚’ه‚™مپˆمپںè‡ھه·±é›†هگˆهŒ–能مƒ»هˆ†ه²گDNA複هگˆن½“م‚’ن½œم‚ٹه‡؛مپ™وٹ€è،“é کهںںمپ§مپ‚م‚‹[179]م€‚DNAمپ¯ç”ں物ه¦çڑ„وƒ…ه ±مپ®ن¼éپ”و‰‹و®µمپ¨مپ—مپ¦مپ§مپ¯مپھمپڈم€پو§‹é€ وگو–™مپ¨مپ—مپ¦ن½؟用مپ™م‚‹مپ“مپ¨م‚‚مپ§مپچم‚‹م€‚مپمپ®çµگوœم€پ2و¬،ه…ƒه‘¨وœںو ¼هگ(م‚؟م‚¤مƒ«مƒ™مƒ¼م‚¹مپ¨DNAم‚ھمƒھم‚¬مƒںو³•مپ®ن¸،و–¹ï¼‰م‚„م€په¤ڑé¢ن½“ه½¢çٹ¶م‚’وŒپمپ¤3و¬،ه…ƒو§‹é€ مپ®ه‰µé€ مپ«مپ¤مپھمپŒمپ£مپں[180]م€‚مƒٹمƒژمƒ،م‚«مƒ‹م‚«مƒ«مƒ‡مƒگم‚¤م‚¹م‚„م‚¢مƒ«م‚´مƒھم‚؛مƒ çڑ„è‡ھه·±é›†هگˆهŒ–م‚‚ه®ں証مپ•م‚Œمپ¦مپٹم‚ٹ[181]م€پمپ“م‚Œم‚‰مپ®DNAو§‹é€ مپ¯م€پ金مƒٹمƒژç²’هگم‚„م‚¹مƒˆمƒ¬مƒ—مƒˆم‚¢مƒ“م‚¸مƒ³م‚؟مƒ³مƒ‘م‚¯è³ھمپھمپ©م€پن»–مپ®هˆ†هگ集هگˆن½“مپ®é‹³ه‹مپ¨مپ™م‚‹مپںم‚پمپ«ن½؟用مپ•م‚Œمپ¦مپ„م‚‹[182]م€‚DNAم‚„ن»–مپ®و ¸é…¸مپ¯م€پم‚¢مƒ—م‚؟مƒمƒ¼ï¼ˆمپ•مپ¾مپ–مپ¾مپھمƒگم‚¤م‚ھمƒ†م‚¯مƒژمƒم‚¸مƒ¼م‚„ç”ں物هŒ»ه¦مپ®ç”¨é€”مپ«ن½؟م‚ڈم‚Œم‚‹م€پ特ه®ڑمپ®و¨™çڑ„هˆ†هگمپ«ه¯¾مپ™م‚‹هگˆوˆگم‚ھمƒھم‚´مƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰مƒھم‚¬مƒ³مƒ‰ï¼‰مپ®هں؛ç¤ژمپ¨مپھمپ£مپ¦مپ„م‚‹[183]م€‚ 系統ه¦مپ¨ن؛؛é،ه¦â†’詳細مپ¯م€Œç³»çµ±ه¦م€چمپٹم‚ˆمپ³م€Œéپ؛ن¼هگç³»ه›³م€چم‚’هڈ‚ç…§
DNAمپ¯و™‚é–“مپ®çµŒéپژمپ¨مپ¨م‚‚مپ«ه¤‰ç•°م‚’è“„ç©چمپ—م€پéپ؛ن¼مپ«م‚ˆمپ£مپ¦و´هڈ²çڑ„مپھوƒ…ه ±م‚’هگ«م‚“مپ§مپٹم‚ٹم€پDNAمپ®ه،©هں؛é…چهˆ—م‚’و¯”較مپ™م‚‹مپ“مپ¨مپ§م€پéپ؛ن¼ه¦è€…مپ¯ç”ں物مپ®é€²هŒ–مپ®و´هڈ²م€پ系統ç™؛ç”ںم‚’وژ¨ه®ڑمپ™م‚‹مپ“مپ¨مپŒمپ§مپچم‚‹[184]م€‚系統ç™؛ç”ںه¦مپ¯é€²هŒ–ç”ں物ه¦مپ«مپٹمپ‘م‚‹ه¼·هٹ›مپھéپ“ه…·مپ§مپ‚م‚‹م€‚ç”ں物種ه†…مپ®DNAé…چهˆ—م‚’و¯”較مپ™م‚‹مپ“مپ¨مپ§م€پ集ه›£éپ؛ن¼ه¦è€…مپ¯ç‰¹ه®ڑمپ®é›†ه›£مپ®و´هڈ²م‚’çں¥م‚‹مپ“مپ¨مپŒمپ§مپچم‚‹م€‚مپ“م‚Œمپ¯م€پç”ںو…‹éپ؛ن¼ه¦مپ‹م‚‰ن؛؛é،ه¦مپ«è‡³م‚‹مپ¾مپ§م€پمپ•مپ¾مپ–مپ¾مپھç ”ç©¶مپ«هˆ©ç”¨مپ§مپچم‚‹م€‚ وƒ…ه ±م‚¹مƒˆمƒ¬مƒ¼م‚¸â†’詳細مپ¯م€ŒDNAمƒ‡م‚¸م‚؟مƒ«مƒ‡مƒ¼م‚؟م‚¹مƒˆمƒ¬مƒ¼م‚¸م€چم‚’هڈ‚ç…§
وƒ…ه ±è¨ک録هھ’ن½“مپ¨مپ—مپ¦مپ®DNAمپ¯م€پé›»هگو©ںه™¨مپ«و¯”مپ¹مپ¦è¨ک録ه¯†ه؛¦مپŒمپ¯م‚‹مپ‹مپ«é«کمپ„مپںم‚پم€پéه¸¸مپ«ه¤§مپچمپھهڈ¯èƒ½و€§م‚’ç§کم‚پمپ¦مپ„م‚‹م€‚مپ—مپ‹مپ—م‚³م‚¹مƒˆمپŒé«کمپڈم€پèھمپ؟و›¸مپچمپ«و™‚é–“مپŒمپ‹مپ‹م‚ٹ(مƒ،مƒ¢مƒھمƒ¬م‚¤مƒ†مƒ³م‚·ï¼‰م€پن؟،é ¼و€§مپŒهچپهˆ†مپ§مپھمپ„مپ“مپ¨مپھمپ©مپ‹م‚‰م€په®ں用هŒ–مپ«مپ¯è‡³مپ£مپ¦مپ„مپھمپ„[185][186]م€‚ و´هڈ²
→詳細مپ¯م€Œهˆ†هگç”ں物ه¦مپ®و´هڈ²م€چم‚’هڈ‚ç…§
  DNAمپŒوœ€هˆمپ«هچک離مپ•م‚Œمپںمپ®مپ¯م€پ1869ه¹´م€پم‚¹م‚¤م‚¹مپ®هŒ»ه¸«مƒ•مƒھمƒ¼مƒ‰مƒھمƒƒمƒ’مƒ»مƒںمƒ¼م‚·م‚§مƒ«مپ«م‚ˆمپ£مپ¦م€په»ƒو£„مپ•م‚Œمپںو‰‹è،“用هŒ…ه¸¯مپ®è†؟(مپ†مپ؟)مپ®ن¸مپ‹م‚‰ه¾®ه°ڈمپھ物è³ھم‚’ç™؛見مپ—مپںو™‚مپ«مپ•مپ‹مپ®مپ¼م‚‹م€‚ç´°èƒو ¸مپ«هکهœ¨مپ™م‚‹مپ“مپ¨مپ‹م‚‰م€په½¼مپ¯مپ“م‚Œم‚’م€ŒمƒŒم‚¯مƒ¬م‚¤مƒ³ï¼ˆè‹±: nuclein)م€چمپ¨ه‘½هگچمپ—مپں[187][188]م€‚1878ه¹´م€پم‚¢مƒ«مƒ–مƒ¬مƒ’مƒˆمƒ»م‚³مƒƒم‚»مƒ«مپŒم€ŒمƒŒم‚¯مƒ¬م‚¤مƒ³م€چمپ®éم‚؟مƒ³مƒ‘م‚¯è³ھوˆگهˆ†مپ§مپ‚م‚‹و ¸é…¸م‚’هچک離مپ—م€پمپمپ®ه¾Œم€پ5مپ¤مپ®و¨™و؛–و ¸é…¸ه،©هں؛م‚’هچک離مپ—مپں[189][190]م€‚ 1909ه¹´م€پمƒ•م‚£مƒ¼مƒگم‚¹مƒ»مƒ¬مƒ´م‚£مƒ¼مƒ³مپ¯RNA(ه½“و™‚مپ¯م€Œé…µو¯چو ¸é…¸ï¼ˆè‹±: yeast nucleic acid)م€چمپ¨ه‘¼م‚“مپ )مپ®ه،©هں؛م€پç³–م€پمƒھمƒ³é…¸مپ®مƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰هچکن½چم‚’هگŒه®ڑمپ—مپں[191][192][193]م€‚1929ه¹´م€پمƒ¬مƒ´م‚£مƒ¼مƒ³مپ¯DNA(ه½“و™‚مپ¯م€Œèƒ¸è…؛و ¸é…¸ï¼ˆè‹±: thymus nucleic acid)م€چ)ه†…مپ®مƒ‡م‚ھم‚م‚·مƒھمƒœمƒ¼م‚¹ç³–م‚’هگŒه®ڑمپ—مپں[194]م€‚مƒ¬مƒ´م‚£مƒ¼مƒ³مپ¯م€پDNAمپ¯مƒھمƒ³é…¸هں؛مپ«م‚ˆمپ£مپ¦çµگهگˆمپ•م‚Œمپں4مپ¤مپ®مƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰هچکن½چمپ‹م‚‰مپھم‚‹ç´گمپ§و§‹وˆگمپ•م‚Œمپ¦مپ„م‚‹مپ“مپ¨م‚’وڈگو،ˆمپ—مپں(مƒ†مƒˆمƒ©مƒŒم‚¯مƒ¬م‚ھمƒپمƒ‰ن»®èھ¬ï¼‰م€‚مƒ¬مƒ´م‚£مƒ¼مƒ³مپ¯م€پمپ“مپ®éژ–مپ¯çںمپڈم€په،©هں؛مپŒن¸€ه®ڑمپ®é †ه؛ڈمپ§ç¹°م‚ٹè؟”مپ•م‚Œمپ¦مپ„م‚‹مپ¨è€ƒمپˆمپںم€‚1927ه¹´م€پمƒ‹م‚³مƒ©م‚¤مƒ»م‚³مƒ«مƒ„م‚©مƒ•مپ¯م€پéپ؛ن¼ه½¢è³ھمپ¯م€Œمپم‚Œمپم‚Œمپ®éژ–م‚’鋳ه‹مپ¨مپ—مپ¦هچٹن؟هکçڑ„مپ«è¤‡è£½مپ•م‚Œم‚‹2وœ¬مپ®éڈ،هƒڈéژ–م€چمپ‹م‚‰مپھم‚‹م€Œه·¨ه¤§مپھéپ؛ن¼هˆ†هگم€چم‚’ن»‹مپ—مپ¦éپ؛ن¼مپ™م‚‹مپ¨وڈگو،ˆمپ—مپں[195][196]م€‚1928ه¹´م€پمƒ•مƒ¬مƒ‡مƒھمƒƒم‚¯مƒ»م‚°مƒھمƒ•م‚£م‚¹مپ¯ه®ں験مپ«م‚ˆمپ£مپ¦م€پè‚؛ç‚ژçگƒèڈŒï¼ˆPneumococcus)مپ®Sه‹èڈŒمپ®ه½¢è³ھمپŒم€پو»و»…مپ—مپںSه‹èڈŒمپ¨ç”ںمپچمپںRه‹èڈŒمپ¨م‚’و··هگˆمپ™م‚‹مپ“مپ¨مپ«م‚ˆمپ£مپ¦م€پRه‹èڈŒمپ«è»¢وڈ›مپ§مپچم‚‹مپ“مپ¨م‚’ç™؛見مپ—مپں(م‚°مƒھمƒ•م‚£م‚¹مپ®ه®ں験)[197][198]م€‚مپ“مپ®ه®ں験系مپ¯م€پDNAمپŒéپ؛ن¼وƒ…ه ±م‚’ن¼éپ”مپ—مپ¦مپ„م‚‹مپ“مپ¨م‚’هˆم‚پمپ¦وکژç¢؛مپ«ç¤؛ه”†مپ—مپںم€‚ 1933ه¹´م€پم‚¦مƒ‹مپ®وœھهڈ—ç²¾هچµم‚’ç ”ç©¶مپ—مپ¦مپ„مپںم‚¸مƒ£مƒ³مƒ»مƒ–مƒ©مƒƒم‚·م‚§ï¼ˆJean Brachet)مپ¯م€پDNAمپ¯ç´°èƒو ¸مپ«هکهœ¨مپ—م€پRNAمپ¯ç´°èƒè³ھمپ«مپ®مپ؟هکهœ¨مپ™م‚‹مپ“مپ¨م‚’وڈگو،ˆمپ—مپںم€‚ه½“و™‚مپ¯م€پé…µو¯چو ¸é…¸ï¼ˆRNA)مپ¯و¤چ物مپ مپ‘مپ«م€پ胸è…؛و ¸é…¸ï¼ˆDNA)مپ¯ه‹•ç‰©مپ مپ‘مپ«هکهœ¨مپ™م‚‹مپ¨è€ƒمپˆم‚‰م‚Œمپ¦مپ„مپںم€‚ه¾Œè€…مپ¯ç´°èƒه†…pHم‚’ç·©è،مپ™م‚‹و©ں能م‚’وŒپمپ¤ه››é‡ڈن½“مپ§مپ‚م‚‹مپ¨è€ƒمپˆم‚‰م‚Œمپ¦مپ„مپں[199][200]م€‚ 1937ه¹´م€پم‚¦م‚£مƒھم‚¢مƒ مƒ»م‚¢م‚¹مƒˆمƒ™مƒھمƒ¼مپ¯م€پDNAمپŒè¦ڈه‰‡و£مپ—مپ„و§‹é€ م‚’وŒپمپ£مپ¦مپ„م‚‹مپ“مپ¨م‚’ç¤؛مپ™Xç·ڑه›وٹکمƒ‘م‚؟مƒ¼مƒ³م‚’هˆم‚پمپ¦ن½œوˆگمپ—مپں[201]م€‚ 1943ه¹´م€پم‚ھم‚؛مƒ¯مƒ«مƒ‰مƒ»م‚¢مƒ™مƒھمƒ¼مپ¯م€په…±هگŒç ”究者مپ§مپ‚م‚‹م‚³مƒھمƒ³مƒ»مƒم‚¯مƒمƒ¼مƒ‰م€پمƒم‚¯مƒھمƒ³مƒ»مƒمƒƒم‚«مƒ¼مƒ†م‚£مپ¨مپ¨م‚‚مپ«م€پDNAمپŒه½¢è³ھ転وڈ›هژںçگ†مپ§مپ‚م‚‹مپ“مپ¨م‚’çھپمپچو¢م‚پم€پم‚°مƒھمƒ•م‚£م‚¹مپ®وڈگو،ˆم‚’و”¯وŒپمپ—مپں(م‚¢مƒ™مƒھمƒ¼-مƒم‚¯مƒمƒ¼مƒ‰-مƒمƒƒم‚«مƒ¼مƒ†م‚£مپ®ه®ں験)[202]م€‚م‚¨مƒ«مƒ´م‚£مƒ³مƒ»م‚·مƒ£مƒ«م‚¬مƒ•مپ¯م€پçڈ¾هœ¨م€Œم‚·مƒ£مƒ«م‚¬مƒ•مپ®و³•ه‰‡م€چمپ¨مپ—مپ¦çں¥م‚‰م‚Œم‚‹è¦‹è§£م‚’ç™؛è،¨مپ—م€پمپ©مپ®ç”ں物種مپ®DNAمپ«مپٹمپ„مپ¦م‚‚م€پم‚°م‚¢مƒ‹مƒ³مپ®é‡ڈمپ¯م‚·مƒˆم‚·مƒ³مپ¨ç‰مپ—مپڈم€پم‚¢مƒ‡مƒ‹مƒ³مپ®é‡ڈمپ¯مƒپمƒںمƒ³مپ¨ç‰مپ—مپڈمپھمپ‘م‚Œمپ°مپھم‚‰مپھمپ„مپ¨è؟°مپ¹مپں[203][204]م€‚  1951ه¹´وœ«م€پمƒ•مƒ©مƒ³م‚·م‚¹مƒ»م‚¯مƒھمƒƒم‚¯مپ¯م€پ英ه›½م‚±مƒ³مƒ–مƒھمƒƒم‚¸ه¤§ه¦مپ®م‚مƒ£مƒ´م‚§مƒ³مƒ‡م‚£مƒƒم‚·مƒ¥ç ”究و‰€مپ§م‚¸م‚§مƒ¼مƒ م‚؛مƒ»مƒ¯مƒˆم‚½مƒ³مپ¨مپ¨م‚‚مپ«ç ”究م‚’ه§‹م‚پمپںم€‚éپ؛ن¼مپ«مپٹمپ‘م‚‹DNAمپ®ه½¹ه‰²مپ¯م€پ1952ه¹´مپ«م‚¢مƒ«مƒ•مƒ¬مƒƒمƒ‰مƒ»مƒڈمƒ¼م‚·مƒ¼مپ¨مƒمƒ¼م‚µمƒ»مƒپم‚§م‚¤م‚¹مپŒè،Œمپ£مپںن¸€é€£مپ®ه®ں験(مƒڈمƒ¼م‚·مƒ¼-مƒپم‚§م‚¤م‚¹ه®ں験)مپ§م€پDNAمپŒè…¸ه†…ç´°èڈŒمƒ•م‚،مƒ¼م‚¸T2مپ®éپ؛ن¼ç‰©è³ھمپ§مپ‚م‚‹مپ“مپ¨م‚’ç¤؛مپ—مپ¦ç¢؛èھچمپ•م‚Œمپں[205]م€‚ 1952ه¹´5وœˆم€پمƒم‚¶مƒھمƒ³مƒ‰مƒ»مƒ•مƒ©مƒ³م‚¯مƒھمƒ³مپ®وŒ‡ه°ژن¸‹مپ§ç ”究م‚’مپ—مپ¦مپ„مپںه¤§ه¦é™¢ç”ںم€پمƒ¬م‚¤مƒ¢مƒ³مƒ‰مƒ»م‚´م‚¹مƒھمƒ³م‚°مپ¯م€پé«کو°´ه’Œمƒ¬مƒ™مƒ«مپ§مپ®DNA Xç·ڑه›وٹکهƒڈم‚’و’®ه½±مپ—م€پم€ŒPhoto 51م€چمپ¨مƒ©مƒ™مƒ«م‚’ن»کمپ‘مپں[206]م€‚مپ“مپ®ه†™çœںمپ¯م€پمƒ¢مƒ¼مƒھم‚¹مƒ»م‚¦م‚£مƒ«م‚مƒ³م‚¹مپ‹م‚‰مƒ¯مƒˆم‚½مƒ³مپ¨م‚¯مƒھمƒƒم‚¯مپ«و¸،مپ•م‚Œمپںم‚‚مپ®مپ§م€په½¼م‚‰مپŒDNAمپ®و£مپ—مپ„و§‹é€ م‚’ه¾—م‚‹ن¸ٹمپ§و¥µم‚پمپ¦é‡چè¦پمپھم‚‚مپ®مپ§مپ‚مپ£مپںم€‚مƒ•مƒ©مƒ³م‚¯مƒھمƒ³مپ¯م‚¯مƒھمƒƒم‚¯مپ¨مƒ¯مƒˆم‚½مƒ³مپ«م€پن¸»éژ–مپ¯ه¤–هپ´مپ«مپھمپ‘م‚Œمپ°مپھم‚‰مپھمپ„مپ¨èھمپ£مپںم€‚مپم‚Œمپ¾مپ§مپ¯م€پمƒ©م‚¤مƒٹم‚¹مƒ»مƒمƒ¼مƒھمƒ³م‚°م‚„م€پمƒ¯مƒˆم‚½مƒ³مپ¨م‚¯مƒھمƒƒم‚¯م‚‰مپ¯م€پéژ–مپŒه†…هپ´مپ«مپ‚مپ£مپ¦ه،©هں؛مپŒه¤–هپ´م‚’هگ‘مپ„مپںèھ¤مپ£مپںمƒ¢مƒ‡مƒ«م‚’وŒپمپ£مپ¦مپ„مپںم€‚مƒ•مƒ©مƒ³م‚¯مƒھمƒ³مپŒDNAçµگو™¶مپ®ç©؛間群م‚’特ه®ڑمپ—مپںمپ“مپ¨مپ§م€پم‚¯مƒھمƒƒم‚¯مپ¯م€پDNAمپ®ن؛Œوœ¬éژ–مپŒé€†ه¹³è،Œمپ§مپ‚م‚‹مپ“مپ¨م‚’çھپمپچو¢م‚پمپں[207]م€‚1953ه¹´2وœˆم€پمƒ©م‚¤مƒٹم‚¹مƒ»مƒمƒ¼مƒھمƒ³م‚°مپ¨مƒمƒگمƒ¼مƒˆمƒ»م‚³مƒھمƒ¼مپ¯م€پمƒھمƒ³é…¸مپŒè»¸مپ®è؟‘مپڈمپ«مپ‚م‚ٹم€په،©هں؛مپŒه¤–هپ´مپ«مپ‚م‚‹م€پ3وœ¬مپ®éژ–مپŒçµ،مپ؟هگˆمپ£مپںو ¸é…¸مپ®مƒ¢مƒ‡مƒ«م‚’وڈگو،ˆمپ—مپںم€‚مƒ¯مƒˆم‚½مƒ³مپ¨م‚¯مƒھمƒƒم‚¯مپ¯مپمپ®مƒ¢مƒ‡مƒ«م‚’ه®Œوˆگمپ•مپ›م€پçڈ¾هœ¨مپ§مپ¯DNAن؛Œé‡چم‚‰مپ›م‚“مپ®وœ€هˆمپ®و£مپ—مپ„مƒ¢مƒ‡مƒ«مپ¨مپ—مپ¦هڈ—مپ‘ه…¥م‚Œم‚‰م‚Œمپ¦مپ„م‚‹[208]م€‚1953ه¹´2وœˆ28و—¥م€پم‚¯مƒھمƒƒم‚¯مپ¯م€پ英ه›½م‚±مƒ³مƒ–مƒھمƒƒم‚¸مپ®م‚¶مƒ»م‚¤مƒ¼م‚°مƒ«مƒ‘مƒ–مپ§ه¸¸é€£ه®¢مپ®مƒ©مƒ³مƒپم‚؟م‚¤مƒ م‚’ن¸و–مپ—م€په½¼مپ¨مƒ¯مƒˆم‚½مƒ³مپŒم€Œç”ںه‘½مپ®ç§که¯†م‚’ç™؛見مپ—مپںم€چمپ¨ç™؛è،¨مپ—مپں[209]م€‚ 1953ه¹´4وœˆ25و—¥م€پ雑èھŒم€ŒNatureم€چمپ¯م€پمƒ¯مƒˆم‚½مƒ³مپ¨م‚¯مƒھمƒƒم‚¯مپ®ن؛Œé‡چم‚‰مپ›م‚“و§‹é€ DNAمپ¨مپم‚Œم‚’و”¯وŒپمپ™م‚‹è¨¼و‹ م‚’ç¤؛مپ™ن¸€é€£مپ®5وœ¬مپ®è«–و–‡م‚’وژ²è¼‰مپ—مپں[210]م€‚مپمپ®و§‹é€ مپ¯م€پم€ژMOLECULAR STRUCTURE OF NUCLEIC ACIDS A Structure for Deoxyribose Nucleic Acid(و ¸é…¸مپ®هˆ†هگو§‹é€ : مƒ‡م‚ھم‚م‚·مƒھمƒœمƒ¼م‚¹و ¸é…¸مپ®و§‹é€ )م€ڈمپ¨é،Œمپ•م‚Œمپںمƒ¬م‚؟مƒ¼مپ§ه ±ه‘ٹمپ•م‚Œم€پمپمپ®ن¸مپ§ه½¼م‚‰مپ¯و¬،مپ®م‚ˆمپ†مپ«è؟°مپ¹مپ¦مپ„م‚‹م€‚م€ژç§پمپںمپ،مپŒن»®ه®ڑمپ—مپں特異çڑ„مپھه¯¾ه½¢وˆگمپŒم€پéپ؛ن¼ç‰©è³ھمپ®è¤‡è£½مƒ،م‚«مƒ‹م‚؛مƒ مپ§مپ‚م‚‹هڈ¯èƒ½و€§م‚’هچ³ه؛§مپ«ç¤؛ه”†مپ—مپ¦مپ„م‚‹مپ“مپ¨م‚’م€پç§پمپںمپ،مپ¯è¦‹é€ƒمپ•مپھمپ‹مپ£مپں[9]م€ڈم€‚مپ“مپ®ه¾Œم€پمƒ•مƒ©مƒ³م‚¯مƒھمƒ³مپ¨م‚´م‚¹مƒھمƒ³م‚°مپ®مƒ¬م‚؟مƒ¼مپŒç¶ڑمپچم€په½¼م‚‰è‡ھè؛«مپ®Xç·ڑه›وٹکمƒ‡مƒ¼م‚؟مپ¨ç‹¬è‡ھمپ®è§£وگو–¹و³•مپŒهˆم‚پمپ¦ه…¬è،¨مپ•م‚Œمپں[48][211]م€‚مپ•م‚‰مپ«م€پم‚¦م‚£مƒ«م‚مƒ³م‚¹مپ¨ه½¼مپ®هگŒهƒڑ2هگچمپ«م‚ˆم‚‹مƒ¬م‚؟مƒ¼مپŒç¶ڑمپچم€پç”ںن½“ه†…مپ«مپٹمپ‘م‚‹B-DNA Xç·ڑمƒ‘م‚؟مƒ¼مƒ³مپ®è§£وگمپŒه ±ه‘ٹمپ•م‚Œمپ¦مپٹم‚ٹم€پç”ںن½“ه†…مپ«مƒ¯مƒˆم‚½مƒ³مپ¨م‚¯مƒھمƒƒم‚¯مپ®و§‹é€ مپŒهکهœ¨مپ™م‚‹مپ“مپ¨م‚’è£ڈن»کمپ‘مپ¦مپ„مپں[49]م€‚ 1962ه¹´م€پمƒ•مƒ©مƒ³م‚¯مƒھمƒ³مپ®و»ه¾Œم€پمƒ¯مƒˆم‚½مƒ³م€پم‚¯مƒھمƒƒم‚¯م€پم‚¦م‚£مƒ«م‚مƒ³م‚¹مپ®3هگچمپ¯مƒژمƒ¼مƒ™مƒ«ç”ںçگ†ه¦مƒ»هŒ»ه¦è³م‚’ه…±هگŒهڈ—è³مپ—مپں[212]م€‚مƒژمƒ¼مƒ™مƒ«è³مپ¯هکه‘½ن¸مپ®هڈ—è³è€…مپ«مپ®مپ؟وژˆن¸ژمپ•م‚Œم‚‹م€‚2023ه¹´4وœˆم€پ科ه¦è€…مپںمپ،مپ¯و–°مپںمپھ証و‹ مپ«هں؛مپ¥مپچم€پمƒم‚¶مƒھمƒ³مƒ‰مƒ»مƒ•مƒ©مƒ³م‚¯مƒھمƒ³مپ¯DNAç™؛見مپ®éپژ程مپ«è²¢çŒ®مپ—مپںمپ مپ‘مپ§مپھمپڈم€Œه¯¾ç‰مپھه½¹ه‰²م€چم‚’وœمپںمپ—مپںن؛؛物مپ§مپ‚م‚ٹم€پç™؛見ه¾Œمپ«ç™؛è،¨مپ•م‚Œمپںم‚ˆمپ†مپھ貢献者مپ§مپ¯مپھمپ„مپ¨çµگè«–مپ¥مپ‘مپں[213][214][215]م€‚èھ°مپŒمپ“مپ®ç™؛見مپ®هٹں績م‚’称مپˆم‚‰م‚Œم‚‹مپ¹مپچمپ‹مپ«مپ¤مپ„مپ¦مپ¯è°è«–مپŒç¶ڑمپ„مپ¦مپ„م‚‹[216]م€‚ 1957ه¹´مپ«è،Œم‚ڈم‚Œمپںه½±éں؟هٹ›مپ®مپ‚م‚‹è¬›و¼”مپ§م€پم‚¯مƒھمƒƒم‚¯مپ¯م€پهˆ†هگç”ں物ه¦مپ«مپٹمپ‘م‚‹م‚»مƒ³مƒˆمƒ©مƒ«مƒ»مƒ‰م‚°مƒم‚’و‰“مپ،ه‡؛مپ—م€پDNAم€پRNAم€پم‚؟مƒ³مƒ‘م‚¯è³ھمپ®é–¢ن؟‚م‚’ن؛ˆè¨€مپ—م€پم€Œم‚¢مƒ€مƒ—م‚؟مƒ¼ن»®èھ¬م€چم‚’ه…¬مپ«مپ—مپں[217]م€‚ن؛Œé‡چم‚‰مپ›م‚“و§‹é€ مپŒç¤؛ه”†مپ™م‚‹è¤‡è£½و©ںو§‹مپ®وœ€çµ‚ç¢؛èھچمپ¯م€پ1958ه¹´مپ®مƒ،م‚»مƒ«م‚½مƒ³-م‚¹م‚؟مƒ¼مƒ«مپ®ه®ں験مپ«م‚ˆمپ£مپ¦مپھمپ•م‚Œمپں[218]م€‚م‚¯مƒھمƒƒم‚¯مپ¨ه…±هگŒç ”究者م‚‰مپ«م‚ˆم‚‹و›´مپھم‚‹ç ”究مپ«م‚ˆمپ£مپ¦م€پéپ؛ن¼وڑ—هڈ·مپŒم‚³مƒ‰مƒ³مپ¨ه‘¼مپ°م‚Œم‚‹ه،©هں؛مپ®éé‡چ複مƒˆمƒھمƒ—مƒ¬مƒƒمƒˆï¼ˆن¸‰é€£ç¬¦ï¼‰مپ«هں؛مپ¥مپ„مپ¦مپ„م‚‹مپ“مپ¨مپŒوکژم‚‰مپ‹مپ«مپ•م‚Œم€پمƒڈمƒ¼مƒ»م‚´مƒ“مƒ³مƒ‰مƒ»م‚³مƒ©مƒٹم€پمƒمƒگمƒ¼مƒˆمƒ»Wمƒ»مƒ›مƒھمƒ¼م€پمƒمƒ¼م‚·مƒ£مƒ«مƒ»مƒ‹مƒ¼مƒ¬مƒ³مƒگمƒ¼م‚°مپ«م‚ˆمپ£مپ¦éپ؛ن¼وڑ—هڈ·مپ®è§£èھمپŒهڈ¯èƒ½مپ¨مپھمپ£مپں[219]م€‚هˆ†هگç”ں物ه¦مپ®èھ•ç”ںمپ¯م€پمپ“م‚Œم‚‰مپ®ç™؛見مپŒهں؛ç¤ژمپ¨مپھمپ£مپں[220]م€‚ 1986ه¹´م€پ英ه›½مپ®è¦ه¯ںمپŒمƒ¬م‚¹م‚؟مƒ¼ه¤§ه¦مپ®م‚¢مƒ¬مƒƒم‚¯مƒ»م‚¸م‚§مƒ•مƒھمƒ¼م‚؛مپ«ه¼·ه§¦و®؛ن؛؛مپ«é–¢مپ™م‚‹ه®¹ç–‘者مپ®è‡ھ白مپ®و¤œè¨¼مپ¾مپںمپ¯هڈچ証م‚’ن¾é ¼مپ—مپںمپ¨مپچم€پDNAé‘‘ه®ڑمپ¯هˆم‚پمپ¦çٹ¯ç½ھوچœوں»مپ«هˆ©ç”¨مپ•م‚Œمپںم€‚مپ“مپ®ç‰¹هˆ¥مپھن؛‹ن»¶مپ§مپ¯م€په®¹ç–‘者مپ¯2ن»¶مپ®ه¼·ه§¦و®؛ن؛؛م‚’è‡ھ白مپ—مپ¦مپ„مپںمپŒم€په¾Œمپ«è‡ھ白م‚’و’¤ه›مپ—مپںم€‚ه¤§ه¦مپ®ç ”究و‰€مپ§مپ®DNAé‘‘ه®ڑمپ«م‚ˆمپ£مپ¦م€په®¹ç–‘者مپ®ه½“هˆمپ®م€Œè‡ھ白م€چمپ®çœںه®ںو€§مپ¯مپ™مپگمپ«هگ¦ه®ڑمپ•م‚Œم€په®¹ç–‘者مپ¯ه¼·ه§¦و®؛ن؛؛مپ®ه®¹ç–‘م‚’و™´م‚‰مپ™مپ“مپ¨مپŒمپ§مپچمپں[221]م€‚ 符هڈ·ن½چç½®
هڈ‚ç…§é …ç›®
è„ڑو³¨
وژ¨è–¦و–‡çŒ®
ه¤–部مƒھمƒ³م‚¯
|
|||||||||||||||||||||||||||||||||||||
Portal di Ensiklopedia Dunia