核DNAがヘキスト染色 されたHeLa細胞 。中央と右の細胞は間期 にあり、核全体が標識されている。左の細胞は有糸分裂 の最中であり、DNAが凝縮されている。 細胞核 (さいぼうかく、英 : cell nucleus 、単に核 とも)は、真核生物 にみられる、膜に覆われた細胞小器官 である。通常、真核生物の細胞には核は1つだけ存在するが、哺乳類 の赤血球 は核を持たず、破骨細胞 には多数の核が存在するなど、例外的な細胞種もいくつか存在する。核を構成する主要な構造は核膜 と核マトリックス (英語版 )
核にはその細胞のゲノム のほぼ全てが含まれており、多くの場合、核DNA は複数の染色体 へと組織化されている。染色体は長い直鎖状のDNA 分子にさまざまな種類のタンパク質 (ヒストン など)が点在する構成をしており、タンパク質はDNAを保護し組織化する役割を果たしている。また、染色体内に存在する遺伝子 は細胞機能を促進するように構造化されている。核は遺伝子の完全性を維持し、遺伝子発現 を調節することで細胞の活動を制御している。
巨大な分子は核膜を透過することができないため、核膜を越える輸送の調節には核膜孔 が必要とされる。核膜の二重膜構造を貫通する核膜孔は、輸送タンパク質を介した能動的輸送を必要とする巨大分子が通過するためのチャネルとなっており、また低分子やイオン は孔を自由に通過する。遺伝子発現と染色体の維持の双方のプロセスにおいて、タンパク質やRNA などの高分子が核膜孔を通って移動することが必要である。核の内部には膜に覆われた小区画は存在しないが、特定のタンパク質、RNA分子、染色体の特定の部分から構成される核内構造体が多数存在する。こうした構造体の中では核小体 が最もよく知られており、主にリボソーム の組み立てに関与している。
細胞核には細胞の遺伝物質の大部分が複数の直鎖状のDNA分子の形で含まれており、染色体と呼ばれる構造に組織化されている。ヒトの各細胞にはおおよそ2 mの長さのDNAが含まれている[ 1] :405 。細胞周期 のほとんどの期間では、核内のDNAはクロマチン と呼ばれるDNA-タンパク質複合体へ組織化されている。細胞分裂 の期間には、核型 の図で良く知られた、明確な染色体構造を形成しているのが観察される。また、細胞内の遺伝子の一部は、核ではなくミトコンドリア に位置している[ 1] :438 。
クロマチンには2つの種類が存在する。ユークロマチン はDNAの詰め込みがが比較的緩やかな形態であり、高頻度で発現している遺伝子が含まれている[ 2] ヘテロクロマチン はユークロマチンよりもコンパクトにDNAが詰め込まれた形態であり、まれにしか転写 されないDNAが含まれている。ヘテロクロマチンにはさらに、特定の細胞種または特定の発生段階でのみヘテロクロマチンとして組織化される条件的ヘテロクロマチン(facultative heterochromatin)領域と、テロメア やセントロメア のように染色体の構造的構成要素として常にヘテロクロマチン化している構成的ヘテロクロマチン(constitutive heterochromatin)領域に分類される[ 3] 間期 の間のクロマチンは、各染色体ごとに染色体テリトリー (英語版 ) [ 4] [ 5] [ 6]
特定種のクロマチン構造、特にヌクレオソーム に対する抗体 は、全身性エリテマトーデス など多くの自己免疫疾患 と関連している[ 7] 抗核抗体 と呼ばれ、多発性硬化症 の患者でも免疫系の全般的機能不全の一部として観察される[ 8]
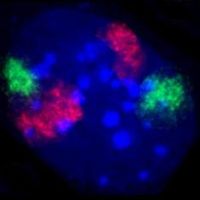
DNAが青く染色された、マウス線維芽細胞の核。さらにFISH 法によって2番染色体(赤)と9番染色体(緑)の染色体テリトリーが染色されている。
リボソームが点在する核膜外膜、核膜孔、クロマチンへと組織化されたDNA、核小体の模式図。 核にはその細胞のDNAのほぼ全てが含まれている。核内のDNAは核マトリックスと呼ばれる繊維状の中間径フィラメント ネットワークに囲まれており、核膜と呼ばれる二重膜構造によって内包されている。核膜は、細胞の他の部分から核質 と呼ばれる核内の流体を分離する役割を果たしている。核の大きさは細胞の大きさと相関しており、その比率はさまざまな細胞種や生物種で報告されている[ 9] [ 10] :178 。動物細胞では核は最も大きな細胞小器官であり[ 11] :12 、ヒト細胞では核の直径は約6 μmである[ 10] :179 。
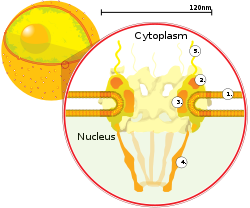
核膜表面の核膜孔の断面図。①核膜、②アウターリング、③スポーク、④核バスケット、⑤細胞質フィラメント。 核膜 は内膜と外膜の2つの脂質二重層 によって構成されており、核膜孔 は両者を貫通している[ 10] :649 。内膜と外膜は細胞の遺伝物質を他の細胞内容物から分離する役割を果たしており、核内が細胞内の他の部分とは異なる環境となるよう維持している。これら2つの膜は核の大部分を囲んで並置されているが、その形状や含有物に関しては大きく異なる。内膜は核の内容物を取り囲んでおり、明確な境界を形成している[ 11] :14 。内膜にはさまざまなタンパク質が埋め込まれており、これらは中間径フィラメントに結合して核の構造の形成に寄与している[ 10] :649 。外膜は内膜を内包しており、隣接する小胞体 膜と連続的に膜を形成している。このように外膜は小胞体膜の一部でもあるため、膜を越えてタンパク質を活発に翻訳 しているリボソーム が膜上に点在している[ 10] :649 。内膜と外膜の間の領域は perinuclear space と呼ばれ、この空間は小胞体内腔と連続している[ 10] :649 。
哺乳類の核膜には、核膜を貫通する核膜孔複合体(NPC)が一般的には3000個から4000個存在している[ 10] :650 。内膜と外膜が融合する地点において、NPCは8回対称のリング構造を形成している[ 12] グリア細胞 では数百個しか存在しないのに対し、大きなプルキンエ細胞 では約20000個も存在する[ 10] :650 。NPCは核質と細胞質基質 の間での分子選択的輸送を可能にしている[ 13] ヌクレオポリン と呼ばれる約30種類の異なるタンパク質から構成されている[ 10] :649 。大きさは60–80 MDa であり、酵母 では約50個、脊椎動物 では数百個のタンパク質が含まれている[ 11] :622–4 。核膜孔全体の直径は100 nmであるが、孔の中心部には調節システムが存在するため、分子が自由に拡散することができる隙間はわずか9 nmである。このサイズは水溶性の小分子の通過は可能となるが、核酸やタンパク質などは核を自由に出入りすることはできず、こうした分子は核内へ能動的に輸送する必要がある。このリング構造からは核バスケット (nuclear basket) と呼ばれる構造が核質側へ突出しており、細胞質側へは複数の繊維状構造が伸びている。どちらの構造も核輸送タンパク質の結合を媒介している[ 1] :509–10 。
ほとんどのタンパク質、リボソームサブユニット、そして一部のRNA は、カリオフェリン と呼ばれる輸送因子ファミリーによって、核膜孔複合体を通って輸送される。核内への移動を媒介するカリオフェリンはインポーチン 、核外への移動を媒介するものはエクスポーチン と呼ばれる。ほとんどのカリオフェリンはその積み荷と直接的に相互作用するが、一部はアダプタータンパク質 を介して結合する[ 14] コルチゾール やアルドステロン などのステロイドホルモン や、細胞間シグナル伝達 に関与する他の脂溶性低分子は細胞膜を通過して細胞質へ拡散し、そこで核内受容体 と結合して核内へと輸送される。核内受容体はリガンド の結合時には転写因子 として機能する[ 1] :482 。
動物細胞の核は、中間径フィラメント ネットワークによって機械的に支持されている。核膜の内側は核ラミナ がメッシュ状に組織化されたネットワークを形成している一方で、核膜の細胞質側の支持は組織化の程度は比較的低い。双方の系が核膜の機械的支持や、染色体や核膜孔のアンカー部位として機能している[ 15]
核ラミナの大部分はラミン タンパク質で構成されている。他の全てのタンパク質と同様にラミンは細胞質で合成される。その後で核の内部へ輸送され、そこで重合して既存の核ラミナネットワークに組み込まれる[ 16] [ 17] エメリン 、細胞質側のネスプリン (英語版 ) [ 18] 蛍光顕微鏡 で観察可能な他の規則的構造を形成している[ 19] [ 20] 核小体 からは排除されており、細胞周期 の間期 に存在する[ 21] クロマチン と相互作用しており、これらの構造を破壊するとタンパク質をコードする遺伝子の転写 が阻害される[ 22]
他の中間径フィラメントの構成要素と同様に、ラミンの単量体 はα-ヘリカル ドメインを持っており、2分子の単量体が互いに巻きついてコイルドコイル と呼ばれる二量体 構造を形成する。そして、2つの二量体が逆平行型に並んで結合し、プロトフィラメント(protofilament)と呼ばれる四量体 構造が形成される。さらに8つのプロトフィラメントが並び、ねじれることでロープ状のフィラメントが形成される。こうしたフィラメントが動的に重合・脱重合を行い、その競合によってフィラメントの長さが変化する[ 15]
フィラメント重合に欠陥をもたらすラミン遺伝子の変異は、ラミノパチー と呼ばれる希少遺伝子疾患 群の原因となる。ラミノパチーの中で最も有名なものはプロジェリア と呼ばれる疾患ファミリーであり、患者では早老 が引き起こされる。老化の表現型 を生じさせる生化学 的変化の正確な機構は、まだ十分には理解されていない[ 23]
細胞核の電子顕微鏡像。核小体 が暗く染色されている。 核小体 は、核内で濃密に染色される、膜を持たない構造体(核内構造体 (英語版 ) リボソームRNA (rRNA)をコードするDNA(rDNA )のタンデムリピートの周囲に形成される。こうしたDNA領域は核小体形成域 と呼ばれる。核小体の主な役割はrRNAの合成とリボソーム の組み立てである。核小体の構造的凝集はその活性に依存している。核小体でのリボソームの組み立てが核小体構成要素間の一過的結合をもたらし、それによってさらにリボソームの組み立てが促進され、さらに結合が進行する。このモデルは、rDNAの不活性化によって組織的な核小体構造の混合が引き起こされる観察によって支持されている[ 24]
リボソームの組み立ての最初の段階では、RNAポリメラーゼI と呼ばれるタンパク質がrDNAを転写し、大きなpre-rRNA前駆体が形成される。そして、5.8S 、18S (英語版 ) 28S rRNA のサブユニットへ切断される[ 10] :328 [ 25] 核小体低分子RNA (snoRNA)の助けによって行われる。snoRNAのいくつかは、リボソームの機能に関連する遺伝子をコードするmRNA からスプライシング されたイントロン に由来する。組み立てられたリボソームサブユニットは核膜孔を通過する最も大きな構造体である[ 1] :526 。
電子顕微鏡 による観察では、核小体は3つの判別可能な領域から構成されていることが観察される。最も内側の繊維状中心部(fibrillar center [FC])、それを取り囲む高密度繊維状部(dense fibrillar component [DFC]、フィブリラリン (英語版 ) ヌクレオリン (英語版 ) ヌクレオフォスミン が含まれる)である。rDNAの転写はFCまたはFC-DFC境界で起こり、そのため細胞でrRNAの転写が増加するとFCがより多く検出されるようになる。rRNAの切断と修飾の大部分はDFCで行われ、リボソームサブユニットへのタンパク質の組み込みを伴う後半のステップはGCで行われる[ 25]
核スペックル(スペックル、スプライシングスペックル、speckle)は、pre-mRNAスプライシング因子 に富む核内構造体で、哺乳類細胞の核質のクロマチン間領域(interchromatin region)に位置している[ 26] クロマチン間顆粒 (英語版 ) p53 と協働し、特定の遺伝子の活性を直接高める場合がある。p53の標的遺伝子の中でも、スペックルと関連したものとそうでないものとは機能的に異なっている[ 27]
核スペックルの構成、構造、挙動についての研究からは、核の機能的区画化と、遺伝子発現装置、スプライシングsnRNP 、pre-mRNAスプライシングに必要な他のタンパク質の組織化に関与しているというモデルが立てられている[ 28] [ 29] [ 30] リン酸化 による調節を介して変化する[ 31] [ 32] 両生類 の卵母細胞 の核や、キイロショウジョウバエ Drosophila melanogaster の胚 で観察されている。両生類の核の電子顕微鏡像では、B snurposome は単独で存在するか、もしくはカハール体 に付着しているように見える[ 33] [ 34] [ 35]
カハール体 典型的な核には、カハール体 (カハール小体、カハールボディ)またはコイル体(coiled body)と呼ばれる1個から10個のコンパクトな構造が存在し、その直径は生物種や細胞種によって異なるが 0.2 µm から 2.0 µm 程度である[ 36] 電子顕微鏡 下での観察では絡まった糸玉のような形状をしており[ 37] コイリン (英語版 ) [ 38] 核小体低分子RNA (snoRNA)や核内低分子RNA (snRNA)の成熟や、ヒストン mRNAの修飾などに関与している[ 36]
カハール体に類似した構造として、Gem(Gem小体、Gems、Gemini of coiled bodies、Gemini of Cajal bodies)がある。その名前はふたご座 (Gemini)に由来し、カハール体との密接な関係を表している。Gemは大きさと形状の面ではカハール体と類似しており、実際、顕微鏡下で視覚的に区別することはできない[ 38] 核内低分子リボヌクレオタンパク質 (snRNP)が含まれないが、snRNPの生合成に関連した機能を持つSMN (英語版 ) [ 39] [ 39] [ 38] 微細構造 の解析によって、Gemとカハール体との差異はコイリンにあることが示された。すなわち、カハール体はSMNとコイリンを含んでおり、GemはSMNを含むがコイリンを含まない[ 40]
核内構造体のサイズ
構造の名称
構造の直径
出典
カハール体
0.2–2.0 μm
[ 36]
クラストソーム
0.2–0.5 μm
[ 41]
PIKA
5 μm
[ 37]
PMLボディ
0.2–1.0 μm
[ 42]
パラスペックル
0.5–1.0 μm
[ 43]
核スペックル
20–25 nm
[ 37]
上述したサンティアゴ・ラモン・イ・カハール によって最初に記載された核内構造体(核小体、核スペックル、カハール小体)以外にも核には多くの構造体が含まれており、PIKA(polymorphic interphase karyosomal association)、PMLボディ(PML body)、パラスペックル (英語版 ) [ 42]
また、異常な疾患過程の一部として出現する核内構造体もある。一例として、ネマリンミオパチー (英語版 ) アクチン の変異が原因となっており、桿状構造は変異体アクチンやその他の細胞骨格タンパク質から構成されている[ 44]
PIKA
PIKA(polymorphic interphase karyosomal association)は、1991年に顕微鏡研究で初めて記載された。その機能は未だ明らかではないが、活発なDNA複製、転写、そしてRNAのプロセシングとは関連していないと考えられている[ 45] 転写因子 PTFの濃密な局在によって定義されるドメインとしばしば相互作用していることが判明している[ 46]
PMLボディ(PML体、PML小体、promyelocytic leukemia body)は核質中に分散して存在する球形の構造体で、大きさは約0.1–1.0 µm である。他の名称でも知られており、nuclear domain 10 (ND10)、Kremer body、PML oncogenic domain などとも呼ばれる[ 47] PML タンパク質に由来する。核内でカハール体やcleavage bodyと結合して存在していることがしばしば観察される[ 42] Pml 遺伝子を欠失しPMLボディが形成されないマウスも明確な悪影響を示すことなく正常に発生するため、PMLボディはほとんどの基礎的な生物学的機能に必要ではないことが示されている[ 48]
Foxらによって2002年に発見されたパラスペックル (英語版 ) [ 49] HeLa細胞 においてであり、核には一般的に10個から30個存在するとされた[ 50] 初代培養 細胞、形質転換細胞株、組織切片に存在することが知られている[ 51] [ 50]
パラスペックルは核内のタンパク質やRNAを隔離しており、遺伝子発現調節に関与する分子スポンジ(molecular sponge)として機能しているようである[ 52] [ 53] 転写 に依存的であり[ 54] RNAポリメラーゼII による転写がないときにはパラスペックルは消失する。こうした状況下では関連するタンパク質構成要素(PSP1、p54nrb、PSP2、CFI(m)68、PSF)は核小体 で三日月型のキャップ構造体(perinucleolar cap)を形成する。この現象は細胞周期 中でも実証されている。パラスペックルは間期 の間に存在し、有糸分裂 中も終期 を除いて存在する。2つの娘細胞 の核が形成される終期にはRNAポリメラーゼIIによる転写が行われないため、タンパク質構成要素は代わりにperinucleolar capを形成する[ 51]
Perichromatin fibril
Perichromatin fibrilと呼ばれる繊維状構造体は、電子顕微鏡下でのみ観察可能である。転写が活発なクロマチンに近接して位置し、pre-mRNAのプロセシングが活発に行われている場所であるという仮説が立てられている[ 34]
クラストソーム(clastosome)は小さな構造体で (0.2–0.5 µm)、構造体周縁部のカプセル構造のために厚いリング状に観察される[ 41] klastos (壊れた)と soma (体)に由来する[ 41] プロテアソーム 阻害剤によって処理されると解体される[ 41] [ 55] [ 56] 浸透圧ストレス もクラストソームの形成を引き起こすことが示されている[ 57] [ 56]
核は翻訳の場である細胞質から隔離された遺伝子転写のための場所を提供することで、原核生物 には利用できないレベルでの遺伝子調節 を可能にしている。核の主要な機能は遺伝子発現 の制御と細胞周期 進行中のDNA複製 の媒介である[ 1] :171 。
核膜は核の内容物の制御を可能にし、必要に応じてそれらを残りの細胞質部分から分離している。このことは、核膜の両側での過程の制御に重要な意味を持つ。細胞質での過程を制限する必要があるケースの大部分では、主要な関連因子は核へ除去され、またそこで転写因子 と相互作用してその経路の特定の酵素の産生をダウンレギュレーションする。こうした調節は、エネルギー産生のためにグルコース を分解する経路である解糖系 でも行われている。ヘキソキナーゼ は解糖系の第一段階を担う酵素であり、グルコースからグルコース-6-リン酸 を形成する。グルコース-6-リン酸から合成される分子であるフルクトース-6-リン酸 が高濃度で存在すると、調節タンパク質によってヘキソキナーゼは核へ除去され[ 58] リプレッサー 複合体を形成して解糖系に関与する遺伝子の発現を低下させる[ 59]
また細胞は、どの遺伝子を転写するかを制御するため、遺伝子発現調節を担う転写因子の一部を隔離し、他のシグナル伝達経路によって活性化されない限りそれらがDNAに物理的にアクセスすることがないようにしている。これによって、不適切な遺伝子発現がたとえ低レベルであっても生じることがないよう防がれている。一例として、ほとんどの炎症 反応に関与しているNF-κB によって制御される遺伝子の場合、シグナル伝達分子TNF-α によって開始される経路など、シグナル伝達経路への応答として転写が誘導される。TNF-αが細胞膜 の受容体 に結合すると細胞内のシグナル伝達タンパク質が膜へリクルートされ、最終的にNF-κBが活性化される。活性化されたNF-κBは核局在化シグナル によって核膜孔 を通って核内へ輸送され、標的遺伝子の転写を刺激する[ 15]
核膜による区画化は、スプライシング されていないmRNAの翻訳の防止も可能にしている[ 60] イントロン を含んでおり、翻訳によって機能的なタンパク質を産生するためにはイントロンを除去することが必要である。スプライシングは、リボソームがmRNAにアクセスするより前の核内の段階で行われる。核がなければ、リボソームは転写されたばかりの(プロセシングされていない)mRNAを翻訳してしまい、欠陥を有する非機能的タンパク質が合成されてしまう[ 1] :108–15 。
核の主な機能は、遺伝子発現の制御と細胞周期進行中のDNA複製の媒介である[ 1] :171 。複製は核内に局在化された形で生じ、細胞周期間期のS期 に行われる。従来、静的なDNAに沿って複製フォークが進んでゆくことで複製が進行すると考えられてきたが、複製ファクトリー(replication factory)と呼ばれる新たなモデルが提唱されている。このモデルでは、複製フォークはいくつかの固定化されたファクトリー領域に濃縮されており、鋳型となるDNA鎖がベルトコンベアのようにそこを通過するとされている[ 61]
転写時の転写ファクトリーの模式図。この図では、複数の遺伝子が一度に転写されている可能性が示されている。8個のRNAポリメラーゼが描かれているが、実際の数は細胞種に依存して異なる場合がある。また、転写因子や透過性の高いタンパク質コアも描かれている。 遺伝子発現 はまず転写 過程を伴い、そこではDNA を鋳型としてRNA が合成される。タンパク質をコードする遺伝子の場合、この過程で合成されるRNAはmRNA であり、タンパク質を合成するにはその後リボソーム によってmRNAが翻訳 される必要がある。リボソームは核外に位置しているため、合成されたmRNAは核外への輸送が必要である[ 62]
核は転写の場であるため、転写を直接的に媒介したり転写過程の調節に関与したりするさまざまな種類のタンパク質が含まれている。これらには、二本鎖DNAをほどいてDNAへのアクセスを促進するヘリカーゼ 、DNAのプロモーター 領域に結合してRNA分子を合成するRNAポリメラーゼ 、DNAのスーパーコイル構造 を変化させてDNAの巻き戻しなどを助けるトポイソメラーゼ 、発現を調節するさまざまな種類の転写因子などが含まれている[ 63]
新しく合成されたmRNA分子は、一次転写産物 またはpre-mRNAと呼ばれる。pre-mRNAは細胞質への輸送の前に核内で転写後修飾 を受けなければならず、こうした修飾を受けずに細胞質へ移動したmRNAはタンパク質への翻訳に利用されるのではなく、分解の標的となる。核内で行われる転写後修飾は、5'キャッピング 、3'ポリアデニル化 、そしてRNAスプライシング である。核内では、pre-mRNAはhnRNP と呼ばれる複合体中のさまざまなタンパク質と相互作用している。5'キャップの付加は転写と共役した形で行われ、転写後修飾の第一段階となっている。3'末端へのポリ(A)テール付加は、転写が完了した後に行われる[ 1] :509–18 。
RNAスプライシングはスプライソソーム と呼ばれる複合体によって行われ、イントロン (タンパク質をコードしていない領域)がpre-mRNAから除去され、残ったエクソン が1本の連続的なRNA分子として再形成される。この過程は通常、5'キャッピングと3'ポリアデニル化の後に起こるが、多くのエクソンからなる転写産物の場合には転写が完了する前に開始される[ 1] :494 。多くのpre-mRNAは複数通りにスプライシングされ、その結果、異なるタンパク質配列をコードする成熟mRNAが作り出される。この過程は選択的スプライシング として呼ばれ、限られた量のDNAから多様なタンパク質を生み出すことが可能となっている[ 64]
RNAやタンパク質などの高分子は、Ran -GTP核輸送サイクルと呼ばれる過程によって、核膜 を越える能動的輸送が行われる。 高分子の核への出入りは、核膜孔複合体によって緊密に制御されている。低分子は調節を受けることなく核へ入ることができるが[ 65] インポーチン 、核外へ出るにはエクスポーチン と呼ばれるカリオフェリン との相互作用を必要とする。細胞質から核へ移行するべき「積み荷」タンパク質には核局在化シグナル と呼ばれる短いアミノ酸配列が含まれており、そこへインポーチンが結合する。一方、核から細胞質へ輸送されるべきものには核外搬出シグナル が含まれており、エクスポーチンが結合する。インポーチンとエクスポーチンによる輸送は、GTP を加水分解 してエネルギーを放出する酵素であるGTPアーゼ によって調節されている。核輸送における主要なGTPアーゼはRan であり、核に位置するか細胞質に位置するかに応じて、GTPとGDP のいずれかを結合する。インポーチンはGTP結合型Ran(Ran-GTP)の結合によって積み荷を解離するが、一方エクスポーチンは積み荷を結合するためにRan-GTPを必要とする[ 14]
核内輸送は細胞質における積み荷へのインポーチンの結合に依存しており、積み荷を結合したインポーチンは核膜孔を通って核へ移動する、核内では、Ran-GTPがインポーチンから積み荷を降ろすために働き、積み荷を降ろしたインポーチンは核外へ出て再利用される。核外輸送も同様の機構で行われており、エクスポーチンはRan-GTPに促進される形で核内の積み荷を結合し、核膜孔を通って外へ出た後、細胞質で積み荷を降ろす[ 66]
転写後修飾が完了した後の、成熟したmRNAやtRNA を細胞質へ移動するためには、特別な核外搬出タンパク質が存在する。mRNAやtRNAはタンパク質の翻訳において中心的な役割を果たすため、この品質管理機構は重要である。イントロン除去が不完全であったりや誤ったアミノ酸が取り込まれることで誤ったタンパク質が発現されてしまうと、細胞には負の影響が生じる。そのため、修飾が不完全なRNAが細胞質に到着した場合には、翻訳には用いられず分解が行われる[ 1]
蛍光色素 で染色された、有糸分裂中期 のイモリ の肺 細胞。緑色に染色された紡錘体 へ、青色に染色された2セットの染色体 が接着している。1本を除く全ての染色体がすでに中期板(metaphase plate)に位置している。 細胞分裂 過程、またはアポトーシス の影響下(プログラム細胞死 過程)において、核は解体されたり破壊されたりする。これらのイベント中に、核の構造的構成要素(核膜や核ラミナ)は秩序だった分解が行われる。大部分の細胞において、核膜の解体は有糸分裂前期 の終盤に起こる。しかしながら、核の解体は有糸分裂の普遍的特徴ではなく、全ての細胞で起こるわけではない。いくつかの単細胞 真核生物(酵母 など)ではいわゆる"closed mitosis"が起こり、核膜は保持されたままである。Closed mitosisでは、娘染色体は核の両極に移動し、核はその後2つに分割される。一方、高等真核生物の細胞は通常"open mitosis"が起こり、核膜の解体を特徴とする。娘染色体は紡錘体 の両極に移動し、新たな核がその周辺で再び組み立てられる[ 1] :854 。
細胞周期の特定の時点で、細胞は2つへ分裂する。新たな娘細胞はそれぞれ完全な遺伝子のセットを持っていなければならず、そのためには染色体を複製し、それらを別々のセットへと分離することが必要となる。この過程は、複製された染色体(姉妹染色分体 )が微小管 を介してそれぞれ異なる中心体 と結合されているようにすることで行われている。その後、姉妹染色分体は細胞の別々の位置へと引き離される。多くの細胞では、中心体は核外の細胞質に位置しており、核膜が存在しているときには微小管は染色分体へ結合することはできない[ 67] 前中期 の段階までに、核膜は解体される[ 19] CDC2 などのプロテインキナーゼ によるラミン のリン酸化 で調節された過程によって、同時期に核ラミナの脱重合が生じる[ 68] 脱リン酸化 によって再重合する[ 68]
一方で渦鞭毛藻 では、核膜は保持されたままであり、そして中心体は細胞質に位置している。有糸分裂時には微小管は染色体と接触するようになり、セントロメア 領域は核膜に取り込まれる(この機構はclosed mitosis with extranuclear spindleと呼ばれる)。他の多くの原生生物 (繊毛虫 や胞子虫 など)や菌類 は、中心体は核内にあるため、細胞分裂中も核膜は解体されない[ 69]
アポトーシス は、制御された形で細胞の構造要素が破壊される過程であり、細胞死が誘導される。アポトーシスに関連した変化は核やその内容物に直接的影響を及ぼし、クロマチンは凝縮され、核膜や核ラミナは解体される。ラミンネットワークの破壊はアポトーシスの進行を担うカスパーゼ と呼ばれる特別なプロテアーゼ によって制御されており、ラミンが切断されることで核の構造的完全性が失われる。アポトーシスの初期活性のアッセイにおいて、ラミンの切断はカスパーゼ活性の実験的指標として用いられることもある[ 19] [ 19] [ 70]
核膜は、DNAウイルス やRNAウイルス が核へ進入するのを防ぐ障壁として機能している。いくつかのウイルスは、自身の複製と組み立てを行うために核内のタンパク質へアクセスする必要がある。ヘルペスウイルス などのDNAウイルスは、核内で複製と組み立てを行い、核の内膜からの出芽によって外部へ出る。この過程は、内膜の核側のラミナの解体を伴っている[ 19]
かつては、一般的には免疫グロブリン 、特に自己抗体 は核内へ進入することはないと考えられていた。現在では、全身性エリテマトーデス などの疾患状態においてはIgG が核に進入することを示す多くのエビデンスが蓄積されている[ 71]
通常、真核生物の大部分の細胞種は核を1つだけ持っているが、いくつかには核が存在せず、また他のものは複数の核を持っている。哺乳類の赤血球 の成熟のように正常な発生の結果である場合も、細胞分裂時の欠陥から生じている場合もある[ 72]
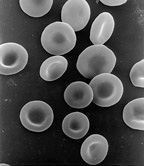
ヒトの赤血球細胞は、他の哺乳類と同様、核を失う。この変化は細胞発生の正常な過程として生じる。 無核細胞には核が存在せず、そのため分裂して娘細胞を生み出すことはできない。最もよく知られた無核細胞は哺乳類の赤血球である。赤血球はミトコンドリア など他の細胞小器官も欠いており、肺から体中の組織へ酸素 を運搬する輸送容器として主にはたらいている。赤血球は骨髄 での赤血球形成 過程を経て成熟し、そこで核、細胞小器官、リボソームを喪失する。核は赤芽球 から網赤血球 への分化過程で排出される[ 73] 変異原 の存在下では、一部の未成熟な小核赤血球の血中への放出が誘発される可能性がある[ 74] [ 75] 被子植物 の師管要素 でも生じる[ 76]
無核細胞は細胞分裂の欠陥から生じることもあり、一方の娘細胞は無核細胞に、他方は2つの核を持つ細胞となる。
多核細胞は複数の核を持つ細胞である。原生生物のアカンタリア の大部分の種[ 77] 菌根 菌の一部の種[ 78] ジアルジア 属の腸管寄生体があり、1細胞当たり2つの核を持つ[ 79] 繊毛虫 は、1細胞に大核 と小核 という2種類の核を有する[ 80] 骨格筋 の筋細胞 は発生過程で多核細胞となる。核は細胞の周縁部に配置され、筋線維 のための細胞内空間が最大化されている[ 1] 骨細胞 の一種である破骨細胞 が挙げられる。ヒトでは何らかの異常によって多核細胞や二核細胞 (英語版 ) 単球 とマクロファージ の融合によって形成される細胞は多核巨細胞(multinucleate giant cell)と呼ばれ、炎症 を伴い[ 81] 腫瘍 形成への関与も示唆されている[ 82]
多くの渦鞭毛藻 も2つの核を持つことが知られている。他の多核細胞とは異なり、2つの核はDNAの系統が異なる。1つは渦鞭毛藻のもので、もう1つは共生した珪藻 のものである[ 83]
核は真核生物の細胞を定義づける主要な特徴であるため、核の進化的起源は多くの思索の対象となってきた。核の存在を説明するために4つの主要な仮説が提唱されているが、どれもいまだ幅広く支持を得るには至っていない[ 84] [ 85] [ 86]
第一のモデルは「栄養共生モデル」(syntrophic model) として知られるもので、古細菌 と細菌 の共生 関係が核を持つ真核生物細胞を作り出したと提唱する。古細菌ドメイン ・細菌ドメインの生物は核を持たない[ 87] メタン菌 に似た太古の古細菌が、現代の粘液細菌 に似た細菌に侵入して生息したことに起源を持ち、最終的に初期の核が形成されたという仮説が立てられている。この理論は真核生物のミトコンドリア と葉緑体 の起源について広く受け入れられている理論(細胞内共生説 )と類似したものであり、その理論ではミトコンドリアと葉緑体は原始的な真核生物と好気性細菌 との内部共生 関係から発展したと考えられている[ 88] [ 89] 活性酸素種 による損傷から、ゲノムを保護する役割を核膜が果たした可能性がある[ 90] ヒストン など特定のタンパク質について似た遺伝子を持っていることから支持される。そして真核細胞が細菌起源であることは、粘液細菌が運動性であること、多細胞性複合体を形成することができること、真核生物と似たキナーゼ やGタンパク質 を持つことから支持される[ 91]
第二のモデルでは、始原真核生物は細菌から内部共生段階を経ることなく進化したと提唱される。このモデルは現代のプランクトミケス門 の細菌の存在に基づいており、これらの細菌には原始的な孔を持つ核構造や他の区画化された膜構造が存在する[ 92] 食作用 で取り込んで核や真核細胞が生み出されたという主張がなされている[ 93]
最も物議を醸しているモデルは「細胞核ウイルス起源説 」と呼ばれるもので、膜に覆われた核や、その他の真核生物の特徴は原核生物 にウイルス が感染したことで生じたとしている。この提案は、直鎖状のDNA、mRNAのキャッピングといった、真核生物とウイルスの間の類似性に基づいている。このモデルの1つのバージョンでは、核は食作用と共に進化し、初期の細胞の「捕食者」が形成されたと示唆されている[ 94] ポックスウイルス が感染したことで生じたと提唱されており、これは現代のポックスウイルスと真核生物のDNAポリメラーゼ の類似性に基づいている[ 95] [ 96] 有性生殖の進化 (英語版 ) [ 97]
より近年に提唱されたexomembrane hypothesisでは、核は外側に2つ目の細胞膜を進化させた単一の祖先細胞に起源を持つとされ、内側の膜が元の細胞を包み込んで核膜となり、リボソームサブユニッなど内部で合成される細胞構成要素の通り道として複雑な孔構造が進化したと示唆されている[ 98]
細胞と細胞核を描いた最古の例とされるアントニ・ファン・レーウェンフック によるスケッチ、1719年。 1882年に発表された、ヴァルター・フレミング によるユスリカ の唾液腺 細胞のスケッチ。核には多糸染色体 が含まれている。 核は最初に発見された細胞小器官である。現存する最古のスケッチはおそらく、初期の顕微鏡学者であったアントニ・ファン・レーウェンフック (1632–1723)によるものである。彼はサケ の赤血球 細胞の中に"lumen"と呼ばれるものを観察した[ 99] [ 100]
また、核はオーストリア の植物画家フランツ・バウアー によって1804年に記載され[ 101] スコットランド の植物学者 ロバート・ブラウン によって1831年にロンドン・リンネ協会 で発表された。ブラウンは顕微鏡下でラン の研究をしている際に、花の外層の細胞に不透明な領域を発見し、それを"areola"または"nucleus"と名付けた[ 102]
ブラウンはその領域の機能については示唆しなかったが、1838年にマティアス・ヤーコプ・シュライデン は核が細胞を生み出す役割を持つと提唱し、"cytoblast"(細胞を構築するもの)という名称を導入した。彼は、新たな細胞が"cytoblast"の周辺に集まって形成されるのを観察したと考えていた。フランツ・ユリウス・フェルディナント・マイエン はこの考えに強く反対しており、細胞が分裂によって増殖することをすでに記載していたため、多くの細胞は核を持たないと考えていた。"Cytoblast"であれ何であれ、細胞が「新たに」生じるという考えは、「全ての細胞は細胞から生じる」("Omnis cellula e cellula")という新たなパラダイムを広めるのに決定的な役割を果たしたロベルト・レーマク (1852)やルドルフ・ルートヴィヒ・カール・フィルヒョウ (1855)の業績とも矛盾していた。核の機能は依然として不明なままであった[ 103]
1877年から1878年の間に、オスカー・ヘルトヴィヒ (英語版 ) ウニ の発生に関するいくつかの研究を発表し、精子 の核が卵母細胞 に進入し、その核と融合することを示した。これは、個体が1個の有核細胞から発生することを初めて示唆したものであった。またこれは、個体発生 は系統発生 を反復する(反復説 )、構造を持たない原始的な原形質 の塊から最初の有核細胞が発生する、としていたエルンスト・ヘッケル の理論と矛盾するものであった。そのため、発生の開始に精子の核が必要であるかどうかがしばらくの間議論の的となった。しかし、ヘルトヴィヒは両生類 や軟体動物 など、他の動物群を用いて自身の観察を確証した。エドゥアルト・シュトラスブルガー は、1884年に植物でも同様の結果を得た。ここから、遺伝において核が果たしている重要な役割の理解への道が開かれることとなった。1873年にアウグスト・ヴァイスマン は、遺伝に関しては母系と父系の生殖細胞 が等価であると予想した。遺伝情報を運搬するという核の機能は、有糸分裂 が発見され、20世紀の初めにメンデルの法則 が再発見されてはじめて明らかとなった。その後、遺伝の染色体説 が発展した[ 104]
^ a b c d e f g h i j k l m Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipursky SL, Darnell J (2004). Molecular Cell Biology ISBN 978-0-7167-2672-2 . https://archive.org/details/studentcompanion0000unse_r7k2
^ Ehrenhofer-Murray AE (June 2004). “Chromatin dynamics at DNA replication, transcription and repair”. European Journal of Biochemistry 271 (12): 2335–49. doi :10.1111/j.1432-1033.2004.04162.x . PMID 15182349 . ^ Grigoryev SA, Bulynko YA, Popova EY (2006). “The end adjusts the means: heterochromatin remodelling during terminal cell differentiation”. Chromosome Research 14 (1): 53–69. doi :10.1007/s10577-005-1021-6 . PMID 16506096 . ^ Schardin M, Cremer T, Hager HD, Lang M (December 1985). “Specific staining of human chromosomes in Chinese hamster x man hybrid cell lines demonstrates interphase chromosome territories” . Human Genetics 71 (4): 281–7. doi :10.1007/BF00388452 . PMID 2416668 . https://epub.ub.uni-muenchen.de/9272/1/cremer_thomas_9272.pdf . ^ Lamond AI, Earnshaw WC (April 1998). “Structure and function in the nucleus” . Science 280 (5363): 547–53. doi :10.1126/science.280.5363.547 . PMID 9554838 . http://azolla.fc.ul.pt/aulas/BiologiaCelular/docs/nucleo.pdf . ^ Kurz A, Lampel S, Nickolenko JE, Bradl J, Benner A, Zirbel RM, Cremer T, Lichter P (December 1996). “Active and inactive genes localize preferentially in the periphery of chromosome territories” . The Journal of Cell Biology 135 (5): 1195–205. doi :10.1083/jcb.135.5.1195 . PMC 2121085 . PMID 8947544 . オリジナル の29 September 2007時点におけるアーカイブ。. https://web.archive.org/web/20070929104104/http://intl.jcb.org/cgi/content/abstract/135/5/1195 . ^ Rothfield NF, Stollar BD (November 1967). “The relation of immunoglobulin class, pattern of anti-nuclear antibody, and complement-fixing antibodies to DNA in sera from patients with systemic lupus erythematosus” . The Journal of Clinical Investigation 46 (11): 1785–94. doi :10.1172/JCI105669 . PMC 292929 . PMID 4168731 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC292929/ . ^ Barned S, Goodman AD, Mattson DH (February 1995). “Frequency of anti-nuclear antibodies in multiple sclerosis”. Neurology 45 (2): 384–5. doi :10.1212/WNL.45.2.384 . PMID 7854544 . ^ Kume K, Cantwell H, Neumann FR, Jones AW, Snijders AP, Nurse P (May 2017). “A systematic genomic screen implicates nucleocytoplasmic transport and membrane growth in nuclear size control” . PLOS Genet 13 (5): e1006767. doi :10.1371/journal.pgen.1006767 . PMC 5436639 . PMID 28545058 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5436639/ . ^ a b c d e f g h i j Alberts B, Johnson A, Lewis J, Morgan D, Raff M, Roberts K, Walter P (2015). Molecular Biology of the Cell (6 ed.). New York: Garland Science
^ a b c Lodish HF, Berk A, Kaiser C, Krieger M, Bretscher A, Ploegh H, Amon A, Martin KC, Darnell JE (2016). Molecular Cell Biology (Eighth ed.). New York: W.H. Freeman. ISBN 978-1-4641-8339-3
^ Shulga N, Mosammaparast N, Wozniak R, Goldfarb DS (May 2000). “Yeast nucleoporins involved in passive nuclear envelope permeability” . The Journal of Cell Biology 149 (5): 1027–38. doi :10.1083/jcb.149.5.1027 . PMC 2174828 . PMID 10831607 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2174828/ . ^ Alberts, Bruce (2019). Essential cell biology (Fifth ed.). New York. p. 242. ISBN 9780393680393 ^ a b Pemberton LF, Paschal BM (March 2005). “Mechanisms of receptor-mediated nuclear import and nuclear export”. Traffic 6 (3): 187–98. doi :10.1111/j.1600-0854.2005.00270.x . PMID 15702987 .
^ a b c “Chapter 4: DNA and Chromosomes”. Molecular Biology of the Cell (4th ed.). New York: Garland Science. (2002). pp. 191–234. ISBN 978-0-8153-4072-0
^ Stuurman N, Heins S, Aebi U (1998). “Nuclear lamins: their structure, assembly, and interactions”. Journal of Structural Biology 122 (1–2): 42–66. doi :10.1006/jsbi.1998.3987 . PMID 9724605 . ^ Goldman AE, Moir RD, Montag-Lowy M, Stewart M, Goldman RD (November 1992). “Pathway of incorporation of microinjected lamin A into the nuclear envelope” . The Journal of Cell Biology 119 (4): 725–35. doi :10.1083/jcb.119.4.725 . PMC 2289687 . PMID 1429833 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2289687/ . ^ Wehnert, Manfred S.; Nguyen, Thuy Duong; Meinke, Peter (2011-12-01). “The LINC complex and human disease” (英語). Biochemical Society Transactions 39 (6): 1693–1697. doi :10.1042/BST20110658 . ISSN 1470-8752 . PMID 22103509 . http://www.biochemsoctrans.org/content/39/6/1693 . ^ a b c d e Goldman RD, Gruenbaum Y, Moir RD, Shumaker DK, Spann TP (March 2002). “Nuclear lamins: building blocks of nuclear architecture”. Genes & Development 16 (5): 533–47. doi :10.1101/gad.960502 . PMID 11877373 .
^ Broers JL, Ramaekers FC (2004). “Dynamics of nuclear lamina assembly and disassembly” . Symposia of the Society for Experimental Biology (56): 177–92. ISBN 9781134279838 . PMID 15565881 . https://books.google.com/books?id=lpR5AgAAQBAJ&pg=PA189 . ^ Moir RD, Yoon M, Khuon S, Goldman RD (December 2000). “Nuclear lamins A and B1: different pathways of assembly during nuclear envelope formation in living cells” . The Journal of Cell Biology 151 (6): 1155–68. doi :10.1083/jcb.151.6.1155 . PMC 2190592 . PMID 11121432 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2190592/ . ^ Spann TP, Goldman AE, Wang C, Huang S, Goldman RD (February 2002). “Alteration of nuclear lamin organization inhibits RNA polymerase II-dependent transcription” . The Journal of Cell Biology 156 (4): 603–8. doi :10.1083/jcb.200112047 . PMC 2174089 . PMID 11854306 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2174089/ . ^ Mounkes LC, Stewart CL (June 2004). “Aging and nuclear organization: lamins and progeria” . Current Opinion in Cell Biology 16 (3): 322–7. doi :10.1016/j.ceb.2004.03.009 . PMID 15145358 . https://zenodo.org/record/1258830 . ^ Hernandez-Verdun D (January 2006). “Nucleolus: from structure to dynamics” . Histochemistry and Cell Biology 125 (1–2): 127–37. doi :10.1007/s00418-005-0046-4 . PMID 16328431 . https://hal.archives-ouvertes.fr/hal-00015455 . ^ a b Lamond AI, Sleeman JE (October 2003). “Nuclear substructure and dynamics”. Current Biology 13 (21): R825-8. Bibcode : 2003CBio...13.R825L . doi :10.1016/j.cub.2003.10.012 . PMID 14588256 .
^
Spector DL, Lamond AI (Feb 2011). “Nuclear speckles” . Cold Spring Harbor Perspectives in Biology 3 (2): a000646. doi :10.1101/cshperspect.a000646 . PMC 3039535 . PMID 20926517 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3039535/ .
^
Alexander KA, Coté A, Nguyen SC, Zhang L, Berger SL (Mar 2021). “p53 mediates target gene association with nuclear speckles for amplified RNA expression” . Molecular Cell 81 (8): S1097-2765(21)00174-X. doi :10.1016/j.molcel.2021.03.006 . PMC 8830378 . PMID 33823140 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC8830378/ .
^ Lamond AI, Spector DL (August 2003). “Nuclear speckles: a model for nuclear organelles”. Nature Reviews. Molecular Cell Biology 4 (8): 605–12. doi :10.1038/nrm1172 . PMID 12923522 . ^ Tripathi K, Parnaik VK (September 2008). “Differential dynamics of splicing factor SC35 during the cell cycle” . Journal of Biosciences 33 (3): 345–54. doi :10.1007/s12038-008-0054-3 . PMID 19005234 . オリジナル の15 November 2011時点におけるアーカイブ。. https://web.archive.org/web/20111115235056/http://www.ias.ac.in/jbiosci/sep2008/345.pdf . ^ Tripathi K, Parnaik VK (September 2008). “Differential dynamics of splicing factor SC35 during the cell cycle”. Journal of Biosciences 33 (3): 345–54. doi :10.1007/s12038-008-0054-3 . PMID 19005234 . ^ Handwerger KE, Gall JG (January 2006). “Subnuclear organelles: new insights into form and function”. Trends in Cell Biology 16 (1): 19–26. doi :10.1016/j.tcb.2005.11.005 . PMID 16325406 . ^ “Cellular component Nucleus speckle ”. UniProt: UniProtKB. 2013年8月30日閲覧。 ^
Gall JG, Bellini M, Wu Z, Murphy C (December 1999). “Assembly of the nuclear transcription and processing machinery: Cajal bodies (coiled bodies) and transcriptosomes” . Molecular Biology of the Cell 10 (12): 4385–402. doi :10.1091/mbc.10.12.4385 . PMC 25765 . PMID 10588665 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC25765/ .
^ a b Matera AG, Terns RM, Terns MP (March 2007). “Non-coding RNAs: lessons from the small nuclear and small nucleolar RNAs”. Nature Reviews. Molecular Cell Biology 8 (3): 209–20. doi :10.1038/nrm2124 . PMID 17318225 .
^ Bhat P, Chow A, Emert B et al (May 2024). “Genome organization around nuclear speckles drives mRNA splicing efficiency.” . Nature 629 (5): 1165–1173. Bibcode : 2024Natur.629.1165B . doi :10.1038/s41586-024-07429-6 . PMC 11164319 . PMID 38720076 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC11164319/ . ^ a b c Cioce M, Lamond AI (2005). “Cajal bodies: a long history of discovery”. Annual Review of Cell and Developmental Biology 21 : 105–31. doi :10.1146/annurev.cellbio.20.010403.103738 . PMID 16212489 .
^ a b c Pollard, Thomas D.; Earnshaw, William C. (2004). Cell Biology ISBN 978-0-7216-3360-2 . https://archive.org/details/cellbiology0000poll
^ a b c Matera AG, Frey MR (August 1998). “Coiled bodies and gems: Janus or gemini?” . American Journal of Human Genetics 63 (2): 317–21. doi :10.1086/301992 . PMC 1377332 . PMID 9683623 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1377332/ .
^ a b Matera AG (August 1998). “Of coiled bodies, gems, and salmon”. Journal of Cellular Biochemistry 70 (2): 181–92. doi :10.1002/(sici)1097-4644(19980801)70:2<181::aid-jcb4>3.0.co;2-k . PMID 9671224 .
^ Navascues J, Berciano MT, Tucker KE, Lafarga M, Matera AG (June 2004). “Targeting SMN to Cajal bodies and nuclear gems during neuritogenesis” . Chromosoma 112 (8): 398–409. doi :10.1007/s00412-004-0285-5 . PMC 1592132 . PMID 15164213 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1592132/ . ^ a b c d Lafarga M, Berciano MT, Pena E, Mayo I, Castaño JG, Bohmann D, Rodrigues JP, Tavanez JP, Carmo-Fonseca M (August 2002). “Clastosome: a subtype of nuclear body enriched in 19S and 20S proteasomes, ubiquitin, and protein substrates of proteasome” . Molecular Biology of the Cell 13 (8): 2771–82. doi :10.1091/mbc.e02-03-0122 . PMC 117941 . PMID 12181345 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC117941/ .
^ a b c Dundr M, Misteli T (June 2001). “Functional architecture in the cell nucleus” . The Biochemical Journal 356 (Pt 2): 297–310. doi :10.1042/0264-6021:3560297 . PMC 1221839 . PMID 11368755 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1221839/ .
^ Bond CS, Fox AH (September 2009). “Paraspeckles: nuclear bodies built on long noncoding RNA” . The Journal of Cell Biology 186 (5): 637–44. doi :10.1083/jcb.200906113 . PMC 2742191 . PMID 19720872 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2742191/ . ^ Goebel HH, Warlo I (January 1997). “Nemaline myopathy with intranuclear rods--intranuclear rod myopathy”. Neuromuscular Disorders 7 (1): 13–9. doi :10.1016/S0960-8966(96)00404-X . PMID 9132135 . ^ Saunders WS, Cooke CA, Earnshaw WC (November 1991). “Compartmentalization within the nucleus: discovery of a novel subnuclear region” . The Journal of Cell Biology 115 (4): 919–31. doi :10.1083/jcb.115.4.919 . PMC 2289954 . PMID 1955462 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2289954/ . ^ Pombo A, Cuello P, Schul W, Yoon JB, Roeder RG, Cook PR, Murphy S (March 1998). “Regional and temporal specialization in the nucleus: a transcriptionally-active nuclear domain rich in PTF, Oct1 and PIKA antigens associates with specific chromosomes early in the cell cycle” . The EMBO Journal 17 (6): 1768–78. doi :10.1093/emboj/17.6.1768 . PMC 1170524 . PMID 9501098 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1170524/ . ^ Zimber A, Nguyen QD, Gespach C (October 2004). “Nuclear bodies and compartments: functional roles and cellular signalling in health and disease”. Cellular Signalling 16 (10): 1085–104. doi :10.1016/j.cellsig.2004.03.020 . PMID 15240004 . ^ Lallemand-Breitenbach V, de Thé H (May 2010). “PML nuclear bodies” . Cold Spring Harbor Perspectives in Biology 2 (5): a000661. doi :10.1101/cshperspect.a000661 . PMC 2857171 . PMID 20452955 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2857171/ . ^ Fox AH, Lamond AI (July 2010). “Paraspeckles” . Cold Spring Harbor Perspectives in Biology 2 (7): a000687. doi :10.1101/cshperspect.a000687 . PMC 2890200 . PMID 20573717 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2890200/ . ^ a b “Nuclear Compartments: Paraspeckles ”. Nuclear Protein Database (2004年). 2008年9月10日時点のオリジナル よりアーカイブ。2007年3月6日閲覧。
^ a b Fox AH, Bond CS, Lamond AI (November 2005). “P54nrb forms a heterodimer with PSP1 that localizes to paraspeckles in an RNA-dependent manner” . Molecular Biology of the Cell 16 (11): 5304–15. doi :10.1091/mbc.E05-06-0587 . PMC 1266428 . PMID 16148043 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1266428/ .
^ Nakagawa S, Yamazaki T, Hirose T (October 2018). “Molecular dissection of nuclear paraspeckles: towards understanding the emerging world of the RNP milieu” . Open Biology 8 (10): 180150. doi :10.1098/rsob.180150 . PMC 6223218 . PMID 30355755 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6223218/ . ^ Pisani G, Baron B (December 2019). “Nuclear paraspeckles function in mediating gene regulatory and apoptotic pathways” . Non-Coding RNA Research 4 (4): 128–134. doi :10.1016/j.ncrna.2019.11.002 . PMC 7012776 . PMID 32072080 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7012776/ . ^ Fox, Archa; Lam, YW; Leung, AK; Lyon, CE; Andersen, J; Mann, M; Lamond, AI (2002). “Paraspeckles: A Novel Nuclear Domain”. Current Biology 12 (1): 13–25. doi :10.1016/S0960-9822(01)00632-7 . PMID 11790299 . ^ Kong XN, Yan HX, Chen L, Dong LW, Yang W, Liu Q, Yu LX, Huang DD, Liu SQ, Liu H, Wu MC, Wang HY (October 2007). “LPS-induced down-regulation of signal regulatory protein {alpha} contributes to innate immune activation in macrophages” . The Journal of Experimental Medicine 204 (11): 2719–31. doi :10.1084/jem.20062611 . PMC 2118489 . PMID 17954568 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2118489/ . ^ a b Carmo-Fonseca M, Berciano MT, Lafarga M (September 2010). “Orphan nuclear bodies” . Cold Spring Harbor Perspectives in Biology 2 (9): a000703. doi :10.1101/cshperspect.a000703 . PMC 2926751 . PMID 20610547 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2926751/ .
^ Sampuda KM, Riley M, Boyd L (April 2017). “Stress induced nuclear granules form in response to accumulation of misfolded proteins in Caenorhabditis elegans” . BMC Cell Biology 18 (1): 18. doi :10.1186/s12860-017-0136-x . PMC 5395811 . PMID 28424053 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5395811/ . ^ Lehninger, Albert L.; Nelson, David L.; Cox, Michael M. (2000). Lehninger principles of biochemistry ISBN 978-1-57259-931-4 . https://archive.org/details/lehningerprincip01lehn ^ Moreno F, Ahuatzi D, Riera A, Palomino CA, Herrero P (February 2005). “Glucose sensing through the Hxk2-dependent signalling pathway”. Biochemical Society Transactions 33 (Pt 1): 265–8. doi :10.1042/BST0330265 . PMID 15667322 . ^ Görlich D, Kutay U (1999). “Transport between the cell nucleus and the cytoplasm”. Annual Review of Cell and Developmental Biology 15 (1): 607–60. doi :10.1146/annurev.cellbio.15.1.607 . PMID 10611974 . ^ Hozák P, Cook PR (February 1994). “Replication factories”. Trends in Cell Biology 4 (2): 48–52. doi :10.1016/0962-8924(94)90009-4 . PMID 14731866 . ^ Nierhaus, Knud H.; Wilson, Daniel N. (2004). Protein Synthesis and Ribosome Structure: Translating the Genome . Wiley-VCH. ISBN 978-3-527-30638-1 ^ Nicolini, Claudio A. (1997). Genome Structure and Function: From Chromosomes Characterization to Genes Technology . Springer. ISBN 978-0-7923-4565-7 ^ Black DL (2003). “Mechanisms of alternative pre-messenger RNA splicing” . Annual Review of Biochemistry 72 (1): 291–336. doi :10.1146/annurev.biochem.72.121801.161720 . PMID 12626338 . https://cloudfront.escholarship.org/dist/prd/content/qt2hg605wm/qt2hg605wm.pdf . ^ Watson JD, Baker TA, Bell SP, Gann A, Levine M, Losick R (2004). “Ch9–10”. Molecular Biology of the Gene (5th ed.). Peason Benjamin Cummings; CSHL Press.. ISBN 978-0-8053-9603-4 ^ Cavazza T, Vernos I (2015). “The RanGTP Pathway: From Nucleo-Cytoplasmic Transport to Spindle Assembly and Beyond” . Frontiers in Cell and Developmental Biology 3 : 82. doi :10.3389/fcell.2015.00082 . PMC 4707252 . PMID 26793706 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4707252/ . ^ Lippincott-Schwartz J (March 2002). “Cell biology: ripping up the nuclear envelope” . Nature 416 (6876): 31–2. Bibcode : 2002Natur.416...31L . doi :10.1038/416031a . PMID 11882878 . https://zenodo.org/record/1233215 . ^ a b Boulikas T (1995). “Phosphorylation of transcription factors and control of the cell cycle”. Critical Reviews in Eukaryotic Gene Expression 5 (1): 1–77. PMID 7549180 .
^ Boettcher B, Barral Y (2013). “The cell biology of open and closed mitosis” . Nucleus (Austin, Tex.) 4 (3): 160–5. doi :10.4161/nucl.24676 . PMC 3720745 . PMID 23644379 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3720745/ . ^ Steen RL, Collas P (April 2001). “Mistargeting of B-type lamins at the end of mitosis: implications on cell survival and regulation of lamins A/C expression” . The Journal of Cell Biology 153 (3): 621–6. doi :10.1083/jcb.153.3.621 . PMC 2190567 . PMID 11331311 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2190567/ . ^ Böhm I (November 2007). “IgG deposits can be detected in cell nuclei of patients with both lupus erythematosus and malignancy”. Clinical Rheumatology 26 (11): 1877–82. doi :10.1007/s10067-007-0597-y . PMID 17364135 . ^ Ressel, Lorenzo (2017). “Nuclear Morphologies” . Normal cell morphology in canine and feline cytology: an identification guide . Hoboken, NJ: John Wiley & Sons. p. 6. ISBN 978-1-119-27891-7 . https://books.google.com/books?id=AkwrDwAAQBAJ&pg=PA6 ^ Skutelsky E, Danon D (June 1970). “Comparative study of nuclear expulsion from the late erythroblast and cytokinesis”. Experimental Cell Research 60 (3): 427–36. doi :10.1016/0014-4827(70)90536-7 . PMID 5422968 . ^ Torous DK, Dertinger SD, Hall NE, Tometsko CR (February 2000). “Enumeration of micronucleated reticulocytes in rat peripheral blood: a flow cytometric study”. Mutation Research 465 (1–2): 91–9. Bibcode : 2000MRGTE.465...91T . doi :10.1016/S1383-5718(99)00216-8 . PMID 10708974 . ^ Hutter KJ, Stöhr M (1982). “Rapid detection of mutagen induced micronucleated erythrocytes by flow cytometry”. Histochemistry 75 (3): 353–62. doi :10.1007/bf00496738 . PMID 7141888 . ^ Ham BK, Lucas WJ (April 2014). “The angiosperm phloem sieve tube system: a role in mediating traits important to modern agriculture”. Journal of Experimental Botany 65 (7): 1799–816. doi :10.1093/jxb/ert417 . PMID 24368503 . ^ Zettler LA, Sogin ML, Caron DA (October 1997). “Phylogenetic relationships between the Acantharea and the Polycystinea: a molecular perspective on Haeckel's Radiolaria” . Proceedings of the National Academy of Sciences of the United States of America 94 (21): 11411–6. Bibcode : 1997PNAS...9411411A . doi :10.1073/pnas.94.21.11411 . PMC 23483 . PMID 9326623 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC23483/ . ^ Horton TR (2006). “The number of nuclei in basidiospores of 63 species of ectomycorrhizal Homobasidiomycetes”. Mycologia 98 (2): 233–8. doi :10.3852/mycologia.98.2.233 . PMID 16894968 . ^ Adam RD (December 1991). “The biology of Giardia spp” . Microbiological Reviews 55 (4): 706–32. doi :10.1128/MMBR.55.4.706-732.1991 . PMC 372844 . PMID 1779932 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC372844/ . ^ Vogt A, Goldman AD, Mochizuki K, Landweber LF (1 August 2013). “Transposon Domestication versus Mutualism in Ciliate Genome Rearrangements” . PLOS Genetics 9 (8): e1003659. doi :10.1371/journal.pgen.1003659 . PMC 3731211 . PMID 23935529 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3731211/ . ^ McInnes A, Rennick DM (February 1988). “Interleukin 4 induces cultured monocytes/macrophages to form giant multinucleated cells” . The Journal of Experimental Medicine 167 (2): 598–611. doi :10.1084/jem.167.2.598 . PMC 2188835 . PMID 3258008 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2188835/ . ^ Goldring SR, Roelke MS, Petrison KK, Bhan AK (February 1987). “Human giant cell tumors of bone identification and characterization of cell types” . The Journal of Clinical Investigation 79 (2): 483–91. doi :10.1172/JCI112838 . PMC 424109 . PMID 3027126 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC424109/ . ^ Imanian B, Pombert JF, Dorrell RG, Burki F, Keeling PJ (2012). “Tertiary endosymbiosis in two dinotoms has generated little change in the mitochondrial genomes of their dinoflagellate hosts and diatom endosymbionts” . PLOS ONE 7 (8): e43763. Bibcode : 2012PLoSO...743763I . doi :10.1371/journal.pone.0043763 . PMC 3423374 . PMID 22916303 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3423374/ . ^ Pennisi E (August 2004). “Evolutionary biology. The birth of the nucleus”. Science 305 (5685): 766–8. doi :10.1126/science.305.5685.766 . PMID 15297641 . ^ Devos DP, Gräf R, Field MC (June 2014). “Evolution of the nucleus” . Current Opinion in Cell Biology 28 (100): 8–15. doi :10.1016/j.ceb.2014.01.004 . PMC 4071446 . PMID 24508984 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4071446/ . ^ López-García P, Moreira D (November 2015). “Open Questions on the Origin of Eukaryotes” . Trends in Ecology & Evolution 30 (11): 697–708. Bibcode : 2015TEcoE..30..697L . doi :10.1016/j.tree.2015.09.005 . PMC 4640172 . PMID 26455774 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4640172/ . ^ Hogan CM (2010). “Archaea” . Encyclopedia of Earth . http://www.eoearth.org/article/Archaea?topic=49496 ^ Margulis, Lynn (1981). Symbiosis in Cell Evolution 206–227 . ISBN 978-0-7167-1256-5 . https://archive.org/details/symbiosisincelle00marg/page/206 ^ Martin W (December 2005). “Archaebacteria (Archaea) and the origin of the eukaryotic nucleus”. Curr Opin Microbiol 8 (6): 630–7. doi :10.1016/j.mib.2005.10.004 . PMID 16242992 . ^ Bernstein, Harris; Bernstein, Carol (2017), Witzany, Guenther, ed. (英語), Sexual Communication in Archaea, the Precursor to Eukaryotic Meiosis doi :10.1007/978-3-319-65536-9_7 , ISBN 978-3-319-65535-2 , http://link.springer.com/10.1007/978-3-319-65536-9_7 ^ López-García P, Moreira D (May 2006). “Selective forces for the origin of the eukaryotic nucleus”. BioEssays 28 (5): 525–33. doi :10.1002/bies.20413 . PMID 16615090 . ^ Fuerst JA (2005). “Intracellular compartmentation in planctomycetes”. Annual Review of Microbiology 59 : 299–328. doi :10.1146/annurev.micro.59.030804.121258 . PMID 15910279 . ^ Hartman H, Fedorov A (February 2002). “The origin of the eukaryotic cell: a genomic investigation” . Proceedings of the National Academy of Sciences of the United States of America 99 (3): 1420–5. Bibcode : 2002PNAS...99.1420H . doi :10.1073/pnas.032658599 . PMC 122206 . PMID 11805300 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC122206/ . ^ Bell PJ (September 2001). “Viral eukaryogenesis: was the ancestor of the nucleus a complex DNA virus?”. Journal of Molecular Evolution 53 (3): 251–6. Bibcode : 2001JMolE..53..251L . doi :10.1007/s002390010215 . PMID 11523012 . ^ Takemura M (May 2001). “Poxviruses and the origin of the eukaryotic nucleus”. Journal of Molecular Evolution 52 (5): 419–25. Bibcode : 2001JMolE..52..419T . doi :10.1007/s002390010171 . PMID 11443345 . ^ Villarreal LP, DeFilippis VR (August 2000). “A hypothesis for DNA viruses as the origin of eukaryotic replication proteins” . Journal of Virology 74 (15): 7079–84. doi :10.1128/JVI.74.15.7079-7084.2000 . PMC 112226 . PMID 10888648 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC112226/ . ^ Bell PJ (November 2006). “Sex and the eukaryotic cell cycle is consistent with a viral ancestry for the eukaryotic nucleus”. Journal of Theoretical Biology 243 (1): 54–63. Bibcode : 2006JThBi.243...54B . doi :10.1016/j.jtbi.2006.05.015 . PMID 16846615 . ^ de Roos AD (2006). “The origin of the eukaryotic cell based on conservation of existing interfaces”. Artificial Life 12 (4): 513–23. doi :10.1162/artl.2006.12.4.513 . PMID 16953783 . ^ Van Leeuwenhoek A (ラテン語). Opera Omnia, seu Arcana Naturae ope exactissimorum Microscopiorum detecta, experimentis variis comprobata, Epistolis ad varios illustres viros J. Arnold et Delphis, A. Beman, Lugdinum Batavorum [The Works of, or arcana of nature by means of exactissimorum microscopes had been detected and confirmed by a variety of experiments, the Epistles to the various illustrious men of valor J. Arnold and Delphi, A. Beman, Lugdina York 1719-1730] Gerlach D (2009). Geschichte der Mikroskopie . Frankfurt am Main, Germany: Verlag Harri Deutsch. ISBN 978-3-8171-1781-9 ^ Cohen WD (1982). “The cytomorphic system of anucleate non-mammalian erythrocytes”. Protoplasma 113 (1): 23–32. Bibcode : 1982Prpls.113...23C . doi :10.1007/BF01283036 . ^ Harris H (1999). The Birth of the Cell ISBN 978-0-300-07384-3 . https://archive.org/details/birthofcell0000harr ^ Brown, Robert (2015-04-02). Bennett, John Joseph; Brown, Robert. eds. “On the Organs and Mode of Fecundation of Orchidex and Asclepiadea” . Miscellaneous Botanical Works I . Cambridge Library Collection - Botany and Horticulture (Cambridge: Cambridge University Press) 1 : 511–514. doi :10.1017/cbo9781107775473.017 . ISBN 978-1-108-07681-4 . https://www.cambridge.org/core/product/identifier/CBO9781107775473A024/type/book_part . ^ Cremer, T. (1986). Von der Zellenlehre Zur Chromosomentheorie: Naturwissenschaftliche Erkenntnis und Theorienwechsel in der Frühen Zell- und Vererbungsforschung . Berlin, Heidelberg: Springer Berlin / Heidelberg. ISBN 978-3-540-13987-4 ^ Cremer, Thomas (1985). Von der Zellenlehre zur Chromosomentheorie . Berlin, Heidelberg, New York, Tokyo: Springer Verlag. ISBN 978-3-540-13987-4 here
Goldman, Robert D.; Gruenbaum, Y; Moir, RD; Shumaker, DK; Spann, TP (2002). “Nuclear lamins: building blocks of nuclear architecture”. Genes Dev. 16 (5): 533–547. doi :10.1101/gad.960502 . PMID 11877373 . 核のラミンについての総説。その構造とさまざまな機能について説明している。 核輸送についての総説。メカニズムの原理とさまざまな輸送経路について説明している。 核についての総説。染色体の構造、核小体や他の核内構造体について記述している。 核の進化についての総説。さまざまな理論について説明している。 Pollard, Thomas D.; William C. Earnshaw (2004). Cell Biology . Philadelphia: Saunders. ISBN 0-7216-3360-9 細胞生物学に焦点を絞った大学レベルの教科書。核の構造と機能、核輸送、核内ドメインについての情報が含まれる。
ウィキメディア・コモンズには、
細胞核 に関連するカテゴリがあります。