クロマチンリモデリング (英 : chromatin remodeling )は、クロマチン 構造の動的な調節である。クロマチンリモデリングは凝縮したゲノム DNA に対する転写 調節装置のタンパク質 のアクセスを可能にし、遺伝子発現 の制御が行われる。こうしたリモデリングは主に、(1) 特異的酵素 による共有結合的なヒストン 修飾(ヒストンアセチル化酵素 、脱アセチル化酵素 、メチル化酵素 、キナーゼ などによるもの)、(2) ヌクレオソーム を動かしたり、除去したり、再構築したりするATP 依存的なクロマチン構造のリモデリング、によって行われる[ 1] 卵細胞 のDNA複製 や修復 、アポトーシス 、染色体分離 (英語版 ) 多能性 など、いくつかの重要な生物学的過程のエピジェネティック な調節を可能にする。クロマチンリモデリングタンパク質の異常は、がん を含むヒトの疾患と関係していることが判明している。いくつかのがんに対しては、クロマチンリモデリング経路を標的とした治療戦略の進化が続いている。
ゲノムの転写調節は主に転写開始前の段階、DNAのコアプロモーター 配列へのコア転写装置(すなわちRNAポリメラーゼ 、転写因子 、アクチベーター とリプレッサー )の結合の制御によって行われている。しかし、核内のDNAはしっかりとパッケージングされており、主にヒストンタンパク質の助けによってヌクレオソームの反復単位が形成され、それらがさらに束ねられて凝縮したクロマチン構造が形成されている。こうした凝縮構造は多くのDNA調節タンパク質を排除し、転写装置との相互作用や遺伝子発現の調節はできない状態となっている。クロマチンリモデリングと呼ばれる過程は、この問題を克服して凝縮DNAに対して動的なアクセスを行うことを目的として行われ、ヌクレオソーム構造を変化させて転写調節のためのDNA領域を露出させたり隠したりする。
定義として、クロマチンリモデリングはヌクレオソームDNAへのアクセスを促進する酵素的過程であり、ヌクレオソームの構造、構成、配置のリモデリングが行われる。
ヌクレオソームDNAへのアクセスは大きく2種類のタンパク質複合体によって行われる。
共有結合によってヒストンを修飾する複合体
ATP依存的なクロマチンリモデリング複合体
ヒストン修飾複合体と呼ばれる特異的なタンパク質複合体が、ヒストンに対するさまざまな化学的要素の付加や除去を触媒する。こうした酵素的な修飾にはアセチル化 、メチル化 、リン酸化 、ユビキチン化 が含まれ、主に修飾はヒストンのN末端 のテール領域に対して行われる。こうした修飾はヒストンとDNAの間の結合親和性に影響を与え、ヒストンに巻き付いている凝縮したDNA構造を緩めたり引き締めたりする。例えば、ヒストンH3 とH4 の特定のリジン 残基のメチル化はヒストン周囲のDNAのさらなる凝縮を引き起こし、転写因子のDNAへの結合を阻害し遺伝子発現を抑制する。反対に、ヒストンのアセチル化はクロマチンの凝縮を緩め、転写因子が結合できるようにDNAを露出させ、遺伝子発現を増加させる[ 2]
よく知られたヒストン修飾には次のようなものがある[ 3]
リジン残基とアルギニン 残基の双方がメチル化を受けることが知られている。メチル化リジンはヒストンコードの中で最もよく理解されている標識の1つであり、特定のリジン残基のメチル化状態は遺伝子の発現状態とよく一致する。H3K4とH3K36のメチル化は転写の活性化と相関している一方、H3K4の脱メチル化はゲノム領域のサイレンシングと相関している。H3K9とH3K27のメチル化は転写抑制と相関している[ 4] ヘテロクロマチン と高度の相関がみられる[ 5]
アセチル化されたヒストンは脱アセチル化ヒストンと同じようにうまくパッキングすることはできないため、クロマチンが「開いた」構造となる傾向がある。
ヒストン修飾にはさらに多くの種類が存在し、高感度の質量分析 によって近年その種類は大きく広がった[ 6]
ヒストンコード (英語版 ) DNAのメチル化 などの類似した修飾とともに、エピジェネティックコード (英語版 )
多くの研究の蓄積により、こうしたコードはヒストンをメチル化したりアセチル化したりする特定の酵素によって書き込まれ(ライター)、脱メチル化や脱アセチル化活性を持つ他の酵素によって消去され(イレーザー)、そして最終的に特定のドメイン(ブロモドメイン 、クロモドメイン など)を介してこうした修飾へリクルートされて結合するタンパク質によって読み取られる(リーダー)ことが示唆されている。これらライター、イレーザー、リーダーによる3つの作用によって、転写調節やDNA損傷修復などに適した局所的環境が確立される[ 7]
ヒストンコード仮説の重要なコンセプトは、ヒストン修飾は単にヒストンとDNAの相互作用を安定化したり不安定化したりするのではなく、専用のタンパク質ドメインによって修飾を特異的に認識する他のタンパク質をリクルートするために利用される、という点である。こうしてリクルートされたタンパク質はその後、クロマチン構造を活発に変化させたり、転写を促進したりする。
遺伝子発現に関するヒストンコードの非常に基礎的な概要を下に示す。
ATP依存性クロマチンリモデリング複合体は、ヌクレオソームを移動させるか、除去するか、再構築するかによって遺伝子発現を調節する。これらのタンパク質複合体は共通したATPアーゼ ドメインを持っており、ATP の加水分解 によるエネルギーによってヌクレオソームをDNAに沿って再配置したり(ヌクレオソームスライディング(nucleosome sliding)とも呼ばれる)、ヒストンを組み立てたり除去したり、ヒストンバリアント の交換を促進したりし、遺伝子の活性化のためにヌクレオソームが存在しないDNA領域を作り出す[ 12] [ 13]
全てのATP依存性クロマチンリモデリング複合体には、SNF2スーパーファミリーに属するATPアーゼサブユニットが含まれている。これらのタンパク質には2つの主要なグループが存在しており、SWI2/SNF2(SWI/SNF)グループとISWI(imitation SWI)グループとして知られている。近年記載されたものの中には、脱アセチル化活性も示すものもある[ 14]
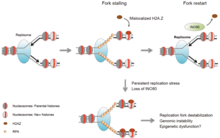
INO80は複製フォークを安定化し、誤って配置されたH2A.Zを除去する。 真核生物のクロマチンリモデリング因子には少なくとも5つのファミリー(SWI/SNF 、ISWI (英語版 ) Mi-2/NuRD (英語版 ) CHD 、INO80 、SWR1)が存在し、最初の2つに関しては特に酵母モデルでよく研究が行われている。リモデリング因子には共通したATPアーゼドメインが存在するが、それらはいくつかの生物学的過程(DNA修復、アポトーシスなど)において特異的に機能する。これは各リモデリング因子のATPアーゼ領域には特有のタンパク質ドメイン(ヘリカーゼ 、ブロモドメインなど)が存在し、また異なるサブユニットを介してリクルートが行われるためである。
いくつかのin vitro
ISWIファミリーのリモデリング因子はDNA複製後のクロマチンの組み立てとクロマチンの高次構造の維持に中心的な役割を果たすことが示されている。
INO80ファミリーとSWI/SNFファミリーのリモデリング因子はDNA二本鎖切断修復とヌクレオチド除去修復 に関与し、p53 を介したDNA損傷応答に重要な役割を果たす。
Mi-2/NuRD/CHDリモデリング複合体は主に核内での転写抑制を媒介し、胚性幹細胞 の多能性の維持に必要である[ 12]
クロマチンリモデリングはしっかりとパッケージされたゲノムに対して転写装置の動的なアクセスを可能にし、遺伝子発現調節において中心的な役割を果たしている。さらに、クロマチンリモデリング因子によるヌクレオソームの移動は、染色体の集合と分離、DNAの複製と修復、胚発生と多能性、細胞周期の進行など、いくつかの重要な生物学的過程に必要不可欠である。クロマチンリモデリングの調節異常によって、適切な細胞機能に必要とされるこうした重要なチェックポイントでの転写調節の喪失が引き起こされ、それによってがんを含むさまざまな疾患が引き起こされる。
クロマチン構造の緩和は、DNA損傷に対する最初期の細胞応答である[ 15] PARP1 によって開始されるようであり、PARP1のDNA損傷部位への蓄積はDNA損傷の発生後1.6秒以内に最大半値に達する[ 16] ADPリボース 結合ドメインを持つクロマチンリモデリング因子ALC1 がPARPの反応産物であるポリADPリボース鎖に迅速に結合する。ALC1のDNA損傷部位へのリクルートは損傷後10秒以内に最大値に達する[ 15] [ 15] MRE11 とNBS1 がリクルートされる。これら2つのDNA修復酵素のリクルートは、MRE11に関しては13秒、NBS1に関しては28秒で最大半値に達する[ 16]
DNA二本鎖切断の形成後のクロマチン構造の緩和の他の過程には、H2AX のリン酸化型であるγH2AXが関与している。ヒストンバリアントH2AXは、ヒトのクロマチン中のH2Aヒストンの約10%を構成している[ 17] ガンマ線 照射による二本鎖切断の形成後20秒で検出され、1分で最大半値に達する[ 17] bp にわたる[ 17]
γH2AXはそれ自身がクロマチンの脱凝縮を引き起こすわけではないが、照射後数秒以内にMDC1 がγH2AXに特異的に結合する[ 18] [ 19] RNF8 とNBS1が蓄積する。NBS1は、γH2AXに結合したMDC1に結合する[ 20] CHD4 (英語版 ) [ 21]
DNA損傷に伴う迅速なクロマチン構造の緩和とDNA修復の開始後にはゆっくりと再凝縮が行われ、約20分でクロマチンは損傷前の状態に近い凝縮状態を回復する[ 15]
クロマチンリモデリングは、細胞周期の進行、DNA修復や染色体分離など、細胞の成長と分裂の重要な段階の微調整を行い、それによって腫瘍形成を抑制する機能を発揮する。こうしたクロマチンリモデリング因子の変異やヒストン修飾の調節異常によって、細胞増殖が自己充足的となり、成長調節シグナルによる制御を受けない状態となる可能性がある。これらはがんの2つの重要な特徴である[ 22]
クロマチンリモデリングの調節異常によって引き起こされるエピジェネティックな不安定性について、乳がん 、大腸がん 、膵臓がん を含むいくつかのがんで研究が行われている。こうした不安定性は広範囲にわたる遺伝子のサイレンシングを引き起こし、主にがん抑制遺伝子に影響を与える。そのため、ヒストン脱アセチル化酵素阻害剤 (HDI)とDNA脱メチル化剤との相乗効果によってエピジェネティックなサイレンシングを克服する戦略がとられている。HDIはいくつかのタイプのがんで主に補助療法として利用されている[ 31] [ 32] p53 のがん抑制活性の調節因子であるp21 (WAF1)の発現を誘導する。ヒストン脱アセチル化酵素はRbタンパク質が細胞増殖を抑制する経路に関与している[ 33] エストロゲン はエストロゲン受容体α (ERα)に結合し、乳がんの形成と進行への関与することが示唆されている分裂促進因子 として良く知られている。近年のデータからは、ヒストン脱アセチル化とDNAメチル化によるクロマチン不活性化がヒトの乳がん細胞におけるERαのサイレンシングの重要な要素であることが示されている[ 34]
HDIとして、ボリノスタット とロミデプシン は、アメリカ食品医薬品局 (FDA)から皮膚T細胞性リンパ腫 (英語版 ) [ 35] [ 36] ベリノスタット (英語版 ) パノビノスタット (LBH589)は2015年2月に多発性骨髄腫 に対してFDAの迅速承認が行われている[ 37] [ 38] バルプロ酸 は子宮頸がんと乳がんに対する臨床試験が行われている[ 39] [ 40]
ヒストンリジンメチルトランスフェラーゼとアルギニンメチルトランスフェラーゼは新たな薬剤標的の有力な候補である[ 41]
クロマチン構造のリモデリングは細胞老化 の過程への関与が示唆されている。細胞老化は、個体レベルでの老化 と関連しているものの異なる過程である。複製老化は恒久的な細胞周期 の停止を意味し、有糸分裂 を終えた細胞は代謝的に活発な状態で存在し続けるが、増殖することはできない[ 43] [ 44] テロメア の短縮、プロジェリア 、前がん状態、他の損傷や疾患によっても生じる。老化細胞には明確に抑制的な表現型の変化が生じ、クロマチン構成の変化やリモデリング因子の存在量の変動、エピジェネティックな修飾の変化が生じた損傷細胞やがん性細胞の増殖を防いでいる可能性がある[ 43] [ 45] [ 46] ヘテロクロマチン が核の中心部に移動し、ユークロマチン と条件的ヘテロクロマチンが核の周縁部に移動するという、クロマチン配置の変化が生じる。これによってクロマチンとラミン の間の相互作用が破壊され、活発に有糸分裂を行う細胞で見られるパターンとは反転した配置となる[ 45] [ 47] トポロジカルドメイン (TAD)は破壊され、ゲノム間のシス の相互作用に影響が生じる[ 48] H3 とH4 、リンカーヒストンH1 が喪失する[ 47] [ 48] S期 にのみ合成されるため、有糸分裂を終えた老化細胞では発現しない[ 47] リソソーム での分解が行われ、より大きな構成の変化とクロマチン相互作用の破壊が引き起こされる[ 46]
クロマチンリモデリング因子の存在量は細胞老化に影響を与える可能性がある。酵母、C. elegans 、マウス、ヒトの培養細胞において、NuRD、ACF1、SWI/SNFなどのATP依存性リモデリング因子のノックダウン やノックアウト によってDNA損傷と老化表現型が生じる[ 46] [ 49] [ 50] [ 49] [ 50] p16 がPRC1/PRC2によってサイレンシングされているのはその1例である[ 51] [ 46] 遺伝子座 というよりはエンハンサー 領域に対して作用し、調節領域周辺に濃密なヘテロクロマチン領域を形成することで細胞周期の再進行を防いでいる[ 52]
^ “Predicting nucleosome positions on the DNA: combining intrinsic sequence preferences and remodeler activities” . Nucleic Acids Research 37 (17): 5641–55. (September 2009). doi :10.1093/nar/gkp610 . PMC 2761276 . PMID 19625488 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2761276/ . ^ “Chromatin remodeling and cancer, Part I: Covalent histone modifications”. Trends in Molecular Medicine 13 (9): 363–72. (September 2007). doi :10.1016/j.molmed.2007.07.003 . PMID 17822958 . ^ “The language of covalent histone modifications”. Nature 403 (6765): 41–5. (January 2000). Bibcode : 2000Natur.403...41S . doi :10.1038/47412 . PMID 10638745 . ^ a b c d “Determination of enriched histone modifications in non-genic portions of the human genome” . BMC Genomics 10 : 143. (March 2009). doi :10.1186/1471-2164-10-143 . PMC 2667539 . PMID 19335899 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2667539/ .
^ Hublitz, Philip; Albert, Mareike; Peters, Antoine (28 April 2009). “Mechanisms of Transcriptional Repression by Histone Lysine Methylation”. The International Journal of Developmental Biology 10 (1387): 335–354. ISSN 1696-3547 . ^ “Identification of 67 histone marks and histone lysine crotonylation as a new type of histone modification” . Cell 146 (6): 1016–28. (September 2011). doi :10.1016/j.cell.2011.08.008 . PMC 3176443 . PMID 21925322 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3176443/ . ^ “Translating the histone code”. Science 293 (5532): 1074–80. (August 2001). doi :10.1126/science.1063127 . PMID 11498575 . ^ “Histone H3K4 demethylases are essential in development and differentiation”. Biochemistry and Cell Biology 85 (4): 435–43. (August 2007). doi :10.1139/o07-057 . PMID 17713579 . ^ a b c d e f g h “High-resolution profiling of histone methylations in the human genome”. Cell 129 (4): 823–37. (May 2007). doi :10.1016/j.cell.2007.05.009 . PMID 17512414 .
^ a b c “DOT1L/KMT4 recruitment and H3K79 methylation are ubiquitously coupled with gene transcription in mammalian cells” . Molecular and Cellular Biology 28 (8): 2825–39. (April 2008). doi :10.1128/MCB.02076-07 . PMC 2293113 . PMID 18285465 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2293113/ .
^ a b c “The landscape of histone modifications across 1% of the human genome in five human cell lines” . Genome Research 17 (6): 691–707. (June 2007). doi :10.1101/gr.5704207 . PMC 1891331 . PMID 17567990 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1891331/ .
^ a b “Chromatin remodeling and cancer, Part II: ATP-dependent chromatin remodeling” . Trends in Molecular Medicine 13 (9): 373–80. (September 2007). doi :10.1016/j.molmed.2007.07.004 . PMC 4337864 . PMID 17822959 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4337864/ .
^ “Chromatin remodelling: the industrial revolution of DNA around histones”. Nature Reviews Molecular Cell Biology 7 (6): 437–47. (June 2006). doi :10.1038/nrm1945 . PMID 16723979 . ^ Vignali, M.; Hassan, A. H.; Neely, K. E.; Workman, J. L. (2000-03-15). “ATP-Dependent Chromatin-Remodeling Complexes” . Molecular and Cellular Biology 20 (6): 1899–1910. doi :10.1128/mcb.20.6.1899-1910.2000 . ISSN 0270-7306 . PMC 110808 . PMID 10688638 . https://doi.org/10.1128/mcb.20.6.1899-1910.2000 . ^ a b c d “The poly(ADP-ribose)-dependent chromatin remodeler Alc1 induces local chromatin relaxation upon DNA damage” . Mol. Biol. Cell 27 (24): 3791–3799. (2016). doi :10.1091/mbc.E16-05-0269 . PMC 5170603 . PMID 27733626 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5170603/ .
^ a b “PARP1-dependent kinetics of recruitment of MRE11 and NBS1 proteins to multiple DNA damage sites”. J. Biol. Chem. 283 (2): 1197–208. (2008). doi :10.1074/jbc.M706734200 . PMID 18025084 .
^ a b c “DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139”. J. Biol. Chem. 273 (10): 5858–68. (1998). doi :10.1074/jbc.273.10.5858 . PMID 9488723 .
^ “RNF8 ubiquitylates histones at DNA double-strand breaks and promotes assembly of repair proteins”. Cell 131 (5): 887–900. (2007). doi :10.1016/j.cell.2007.09.040 . PMID 18001824 . ^ “MDC1 directly binds phosphorylated histone H2AX to regulate cellular responses to DNA double-strand breaks”. Cell 123 (7): 1213–26. (2005). doi :10.1016/j.cell.2005.09.038 . PMID 16377563 . ^ “Phospho-dependent interactions between NBS1 and MDC1 mediate chromatin retention of the MRN complex at sites of DNA damage” . EMBO Rep. 9 (8): 795–801. (2008). doi :10.1038/embor.2008.103 . PMC 2442910 . PMID 18583988 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2442910/ . ^ “The NuRD chromatin-remodeling complex regulates signaling and repair of DNA damage” . J. Cell Biol. 190 (5): 741–9. (2010). doi :10.1083/jcb.201001048 . PMC 2935570 . PMID 20805320 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2935570/ . ^ “The hallmarks of cancer”. Cell 100 (1): 57–70. (January 2000). doi :10.1016/S0092-8674(00)81683-9 . PMID 10647931 . ^ “Truncating mutations of hSNF5/INI1 in aggressive paediatric cancer”. Nature 394 (6689): 203–6. (July 1998). Bibcode : 1998Natur.394..203V . doi :10.1038/28212 . PMID 9671307 . ^ “The spectrum of SWI/SNF mutations, ubiquitous in human cancers” . PLOS ONE 8 (1): e55119. (2013). Bibcode : 2013PLoSO...855119S . doi :10.1371/journal.pone.0055119 . PMC 3552954 . PMID 23355908 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3552954/ . ^ a b “The Many Roles of BAF (mSWI/SNF) and PBAF Complexes in Cancer” . Cold Spring Harbor Perspectives in Medicine 6 (8): a026930. (August 2016). doi :10.1101/cshperspect.a026930 . PMC 4968166 . PMID 27413115 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4968166/ .
^ “The retinoblastoma protein and BRG1 form a complex and cooperate to induce cell cycle arrest”. Cell 79 (1): 119–30. (October 1994). doi :10.1016/0092-8674(94)90405-7 . PMID 7923370 . ^ “Frequent BRG1/SMARCA4-inactivating mutations in human lung cancer cell lines”. Human Mutation 29 (5): 617–22. (May 2008). doi :10.1002/humu.20730 . PMID 18386774 . ^ a b c “Dominant-negative SMARCA4 mutants alter the accessibility landscape of tissue-unrestricted enhancers” . Nature Structural & Molecular Biology 25 (1): 61–72. (January 2018). doi :10.1038/s41594-017-0007-3 . PMC 5909405 . PMID 29323272 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5909405/ .
^ a b “Smarca4 ATPase mutations disrupt direct eviction of PRC1 from chromatin” . Nature Genetics 49 (2): 282–288. (February 2017). doi :10.1038/ng.3735 . PMC 5373480 . PMID 27941795 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5373480/ .
^ “Chromatin remodeling: why it is important in cancer”. Oncogene 20 (24): 2988–90. (May 2001). doi :10.1038/sj.onc.1204322 . PMID 11420713 . ^ “Histone deacetylase inhibitors: discovery and development as anticancer agents”. Expert Opinion on Investigational Drugs 14 (12): 1497–511. (December 2005). doi :10.1517/13543784.14.12.1497 . PMID 16307490 . ^ “Histone deacetylase inhibitors: a new class of potential therapeutic agents for cancer treatment” . Clinical Cancer Research 8 (3): 662–4. (2002). PMID 11895892 . http://clincancerres.aacrjournals.org/content/8/3/662.full.pdf . ^ “Histone deacetylase inhibitor selectively induces p21WAF1 expression and gene-associated histone acetylation” . Proceedings of the National Academy of Sciences of the United States of America 97 (18): 10014–9. (August 2000). Bibcode : 2000PNAS...9710014R . doi :10.1073/pnas.180316197 . PMC 27656 . PMID 10954755 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC27656/ . ^ “Quantitation of HDAC1 mRNA expression in invasive carcinoma of the breast*”. Breast Cancer Research and Treatment 94 (1): 11–6. (November 2005). doi :10.1007/s10549-005-6001-1 . PMID 16172792 . ^ “Zolinza (vorinostat) dosing, indications, interactions, adverse effects, and more ”. reference.medscape.com . 2020年9月20日 閲覧。 ^ “New Drug Approvals - Pt. XXII - Romidepsin (Istodax) ”. chembl.blogspot.com . 2020年9月20日 閲覧。 ^ Research, Center for Drug Evaluation and (2020-07-30). “Drug Trials Snapshot: FARYDAK (panobinostat)” (英語). FDA . https://www.fda.gov/drugs/drug-approvals-and-databases/drug-trials-snapshot-farydak-panobinostat . ^ “FDA Approves Panobinostat for Some Patients with Multiple Myeloma - National Cancer Institute ” (英語). www.cancer.gov (2015年3月19日). 2020年9月20日 閲覧。 ^ Coronel, Jaime; Cetina, Lucely; Pacheco, Irlanda; Trejo-Becerril, Catalina; González-Fierro, Aurora; de la Cruz-Hernandez, Erick; Perez-Cardenas, Enrique; Taja-Chayeb, Lucia et al. (2011-12). “A double-blind, placebo-controlled, randomized phase III trial of chemotherapy plus epigenetic therapy with hydralazine valproate for advanced cervical cancer. Preliminary results” . Medical Oncology (Northwood, London, England) 28 Suppl 1 : S540–546. doi :10.1007/s12032-010-9700-3 . ISSN 1559-131X . PMID 20931299 . https://www.ncbi.nlm.nih.gov/pubmed/20931299 . ^ Munster, Pamela; Marchion, Douglas; Bicaku, Elona; Lacevic, Mira; Kim, Jongphil; Centeno, Barbara; Daud, Adil; Neuger, Anthony et al. (2009-04-01). “Clinical and biological effects of valproic acid as a histone deacetylase inhibitor on tumor and surrogate tissues: phase I/II trial of valproic acid and epirubicin/FEC” . Clinical Cancer Research: An Official Journal of the American Association for Cancer Research 15 (7): 2488–2496. doi :10.1158/1078-0432.CCR-08-1930 . ISSN 1078-0432 . PMID 19318486 . https://www.ncbi.nlm.nih.gov/pubmed/19318486 . ^ “Toward the development of potent and selective bisubstrate inhibitors of protein arginine methyltransferases”. Bioorganic & Medicinal Chemistry Letters 20 (7): 2103–5. (April 2010). doi :10.1016/j.bmcl.2010.02.069 . PMID 20219369 . ^ “The biology of chromatin remodeling complexes”. Annual Review of Biochemistry 78 : 273–304. (2009). doi :10.1146/annurev.biochem.77.062706.153223 . PMID 19355820 . ^ a b Parry, Aled John; Narita, Masashi (2016). “Old cells, new tricks: chromatin structure in senescence” . Mammalian Genome 27 (7–8): 320–331. doi :10.1007/s00335-016-9628-9 . ISSN 0938-8990 . PMC 4935760 . PMID 27021489 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4935760/ .
^ Hayflick, L.; Moorhead, P. S. (1961-12-01). “The serial cultivation of human diploid cell strains”. Experimental Cell Research 25 (3): 585–621. doi :10.1016/0014-4827(61)90192-6 . ISSN 0014-4827 . PMID 13905658 . ^ a b Chandra, Tamir; Ewels, Philip Andrew; Schoenfelder, Stefan; Furlan-Magaril, Mayra; Wingett, Steven William; Kirschner, Kristina; Thuret, Jean-Yves; Andrews, Simon et al. (2015-01-29). “Global Reorganization of the Nuclear Landscape in Senescent Cells” . Cell Reports 10 (4): 471–483. doi :10.1016/j.celrep.2014.12.055 . ISSN 2211-1247 . PMC 4542308 . PMID 25640177 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4542308/ .
^ a b c d Sun, Luyang; Yu, Ruofan; Dang, Weiwei (2018-04-16). “Chromatin Architectural Changes during Cellular Senescence and Aging” . Genes 9 (4): 211. doi :10.3390/genes9040211 . ISSN 2073-4425 . PMC 5924553 . PMID 29659513 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5924553/ .
^ a b c Criscione, Steven W.; Teo, Yee Voan; Neretti, Nicola (2016). “The chromatin landscape of cellular senescence” . Trends in Genetics 32 (11): 751–761. doi :10.1016/j.tig.2016.09.005 . ISSN 0168-9525 . PMC 5235059 . PMID 27692431 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5235059/ .
^ a b Yang, Na; Sen, Payel (2018-11-03). “The senescent cell epigenome” . Aging (Albany NY) 10 (11): 3590–3609. doi :10.18632/aging.101617 . ISSN 1945-4589 . PMC 6286853 . PMID 30391936 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6286853/ .
^ a b Basta, Jeannine; Rauchman, Michael (2015). “The Nucleosome Remodeling and Deacetylase (NuRD) Complex in Development and Disease” . Translational Research 165 (1): 36–47. doi :10.1016/j.trsl.2014.05.003 . ISSN 1931-5244 . PMC 4793962 . PMID 24880148 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4793962/ .
^ a b Li, Xueping; Ding, Dong; Yao, Jun; Zhou, Bin; Shen, Ting; Qi, Yun; Ni, Ting; Wei, Gang (2019-07-15). “Chromatin remodeling factor BAZ1A regulates cellular senescence in both cancer and normal cells”. Life Sciences 229 : 225–232. doi :10.1016/j.lfs.2019.05.023 . ISSN 1879-0631 . PMID 31085244 .
^ H, Rayess (2012年4月15日). “Cellular senescence and tumor suppressor gene p16 ” (英語). International journal of cancer . doi :10.1002/ijc.27316 . 2020年9月21日 閲覧。 ^ Tasdemir, Nilgun; Banito, Ana; Roe, Jae-Seok; Alonso-Curbelo, Direna; Camiolo, Matthew; Tschaharganeh, Darjus F.; Huang, Chun-Hao; Aksoy, Ozlem et al. (2016). “BRD4 Connects Enhancer Remodeling to Senescence Immune Surveillance” . Cancer Discovery 6 (6): 612–629. doi :10.1158/2159-8290.CD-16-0217 . ISSN 2159-8290 . PMC 4893996 . PMID 27099234 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4893996/ .