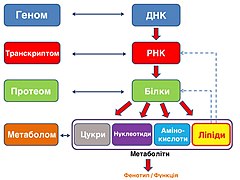
Мультиоміка: спрощено та схематично (без епігеноміки та ін.) Кількість цитувань термінів «Multiomics» і «Multi-omics» у PubMed до 31 грудня 2021 року. Мультиоміка , мульти-оміка , інтегративна оміка — це підхід до біологічного аналізу , спрямований на використання та інтеграцію великої кількості даних , наданої дослідженнями «-омами », такими як геном , протеом , транскриптом , епігеном , епітранскриптом [en] метаболом , інтерактом , мікробіом (метагеном, метатранскриптом, метапротеом) та інші, щоб розвинути комплексне та цілісне розуміння біологічних систем .[ 1] [ 2] [ 3] [ 4]
«Оміксні технології » — це набори передових, високопродуктивних методологій, які використовуються для аналізу широкого діапазону біологічних молекул та їх взаємодії з метою розуміння структури, функції та динаміки біологічних систем на різних рівнях.[ 5] великі дані , щоб знаходити нові асоціації між біологічними об’єктами, точно визначати відповідні біомаркери [en] [ 6] [ 7] [ 8] [ 9]
Мультиоміка має на меті вийти за рамки редукціоністського погляду на розгляд кожного типу даних омік ізольовано. Натомість мультиоміка, як підхід, визнає, що ціле більше, ніж сума його частин. Підходи мультиоміки можуть прояснити, як зміни на одному рівні (наприклад, генетична мутація) можуть поширюватися на інші рівні (наприклад, зміни у функції білка та виробництві метаболітів), таким чином сприяючи складним рисам або хворобливим станам.[ 10] [ 11]
Цей комплексний підхід є особливо ефективним у вирішенні складних біологічних питань і все частіше застосовується в таких сферах, як біомедицина [ 7] біомедична інженерія , біотехнологія , експериментальна біологія , біоінженерія та синтетична біологія , сільське господарство й дослідження навколишнього середовища тощо. Наприклад, у персоналізованій медицині інтеграція мультиомних даних — від геномних до навколишніх — пропонує можливість більш точного прогнозування, профілактики та стратегій лікування для конкретних пацієнтів .[ 1] [ 12] [ 13] [ 14] [ 15]
Однак інтеграція кількох типів даних омік не позбавлена проблем. Вони включають потребу в надійних обчислювальних інструментах і статистичних методах для обробки та аналізу великих складних наборів даних ; необхідність стандартизації та перевірки даних; а також питання етики , конфіденційності та обміну даними.[ 16] біологічних систем і хворобливих процесів, і розвивається експоненційно.[ 6]
Поєднання даних оміксних технологій з іншими біомедичними даними для цілісного дослідження здоров'я [ 1] Хронологія основних технологічних розробок і віх в різних аналізах оміксних технологій Зазвичай, дослідження мультиоміки поєднують певні оміксні технології, але ці дані також можуть бути поєднані і з іншими біомедичними даними.[ 1]
Геноміка — це наука про повний набір генів в організмі (геном), включаючи їх організацію, функції та взаємодію.[ 17] проекту «Геном людини» у 2003 році стало знаменним досягненням, яке проклало шлях для передових досліджень генетики людини та інших видів.[ 18] секвенування цілого генома [ 19] [ 20] повногеномне дослідження асоціацій [en] [ 21] порівняльну геноміку [en] [ 22] [ 23] [ 24] [ 25] див. Геноміка , Біоінформатика )
Епігеноміка — це дослідження повного набору епігенетичних модифікацій генетичного матеріалу клітини, відомого як епігеном.[ 26] [ 27] [ 28] [ 29] експресію генів , "вмикаючи" чи "вимикаючи" їх, не змінюючи базову послідовність ДНК.[ 30]
Профіль метилювання ДНК : Вивчення статусу метилювання специфічних залишків цитозину в ДНК. Патерни метилювання часто асоціюються з глушінням генів і можуть бути проаналізовані за допомогою таких методів, як бісульфітне секвенування [en] методи аналізу мікрочипів [en] [ 31] Дані модифікації гістонів — ChIP-Seq (Секвенування імунопреципітацією хроматину): визначення місць і моделей модифікацій гістонів (наприклад, ацетилювання , метилювання ), пов’язаних з регуляцією генів. Секвенування ChIP поєднує імунопреципітацію хроматину з високопродуктивним секвенуванням.Секвенування малих РНК [en] некодуючих РНК (наприклад, мікроРНК [ 32] малих інтерферуючих РНК ), які відіграють вирішальну роль у регуляції генів після транскрипції .[ 33] Аналіз довгих некодуючих РНК (lncRNA) : Дослідження функцій і регуляторних ролей довгих некодуючих РНК у експресії генів та епігенетичній регуляції.[ 34] Методи фіксації конформації хромосом хроматину в клітинному ядрі . Дані Hi-C дають зрозуміти, як хроматинові петлі та взаємодії впливають на регуляцію генів і архітектуру геному .Повноепігеномне дослідження асоціацій [en]
Транскриптоміка передбачає вивчення повного набору транскриптів РНК , які виробляються геномом за певних умов.[ 35] секвенування РНК [en] [ 36] Див. також Транскрипція (біологія) )
Епітранскриптоміка — це дослідження епітранскриптому [en] [ 37] [ 38] [ 39] [ 29] [ 40]
Епітранскриптоміка зосереджена на вивченні посттранскрипційних модифікацій молекул РНК. Ці модифікації, такі як N6-метиладенозин (m6A)[ 41] [ 42] 5-метилцитозин (m5C)[ 43] [ 44] [ 45] трансляції РНК.
Епітранскриптомне секвенування [en] [ 46] [ 47] [ 48] [ 49]
Протеоміка передбачає широкомасштабне вивчення білків , включаючи їх структуру, функції та взаємодії, — протеом .[ 50] [ 51] [ 52] [ 53] [ 54] Мас-спектрометрія та білкові мікрочіпи є широко використовуваними методами в протеоміці.[ 55] [ 56]
Функціональна протеоміка — дослідження білок-білкових взаємодій, субклітинної локалізації та функцій білка в клітинних шляхах.[ 57] [ 58]
Ліпідоміка — це широкомасштабне дослідження шляхів і мереж клітинних ліпідів у біологічних системах.[ 59] аналітичної хімії та мас-спектрометрії[ 60] [ 61] [ 62]
Глікоміка — це комплексне дослідження всіх гліканових структур (вуглеводів ) клітини, тканини або організму.[ 63] [ 64] [ 65] [ 66]
Метаболоміка — це дослідження повного набору дрібномолекулярних хімічних речовин, знайдених у біологічному зразку. Досліджуючи ці метаболіти, дослідники можуть отримати уявлення про фізіологічні стани та реакцію на захворювання або зміни навколишнього середовища.[ 67] [ 68] див. Біоінформатика )
Інтерактоміка відноситься до вивчення інтерактома , усієї сукупності молекулярних взаємодій у клітинах . Це може включати білок-білок[ 69] [ 70] [ 71] [ 72] [ 73] [ 74] [ 75] [ 76] [ 77]
Мікробіоміка передбачає характеристику та аналіз мікробних спільнот, включаючи бактерії , археї , віруси , гриби , найпростіші та водорості , у визначеному середовищі.[ 78] Див. також Мікробіом , Мікробіом людини , Мікробіота кишківника )
Метагеноміка — це дослідження генетичного матеріалу , отриманого безпосередньо зі зразків навколишнього середовища [ 79] [ 80] мікробіологічними методами.[ 81] [ 82]
Метатранскриптоміка — розділ транскриптоміки та мікробіоміки, який займається вивченням транскриптомів у мікробних спільнотах. Він пропонує розуміння активних функціональних елементів спільноти та може проілюструвати, як громади реагують на подразники навколишнього середовища. Метатранскриптоміка також може допомогти ідентифікувати гени, що експресуються окремими членами спільноти, досліджуючи функціональні ролі різних членів спільноти.[ 83] [ 84]
Метапротеоміка передбачає вивчення всього мікробного протеому в певний момент часу. Метапротеоміка надає функціональну інформацію про мікробні спільноти, що робить її ключовим інструментом для розуміння взаємодії мікробів і динаміки спільнот у їхньому природному середовищі існування. Такий підхід також допомагає з’ясувати функціональні ролі окремих членів і спільноти в цілому.[ 85] [ 86] [ 87]
Технології секвенування наступного покоління (NGS) зробили революцію в геноміці та інших галузях оміки.[ 88] [ 89] [ 90] [ 91] [ 92] [ 93] [ 94]
Технологія мікрочипів[ 95] однонуклеотидних поліморфізмів (SNP) і варіацій кількості копій (CNV) у геноміці. Секвенування РНК (RNA-Seq), витіснило технологію мікрочіпів і стало методикою вибору, завдяки своїй здатності вимірювати експресію генів з високою чутливістю та точністю, а також ідентифікувати нові транскрипти.[ 96]
Мас-спектрометрія є ключовим методом у протеоміці[ 56] [ 68] [ 60] [ 65] [ 66]
Необхідним компонентом досліджень мультиоміки є використання обчислювальних інструментів для аналізу та інтеграції великих і складних наборів даних оміки, що є сферою досліджень в біоінформатиці . Інструменти для інтеграції даних включають статистичні підходи, алгоритми машинного навчання та інструменти мережевого аналізу.[ 97] Бази даних і програмні платформи[ 98] [ 99] [ 100]
Паралельно з прогресом у молекулярній , системній та обчислювальній біології , активно розвиваються програми машинного навчання для аналізу біомедичних даних . Інтеграція мультиомічного аналізу даних і машинного навчання призвела до відкриття нових біомаркерів.[ 101] [ 102] [ 103] [ 104] mixOmics реалізує метод, заснований на частковій регресії найменших квадратів [en] [ 105]
Відгалуженням галузі мультиоміки є аналіз багаторівневих одноклітинних даних, які називають одноклітинною [en] [ 106] [ 107] [ 108] [ 109] [ 110]
Методи паралельного одноклітинного геномного та транскриптомного аналізу можуть базуватися на одночасній ампліфікації[ 111] РНК і геномної ДНК .[ 112] некодуючих геномних ділянок та інформації щодо варіації кількості копій. Розширенням цієї методології є інтеграція одноклітинних транскриптомів до одноклітинних метиломів, поєднання одноклітинного бісульфітного секвенування [en] [ 113] [ 114] [ 115] ATAC-seq [en] [ 116] Hi-C .[ 117]
Проблемою є інтеграція протеомних[ 51] [ 54] [ 118] [ 119] [ 118] [ 120] [ 119]
Системи CRISPR-Cas, зокрема CRISPR-Cas9 , зробили революцію в геномних дослідженнях. Їх можна використовувати для точного редагування геномів, що дозволяє проводити функціональні дослідження генів і генетичних варіантів.
Завдяки точності CRISPR-Cas9 дослідники можуть створювати певні генетичні пертубації (наприклад, генні нокаути або специфічні мутації) контрольованим чином, а потім використовувати різні методи оміки (геноміка, транскриптоміка, протеоміка, метаболоміка тощо), щоб зрозуміти наступні наслідки цих змін.[ 121]
За тим же принципом, систему dCas9, де білок Cas9 є «мертвим» або каталітично неактивним, можна використовувати для цілеспрямованого редагування епігенома, якщо його злити з ферментами , які модифікують епігеном, такими як ДНК-метилтрансферази або гістон-ацетилтрансферази .[ 122] [ 123]
Інтеграція даних мультиоміки є вирішальним кроком у дослідженнях мультиоміки, метою якого є надання повного уявлення про біологічні системи на різних молекулярних рівнях. Завдання класифікації методів інтеграції даних мультиоміки є складним через різноманітні методології з різними цілями. Однак їх можна широко класифікувати на основі їхніх базових статистичних стратегій, їхньої біологічної цілі та способу обробки кількох типів даних омік.[ 4]
Існує два загальні підходи до організації вхідних даних для подальшої їх інтеграції та аналізу: методи ансамблю даних і методи ансамблю моделі. Методи ансамблю даних об’єднують дані мультиоміки з різних молекулярних шарів в єдину матрицю як вхідні дані. Навпаки, методи ансамблю моделі аналізують кожні дані омік незалежно, а потім об’єднують результати для побудови інтегративного аналізу.[ 124] [ 125] [ 126] [ 127] [ 126]
Послідовний аналіз
Методи на основі канонічний кореляційного аналізу (CCA) і співінерційного аналізу (CIA)
Методи на основі факторного аналізу
Кластеризація на основі ядра
Метод кластеризації на основі матричної факторизації
Метод баєсової кластеризації
Багатовимірний метод кластеризації
Мережі на основі матриці факторизації (MF-Based)
Баєсові мережі Мережі на основі поширення (NP-Based)
Кореляційні та інші мережі
В інтеграції мультиомічних даних є кілька проблем та викликів які стоять перед дослідниками. Вони включають потребу в:
Великих та високоякісні наборах даних, оскільки методи інтеграції часто вимагають великих обсягів даних для надійних результатів.
Методи обробки відсутніх даних, оскільки не всі типи даних омік можуть бути доступними для всіх зразків.
Ефективні методи нормалізації та трансформації для врахування відмінностей у масштабі та розподілі між різними типами даних омік.
Методи врахування пакетниз ефектів та інших джерел небіологічної варіації.[ 128] Оскільки дослідження мультиоміки стають все більш поширеними, виникає потреба в розробці більш складних методів інтеграції, програмних засобів[ 129] [ 130] [ 127] [ 131]
Прогрес в мультиоміці обіцяє заповнити прогалини в розумінні людського здоров’я [ 1] хвороб [ 132] [ 133] [ 134] [ 135] [ 136] неврологічних патологій [ 137] персоналізованої медицини [ 12] [ 13] [ 14] [ 15]
Інтегруючи геномні, транскриптомні, протеомні та інші омічні дані, клініцисти можуть розробити всебічне розуміння здоров’я та хворобливого стану пацієнта, керуючи вибором найбільш ефективних методів лікування та профілактичних заходів. Наприклад, огляд 2023 року виділив такі 5 основних застосувань мультиоміки в трансляційній медицині: 1) виявлення молекулярних моделей, пов’язаних із захворюванням, 2) ідентифікація підтипу, 3) діагностика та прогноз, 4) прогнозування реакції на ліки та 5) розуміння регуляторних процесів.[ 124]
Інтегруючи кілька шарів оміки, дослідники можуть отримати глибше розуміння механізмів захворювання на молекулярному рівні. Наприклад, геномні дані можуть ідентифікувати генетичні варіанти, пов’язані з хворобою, транскриптомні дані можуть виявити пов’язані зміни в експресії генів, протеомні дані можуть показати зміни рівнів і модифікацій білка, а метаболомічні дані можуть ідентифікувати пов’язані метаболічні зміни.[ 124] [ 138]
Дані мультиоміки можуть допомогти класифікувати та підтипувати захворювання, особливо складні захворювання, у більш конкретні категорії на основі їхніх молекулярних профілів. Це може покращити діагностику та лікування. Наприклад, різні підтипи раку або діабету можуть мати різні молекулярні профілі, які по-різному реагують на лікування.[ 139] [ 124]
Дані мультиоміки можуть допомогти у відкритті біомаркерів для діагностики захворювань, прогнозу та відповіді на лікування. Шляхом порівняння профілів омік захворювання та здорового стану можна ідентифікувати потенційні біомаркери.[ 140] [ 141]
Мультиоміка також може відігравати вирішальну роль у відкритті та розробці ліків . Наприклад, розуміння омік-профілів захворювань може допомогти в ідентифікації потенційних мішеней ліків.[ 142] [ 143] [ 138] [ 144] [ 145]
Огляд фаз 1 і 2 проекту мікробіома людини. Друга фаза проекту людського мікробіому вартістю 170 мільйонів доларів була зосереджена на інтеграції даних пацієнтів у різні набори даних омік, враховуючи генетику господаря, клінічну інформацію та склад мікробіому .[ 146] [ 147] мікробіот кишківника та носа з діабетом 2 типу [ 148] кишківника [ 149] мікробіоти вагіни й передчасних пологів [ 150]
Складність взаємодій в імунній системі людини спонукала до створення великої кількості пов’язаних з імунологією багатомасштабних омічних даних.[ 151] [ 152] [ 153] [ 13] [ 154] імунної відповіді на інфекційні захворювання, такі як дитяча чікунгунья [ 155] аутоімунні захворювання .[ 156] вакцин .[ 157] плазми та транскриптома імунної системи у відповідь на вакцинацію проти оперізувального герпесу .[ 158]
Старіння організму зумовлене взаємопов’язаними молекулярними змінами, що охоплюють внутрішні та позаклітинні фактори. Комбінований аналіз високопродуктивних наборів даних мультиоміки на популяційному або одноклітинному рівнях може забезпечити багатовимірний інтегрований профіль гетерогенних процесів старіння з безпрецедентною продуктивністю та деталізацією. Ці нові стратегії дозволяють досліджувати молекулярний профіль і регуляторний статус експресії генів під час старіння, і, у свою чергу, сприяють розробці нових заходів щодо старіння та омолодження .[ 159] [ 160]
У дослідженні 2023 року, опублікованому в Nature Communications старінням на рівні епігеному та комплексним підходом до довголіття та омолодження (див. Механізми старіння ). Вони ідентифікували 22 асоціації з високим ступенем достовірності з епігенетичним старінням і 7 з багатофакторним довголіттям, з кількома генами, пов’язаними з передачею сигналів інсуліну , функцією мітохондрій , клітинною реакцією на стрес і метаболізмом . Дослідження також виявило вплив імунних клітин на прискорення епігенетичного старіння, що передбачає потенційні можливі мішені для терапевтичних втручаннь. Подільші дослідження в цій сфері допоможуть визначити конкретні препарати чи втручання для терапії проти старіння. Інтеграція мультиоміки в точні стратегії омолодження є багатообіцяючою у визначенні лікарських цілей для продовження здорових років життя .[ 161]
У галузі сільського господарства мультиомічні підходи застосовуються для покращення ефективності селекції [ 162] [ 163] сільськогосподарських культур і тварин. Геномні дані можуть ідентифікувати корисні генетичні ознаки, тоді як транскриптомні, протеомні та метаболомні дані можуть допомогти зрозуміти їх вплив. Це може керувати стратегіями розведення та зусиллями щодо генетичної модифікації.[ 164]
Також, застосування мультиоміки в сільському господарстві є важливим для сприяння сталим методам ведення сільського господарства. Оскільки надмірне використання хімічних добрив призвело до численних екологічних проблем, глибоке розуміння агроекосистеми , яка містить рослини , мікроби та ґрунт , є необхідним для сталого сільського господарства. Наприклад, мультиомічне дослідження структури агроекосистеми, опубліковане в PNAS органічний азот є ключовим компонентом, який сприяє врожайності сільськогосподарських культур за умови соляризації ґрунту, навіть при наявності неорганічного азоту.[ 165]
В екології та дослідженнях навколишнього середовища методи мультиоміки використовуються для вивчення екосистем на молекулярному рівні. Наприклад, метагеномні, метатранскриптомні, метапротеомні та метаметаболомні дані можуть дати розуміння складу та функції мікробних спільнот у різних середовищах та в конкретних ризосферах .[ 166] [ 167] [ 168] [ 169]
Проект Bioconductor курує різні пакети R, спрямовані на інтеграцію даних омік:
База даних OmicTools[ 175]
PaintOmics , веб-ресурс для візуалізації наборів даних мультиоміки[ 176] [ 177] SIGMA, програма Java, зосереджена на інтегрованому аналізі наборів даних раку[ 178]
iOmicsPASS, інструмент на C++ для прогнозування фенотипу на основі мультиоміки[ 179]
Grimon , графічний інтерфейс R для візуалізації мультиомічних даних[ 180] Omics Pipe , фреймворк на Python для відтворюваної автоматизації мультиомічного аналізу даних[ 181]
Основним обмеженням класичних омічних досліджень є виділення лише одного рівня біологічної складності. Наприклад, транскриптомні дослідження можуть надавати інформацію на рівні транскриптів , але багато різних об’єктів та процесів впливають на біологічний стан зразка (геномні варіанти , посттрансляційні модифікації , продукти метаболізму, взаємодіючі організми тощо). З появою високопродуктивної біології стає все більш доступним проводити численні вимірювання, дозволяючи трансдоменні (наприклад, рівні РНК і білка) кореляції та висновки. Ці кореляції допомагають будувати більш повні біологічні мережі, заповнюючи прогалини в наших знаннях.
Інтеграція даних, однак, не є легким завданням. Щоб полегшити процес, були створені бази даних і конвеєри для систематичного дослідження мультиомічних даних:
Однак, бази даних містять різні представлення того самого біологічного шляху, що може призвести до різних результатів аналізу статистичного збагачення та прогнозних моделей у контексті точної медицини. Тому рекомендується використовувати бази даних декількох представлень шляхів або інтегративні бази даних.[ 195]
↑ а б в г д Babu, Mohan; Snyder, Michael (2023-06). Multi-Omics Profiling for Health . Molecular & Cellular Proteomics . Т. 22, № 6. с. 100561. doi :10.1016/j.mcpro.2023.100561 . ISSN 1535-9476 . PMC 10220275 PMID 37119971 . Процитовано 14 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Hasin, Yehudit; Seldin, Marcus; Lusis, Aldons (5 травня 2017). Multi-omics approaches to disease . Genome Biology . Т. 18, № 1. с. 83. doi :10.1186/s13059-017-1215-1 . ISSN 1474-760X . PMC 5418815 PMID 28476144 . Процитовано 11 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Pinu, Farhana R.; Beale, David J.; Paten, Amy M.; Kouremenos, Konstantinos; Swarup, Sanjay; Schirra, Horst J.; Wishart, David (2019-04). Systems Biology and Multi-Omics Integration: Viewpoints from the Metabolomics Research Community . Metabolites (англ.) . Т. 9, № 4. с. 76. doi :10.3390/metabo9040076 . ISSN 2218-1989 . PMC 6523452 PMID 31003499 . Процитовано 14 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ а б Kang Ning (2023). Methodologies of Multi-Omics Data Integration and Data Mining: Techniques and Applications (eng) . Springer . ISBN 978-981-19-8209-5 ↑ Hood, Leroy; Tian, Qiang (1 серпня 2012). Systems Approaches to Biology and Disease Enable Translational Systems Medicine . Genomics, Proteomics & Bioinformatics (англ.) . Т. 10, № 4. с. 181—185. doi :10.1016/j.gpb.2012.08.004 . ISSN 1672-0229 . PMC 3844613 PMID 23084773 . Процитовано 11 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ а б Kang Ning (2023). Methodologies of Multi-Omics Data Integration and Data Mining: Techniques and Applications Сінгапур : Springer . с. 167. ISBN 978-981-19-8209-5 ↑ а б Christopher Gerner, Michelle Hill (2021). Integrative Multi-Omics in Biomedical Research (англ) . MDPI , Biomolecules. с. 178. ISBN 978-3-0365-2582-2 ↑ Tarazona, S., Balzano-Nogueira, L., & Conesa, A. (2018). Multiomics Data Integration in Time Series Experiments. DOI :10.1016/bs.coac.2018.06.005
↑ Bersanelli, Matteo; Mosca, Ettore; Remondini, Daniel; Giampieri, Enrico; Sala, Claudia; Castellani, Gastone; Milanesi, Luciano (20 січня 2016). Methods for the integration of multi-omics data: mathematical aspects . BMC Bioinformatics . Т. 17, № 2. с. S15. doi :10.1186/s12859-015-0857-9 . ISSN 1471-2105 . PMC 4959355 PMID 26821531 . Процитовано 11 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Subramanian, Indhupriya; Verma, Srikant; Kumar, Shiva; Jere, Abhay; Anamika, Krishanpal (2020-01). Multi-omics Data Integration, Interpretation, and Its Application . Bioinformatics and Biology Insights (англ.) . Т. 14. с. 117793221989905. doi :10.1177/1177932219899051 . ISSN 1177-9322 . PMC 7003173 PMID 32076369 . Процитовано 11 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Mavromatis, Lucas A.; Rosoff, Daniel B.; Bell, Andrew S.; Jung, Jeesun; Wagner, Josephin; Lohoff, Falk W. (19 квітня 2023). Multi-omic underpinnings of epigenetic aging and human longevity . Nature Communications (англ.) . Т. 14, № 1. с. 2236. doi :10.1038/s41467-023-37729-w . ISSN 2041-1723 . Процитовано 11 червня 2023 . ↑ а б Hood, Leroy; Flores, Mauricio (15 вересня 2012). A personal view on systems medicine and the emergence of proactive P4 medicine: predictive, preventive, personalized and participatory . New Biotechnology (англ.) . Т. 29, № 6. с. 613—624. doi :10.1016/j.nbt.2012.03.004 . ISSN 1871-6784 . Процитовано 11 червня 2023 . ↑ а б в He, Feng Q.; Ollert, Markus; Balling, Rudi; Bode, Sebastian F. N.; Delhalle, Sylvie (6 лютого 2018). A roadmap towards personalized immunology . NPJ Systems Biology and Applications . 4 (1): 9. doi :10.1038/s41540-017-0045-9 . ISSN 2056-7189 . PMC 5802799 PMID 29423275 . ↑ а б Hu, Cheng; Jia, Weiping (18 серпня 2021). Multi-omics profiling: the way toward precision medicine in metabolic diseases . Journal of Molecular Cell Biology . doi :10.1093/jmcb/mjab051 . ISSN 1674-2788 . PMC 8697344 PMID 34406397 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ а б Ahmed, Zeeshan (1 січня 2022). Teplow, David B. (ред.). Chapter Four - Precision medicine with multi-omics strategies, deep phenotyping, and predictive analysis (англ.) . Т. 190. Academic Press. с. 101—125. doi :10.1016/bs.pmbts.2022.02.002 . ↑ Wang, Bo; Mezlini, Aziz M.; Demir, Feyyaz; Fiume, Marc; Tu, Zhuowen; Brudno, Michael; Haibe-Kains, Benjamin; Goldenberg, Anna (2014-03). Similarity network fusion for aggregating data types on a genomic scale . Nature Methods (англ.) . Т. 11, № 3. с. 333—337. doi :10.1038/nmeth.2810 . ISSN 1548-7105 . Процитовано 11 червня 2023 . ↑ Alberts, Bruce; Johnson, Alexander; Lewis, Julian; Raff, Martin; Roberts, Keith; Walter, Peter (31 грудня 2007). Molecular Biology of the Cell . doi :10.1201/9780203833445 . Процитовано 11 червня 2023 . ↑ International Human Genome Sequencing Consortium (2004-10). Finishing the euchromatic sequence of the human genome . Nature (англ.) . Т. 431, № 7011. с. 931—945. doi :10.1038/nature03001 . ISSN 1476-4687 . Процитовано 11 червня 2023 . ↑ Austin-Tse, Christina A.; Jobanputra, Vaidehi; Perry, Denise L.; Bick, David; Taft, Ryan J.; Venner, Eric; Gibbs, Richard A.; Young, Ted; Barnett, Sarah (8 квітня 2022). Best practices for the interpretation and reporting of clinical whole genome sequencing . npj Genomic Medicine (англ.) . Т. 7, № 1. с. 1—13. doi :10.1038/s41525-022-00295-z . ISSN 2056-7944 . Процитовано 11 червня 2023 . ↑ Uelze, Laura; Grützke, Josephine; Borowiak, Maria; Hammerl, Jens Andre; Juraschek, Katharina; Deneke, Carlus; Tausch, Simon H.; Malorny, Burkhard (2020-12). Typing methods based on whole genome sequencing data . One Health Outlook (англ.) . Т. 2, № 1. doi :10.1186/s42522-020-0010-1 . ISSN 2524-4655 . PMC 7993478 PMID 33829127 . Процитовано 11 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Uffelmann, Emil; Huang, Qin Qin; Munung, Nchangwi Syntia; de Vries, Jantina; Okada, Yukinori; Martin, Alicia R.; Martin, Hilary C.; Lappalainen, Tuuli; Posthuma, Danielle (26 серпня 2021). Genome-wide association studies . Nature Reviews Methods Primers (англ.) . Т. 1, № 1. с. 1—21. doi :10.1038/s43586-021-00056-9 . ISSN 2662-8449 . Процитовано 11 червня 2023 . ↑ Setubal, João Carlos; Stoye, Jens; Stadler, Peter F., ред. (2018). Comparative genomics: methods and protocols ISBN 978-1-4939-7461-0 ↑ Genereux, Diane P.; Serres, Aitor; Armstrong, Joel; Johnson, Jeremy; Marinescu, Voichita D.; Murén, Eva; Juan, David; Bejerano, Gill; Casewell, Nicholas R. (2020-11). A comparative genomics multitool for scientific discovery and conservation . Nature (англ.) . Т. 587, № 7833. с. 240—245. doi :10.1038/s41586-020-2876-6 . ISSN 1476-4687 . Процитовано 11 червня 2023 . ↑ Gasperskaja, Evelina; Kučinskas, Vaidutis (25 квітня 2017). The most common technologies and tools for functional genome analysis . Acta medica Lituanica . Т. 24, № 1. с. 1—11. doi :10.6001/actamedica.v24i1.3457 . ISSN 2029-4174 . PMC 5467957 PMID 28630587 . Процитовано 11 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Nusrat, S.; Harbig, T.; Gehlenborg, N. (2019-06). Tasks, Techniques, and Tools for Genomic Data Visualization . Computer Graphics Forum (англ.) . Т. 38, № 3. с. 781—805. doi :10.1111/cgf.13727 . ISSN 0167-7055 . PMC 6876635 PMID 31768085 . Процитовано 11 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Fazzari, Melissa J.; Greally, John M. (2010). Bang, Heejung; Zhou, Xi Kathy; van Epps, Heather L.; Mazumdar, Madhu (ред.). Introduction to Epigenomics and Epigenome-Wide Analysis (англ.) . Totowa, NJ: Humana Press. с. 243—265. doi :10.1007/978-1-60761-580-4_7 . ISBN 978-1-60761-580-4 ↑ Appasani, Krishnarao (2012). Epigenomics, from chromatin biology to therapeutics . Cambridge (GB): Cambridge University Press . ISBN 978-1-107-00382-8 ↑ Hatada, Izuh; Horii, Takuro, ред. (2023). Epigenomics: methods and protocols . Methods in molecular biology. New York, NY: Humana Press, Springer . ISBN 978-1-0716-2723-5 ↑ а б Yujing Li та ін. (2023). Epigenomic and Epitranscriptomic Basis of Development and Human Disease ISBN 978-2-83251-817-5 ↑ Bannister, Andrew J.; Kouzarides, Tony (2011-03). Regulation of chromatin by histone modifications . Cell Research (англ.) . Т. 21, № 3. с. 381—395. doi :10.1038/cr.2011.22 . ISSN 1748-7838 . PMC 3193420 PMID 21321607 . Процитовано 11 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Papanicolau-Sengos, Antonios; Aldape, Kenneth (24 січня 2022). DNA Methylation Profiling: An Emerging Paradigm for Cancer Diagnosis . Annual Review of Pathology: Mechanisms of Disease (англ.) . Т. 17, № 1. с. 295—321. doi :10.1146/annurev-pathol-042220-022304 . ISSN 1553-4006 . Процитовано 6 вересня 2023 . ↑ Androvic, Peter; Benesova, Sarka; Rohlova, Eva; Kubista, Mikael; Valihrach, Lukas (2022-04). Small RNA-Sequencing for Analysis of Circulating miRNAs . The Journal of Molecular Diagnostics . Т. 24, № 4. с. 386—394. doi :10.1016/j.jmoldx.2021.12.006 . ISSN 1525-1578 . Процитовано 6 вересня 2023 . ↑ Li, Jia; Zhang, Zhirong; Zhuang, Yinghua; Wang, Fengchao; Cai, Tao (9 травня 2023). Small RNA transcriptome analysis using parallel single-cell small RNA sequencing . Scientific Reports (англ.) . Т. 13, № 1. с. 7501. doi :10.1038/s41598-023-34390-7 . ISSN 2045-2322 . Процитовано 6 вересня 2023 . ↑ Mattick, John S.; Amaral, Paulo P.; Carninci, Piero; Carpenter, Susan; Chang, Howard Y.; Chen, Ling-Ling; Chen, Runsheng; Dean, Caroline; Dinger, Marcel E. (2023-06). Long non-coding RNAs: definitions, functions, challenges and recommendations . Nature Reviews Molecular Cell Biology (англ.) . Т. 24, № 6. с. 430—447. doi :10.1038/s41580-022-00566-8 . ISSN 1471-0080 . PMC 10213152 PMID 36596869 . Процитовано 6 вересня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Wang, Zhong; Gerstein, Mark; Snyder, Michael (2009-01). RNA-Seq: a revolutionary tool for transcriptomics . Nature Reviews Genetics (англ.) . Т. 10, № 1. с. 57—63. doi :10.1038/nrg2484 . ISSN 1471-0064 . PMC 2949280 PMID 19015660 . Процитовано 11 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Stark, Rory; Grzelak, Marta; Hadfield, James (2019-11). RNA sequencing: the teenage years . Nature Reviews Genetics (англ.) . Т. 20, № 11. с. 631—656. doi :10.1038/s41576-019-0150-2 . ISSN 1471-0064 . Процитовано 11 червня 2023 . ↑ Kumar, Suresh; Mohapatra, Trilochan (2021). Deciphering Epitranscriptome: Modification of mRNA Bases Provides a New Perspective for Post-transcriptional Regulation of Gene Expression . Frontiers in Cell and Developmental Biology . Т. 9. doi :10.3389/fcell.2021.628415 . ISSN 2296-634X . PMC 8010680 PMID 33816473 . Процитовано 11 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Shen, Lisha; Ma, Jinqi; Li, Ping; Wu, Yujin; Yu, Hao (7 березня 2023). Recent advances in the plant epitranscriptome . Genome Biology (англ.) . Т. 24, № 1. doi :10.1186/s13059-023-02872-6 . ISSN 1474-760X . PMC 9990323 PMID 36882788 . Процитовано 10 вересня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Stefan Jurga, Jan Barciszewski (2021). Epitranscriptomics Springer . с. 632. ISBN 978-3-030-71611-0 ↑ Matsumura, Yoshihiro; Wei, Fan-Yan; Sakai, Juro (2023-03). Epitranscriptomics in metabolic disease . Nature Metabolism (англ.) . Т. 5, № 3. с. 370—384. doi :10.1038/s42255-023-00764-4 . ISSN 2522-5812 . Процитовано 11 червня 2023 . ↑ Xia, Zhen; Tang, Min; Ma, Jiayan; Zhang, Hongyan та ін. (28 червня 2021). Epitranscriptomic editing of the RNA N6-methyladenosine modification by dCasRx conjugated methyltransferase and demethylase . Nucleic Acids Research doi :10.1093/nar/gkab517 . ISSN 0305-1048 . Процитовано 10 вересня 2023 . ↑ Liang, Zhanmin; Ye, Haokai; Ma, Jiongming; Wei, Zhen; Wang, Yue; Zhang, Yuxin; Huang, Daiyun; Song, Bowen; Meng, Jia (17 серпня 2023). m6A-Atlas v2.0: updated resources for unraveling the N 6-methyladenosine (m6A) epitranscriptome among multiple species . Nucleic Acids Research doi :10.1093/nar/gkad691 . ISSN 0305-1048 . Процитовано 10 вересня 2023 . ↑ Trixl, Lukas; Lusser, Alexandra (2019-01). The dynamic RNA modification 5‐methylcytosine and its emerging role as an epitranscriptomic mark . WIREs RNA (англ.) . Т. 10, № 1. doi :10.1002/wrna.1510 . ISSN 1757-7004 . PMC 6492194 PMID 30311405 . Процитовано 10 вересня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Ma, Jiongming; Song, Bowen; Wei, Zhen; Huang, Daiyun; Zhang, Yuxin; Su, Jionglong; de Magalhães, João Pedro; Rigden, Daniel J; Meng, Jia (7 січня 2022). m5C-Atlas: a comprehensive database for decoding and annotating the 5-methylcytosine (m5C) epitranscriptome . Nucleic Acids Research (англ.) . Т. 50, № D1. с. D196—D203. doi :10.1093/nar/gkab1075 . ISSN 0305-1048 . Процитовано 10 вересня 2023 . ↑ Zhang, Yuexiu; Zhang, Li-Sheng; Dai, Qing; Chen, Phylip; Lu, Mijia; Kairis, Elizabeth L.; Murugaiah, Valarmathy; Xu, Jiayu; Shukla, Rajni Kant (18 жовтня 2022). 5-methylcytosine (m 5 C) RNA modification controls the innate immune response to virus infection by regulating type I interferons . Proceedings of the National Academy of Sciences (англ.) . Т. 119, № 42. doi :10.1073/pnas.2123338119 . ISSN 0027-8424 . PMC 9586267 . Процитовано 10 вересня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Dominissini, Dan; Moshitch-Moshkovitz, Sharon; Schwartz, Schraga; Salmon-Divon, Mali; Ungar, Lior; Osenberg, Sivan; Cesarkas, Karen; Jacob-Hirsch, Jasmine; Amariglio, Ninette (2012-05). Topology of the human and mouse m6A RNA methylomes revealed by m6A-seq . Nature (англ.) . Т. 485, № 7397. с. 201—206. doi :10.1038/nature11112 . ISSN 1476-4687 . Процитовано 10 вересня 2023 . ↑ McIntyre, Alexa B. R.; Gokhale, Nandan S.; Cerchietti, Leandro; Jaffrey, Samie R.; Horner, Stacy M.; Mason, Christopher E. (20 квітня 2020). Limits in the detection of m6A changes using MeRIP/m6A-seq . Scientific Reports (англ.) . Т. 10, № 1. с. 6590. doi :10.1038/s41598-020-63355-3 . ISSN 2045-2322 . Процитовано 10 вересня 2023 . ↑ Winans, Shelby; Beemon, Karen (2019-08). m5C Goes Viral . Cell Host & Microbe doi :10.1016/j.chom.2019.07.019 . ISSN 1931-3128 . Процитовано 10 вересня 2023 . ↑ He, Zhizhou; Xu, Jing; Shi, Haoran; Wu, Shuxiang (2022-04). m5CRegpred: Epitranscriptome Target Prediction of 5-Methylcytosine (m5C) Regulators Based on Sequencing Features . Genes (англ.) . Т. 13, № 4. с. 677. doi :10.3390/genes13040677 . ISSN 2073-4425 . PMC 9025882 PMID 35456483 . Процитовано 10 вересня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Anderson, N. Leigh; Anderson, Norman G. (1998-08). Proteome and proteomics: New technologies, new concepts, and new words . Electrophoresis (англ.) . Т. 19, № 11. с. 1853—1861. doi :10.1002/elps.1150191103 . ISSN 0173-0835 . Процитовано 11 червня 2023 . ↑ а б Specht, Harrison; Slavov, Nikolai (3 серпня 2018). Transformative Opportunities for Single-Cell Proteomics . Journal of Proteome Research (англ.) . Т. 17, № 8. с. 2565—2571. doi :10.1021/acs.jproteome.8b00257 . ISSN 1535-3893 . PMC 6089608 PMID 29945450 . Процитовано 11 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Cui, Miao; Cheng, Chao; Zhang, Lanjing (2022-11). High-throughput proteomics: a methodological mini-review . Laboratory Investigation (англ.) . Т. 102, № 11. с. 1170—1181. doi :10.1038/s41374-022-00830-7 . ISSN 1530-0307 . Процитовано 11 червня 2023 . ↑ Dupree, Emmalyn J.; Jayathirtha, Madhuri; Yorkey, Hannah; Mihasan, Marius; Petre, Brindusa Alina; Darie, Costel C. (2020-09). A Critical Review of Bottom-Up Proteomics: The Good, the Bad, and the Future of This Field . Proteomes (англ.) . Т. 8, № 3. с. 14. doi :10.3390/proteomes8030014 . ISSN 2227-7382 . PMC 7564415 PMID 32640657 . Процитовано 11 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ а б Rosenberger, Florian A.; Thielert, Marvin; Mann, Matthias (2023-03). Making single-cell proteomics biologically relevant . Nature Methods (англ.) . Т. 20, № 3. с. 320—323. doi :10.1038/s41592-023-01771-9 . ISSN 1548-7105 . Процитовано 11 червня 2023 . ↑ Gstaiger, Matthias; Aebersold, Ruedi (2009-09). Applying mass spectrometry-based proteomics to genetics, genomics and network biology . Nature Reviews Genetics (англ.) . Т. 10, № 9. с. 617—627. doi :10.1038/nrg2633 . ISSN 1471-0064 . Процитовано 11 червня 2023 . ↑ а б Aebersold, Ruedi; Mann, Matthias (2016-09). Mass-spectrometric exploration of proteome structure and function . Nature (англ.) . Т. 537, № 7620. с. 347—355. doi :10.1038/nature19949 . ISSN 1476-4687 . Процитовано 11 червня 2023 . ↑ Kustatscher, Georg; Collins, Tom; Gingras, Anne-Claude; Guo, Tiannan; Hermjakob, Henning; Ideker, Trey; Lilley, Kathryn S.; Lundberg, Emma; Marcotte, Edward M. (2022-07). Understudied proteins: opportunities and challenges for functional proteomics . Nature Methods (англ.) . Т. 19, № 7. с. 774—779. doi :10.1038/s41592-022-01454-x . ISSN 1548-7105 . Процитовано 6 вересня 2023 . ↑ Van den Broeck, Lisa; Bhosale, Dinesh Kiran; Song, Kuncheng; Fonseca de Lima, Cássio Flavio; Ashley, Michael; Zhu, Tingting; Zhu, Shanshuo; Van De Cotte, Brigitte; Neyt, Pia (3 серпня 2023). Functional annotation of proteins for signaling network inference in non-model species . Nature Communications (англ.) . Т. 14, № 1. doi :10.1038/s41467-023-40365-z . ISSN 2041-1723 . Процитовано 10 вересня 2023 . ↑ Wenk, Markus R. (2005-07). The emerging field of lipidomics . Nature Reviews Drug Discovery (англ.) . Т. 4, № 7. с. 594—610. doi :10.1038/nrd1776 . ISSN 1474-1784 . Процитовано 11 червня 2023 . ↑ а б Wu, Zhuojun; Bagarolo, Giulia Ilaria; Thoröe-Boveleth, Sven; Jankowski, Joachim (1 січня 2020). “Lipidomics”: Mass spectrometric and chemometric analyses of lipids . Advanced Drug Delivery Reviews (англ.) . Т. 159. с. 294—307. doi :10.1016/j.addr.2020.06.009 . ISSN 0169-409X . Процитовано 11 червня 2023 . ↑ Han, Xianlin; Gross, Richard W. (2022-02). The foundations and development of lipidomics . Journal of Lipid Research . Т. 63, № 2. с. 100164. doi :10.1016/j.jlr.2021.100164 . ISSN 0022-2275 . PMC 8953652 PMID 34953866 . Процитовано 11 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Kyle, Jennifer E.; Stratton, Kelly G.; Zink, Erika M.; Kim, Young-Mo; Bloodsworth, Kent J.; Monroe, Matthew E.; Waters, Katrina M.; Webb-Robertson, Bobbie-Jo M.; Koeller, David M. (21 квітня 2021). A resource of lipidomics and metabolomics data from individuals with undiagnosed diseases . Scientific Data (англ.) . Т. 8, № 1. с. 114. doi :10.1038/s41597-021-00894-y . ISSN 2052-4463 . Процитовано 11 червня 2023 . ↑ Raman, Rahul; Raguram, S.; Venkataraman, Ganesh; Paulson, James C.; Sasisekharan, Ram (2005-11). Glycomics: an integrated systems approach to structure-function relationships of glycans . Nature Methods (англ.) . Т. 2, № 11. с. 817—824. doi :10.1038/nmeth807 . ISSN 1548-7105 . Процитовано 11 червня 2023 . ↑ Miyoshi, Eiji; Kamada, Yoshihiro; Suzuki, Tadashi (2020-02). Functional glycomics: Application to medical science and hepatology . Hepatology Research (англ.) . Т. 50, № 2. с. 153—164. doi :10.1111/hepr.13459 . ISSN 1386-6346 . Процитовано 11 червня 2023 . ↑ а б Park, Heajin; Jung, Jaesoo; Rodrigues, Emily; Kitova, Elena N.; Macauley, Matthew S.; Klassen, John S. (20 жовтня 2020). Mass Spectrometry-Based Shotgun Glycomics for Discovery of Natural Ligands of Glycan-Binding Proteins . Analytical Chemistry (англ.) . Т. 92, № 20. с. 14012—14020. doi :10.1021/acs.analchem.0c02931 . ISSN 0003-2700 . Процитовано 11 червня 2023 . ↑ а б de Haan, N.; Wuhrer, M.; Ruhaak, L. R. (1 листопада 2020). Mass spectrometry in clinical glycomics: The path from biomarker identification to clinical implementation . Clinical Mass Spectrometry (англ.) . Т. 18. с. 1—12. doi :10.1016/j.clinms.2020.08.001 . ISSN 2376-9998 . PMC 8600986 PMID 34820521 . Процитовано 11 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Wishart, David S. (2016-07). Emerging applications of metabolomics in drug discovery and precision medicine . Nature Reviews Drug Discovery (англ.) . Т. 15, № 7. с. 473—484. doi :10.1038/nrd.2016.32 . ISSN 1474-1784 . Процитовано 11 червня 2023 . ↑ а б Alseekh, Saleh; Aharoni, Asaph; Brotman, Yariv; Contrepois, Kévin; D’Auria, John; Ewald, Jan; C. Ewald, Jennifer; Fraser, Paul D.; Giavalisco, Patrick (2021-07). Mass spectrometry-based metabolomics: a guide for annotation, quantification and best reporting practices . Nature Methods (англ.) . Т. 18, № 7. с. 747—756. doi :10.1038/s41592-021-01197-1 . ISSN 1548-7105 . Процитовано 12 червня 2023 . ↑ Rivas, Javier De Las; Fontanillo, Celia (24 черв. 2010 р.). Protein–Protein Interactions Essentials: Key Concepts to Building and Analyzing Interactome Networks . PLOS Computational Biology (англ.) . Т. 6, № 6. с. e1000807. doi :10.1371/journal.pcbi.1000807 . ISSN 1553-7358 . PMC 2891586 PMID 20589078 . Процитовано 11 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Sharifi Tabar, Mehdi; Parsania, Chirag; Chen, Hong; Su, Xiao-Dong; Bailey, Charles G.; Rasko, John E. J. (22 серпня 2022). Illuminating the dark protein-protein interactome . Cell Reports Methods (англ.) . Т. 2, № 8. с. 100275. doi :10.1016/j.crmeth.2022.100275 . ISSN 2667-2375 . PMC 9421580 PMID 36046620 . Процитовано 11 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Choi, Soon Gang; Richardson, Aaron; Lambourne, Luke; Hill, David E.; Vidal, Marc (2018). Oñate-Sánchez, Luis (ред.). Protein Interactomics by Two-Hybrid Methods (англ.) . New York, NY: Springer. с. 1—14. doi :10.1007/978-1-4939-7871-7_1 . ISBN 978-1-4939-7871-7 PMC 6948107 PMID 29855947 . {{cite book }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Hicks, Kevin G.; Cluntun, Ahmad A.; Schubert, Heidi L.; Hackett, Sean R.; Berg, Jordan A.; Leonard, Paul G.; Ajalla Aleixo, Mariana A.; Zhou, Youjia; Bott, Alex J. (10 березня 2023). Protein-metabolite interactomics of carbohydrate metabolism reveal regulation of lactate dehydrogenase . Science (англ.) . Т. 379, № 6636. с. 996—1003. doi :10.1126/science.abm3452 . ISSN 0036-8075 . Процитовано 11 червня 2023 . ↑ Burton, Antony J.; Haugbro, Michael; Gates, Leah A.; Bagert, John D.; Allis, C. David; Muir, Tom W. (2020-06). In situ chromatin interactomics using a chemical bait and trap approach . Nature Chemistry (англ.) . Т. 12, № 6. с. 520—527. doi :10.1038/s41557-020-0474-8 . ISSN 1755-4349 . Процитовано 11 червня 2023 . ↑ Plotnikova, Olga; Baranova, Ancha; Skoblov, Mikhail (2019). Comprehensive Analysis of Human microRNA–mRNA Interactome . Frontiers in Genetics . Т. 10. doi :10.3389/fgene.2019.00933 . ISSN 1664-8021 . PMC 6792129 PMID 31649721 . Процитовано 14 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ James, Katherine; Muñoz-Muñoz, Jose (26 квітня 2022). Gilbert, Jack A. (ред.). Computational Network Inference for Bacterial Interactomics . mSystems (англ.) . Т. 7, № 2. doi :10.1128/msystems.01456-21 . ISSN 2379-5077 . PMC 9040873 PMID 35353009 . Процитовано 11 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Vidal, Marc; Cusick, Michael E.; Barabási, Albert-László (2011-03). Interactome Networks and Human Disease . Cell . Т. 144, № 6. с. 986—998. doi :10.1016/j.cell.2011.02.016 . ISSN 0092-8674 . PMC 3102045 PMID 21414488 . Процитовано 11 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Santorelli, Lucia; Caterino, Marianna; Costanzo, Michele (2022-12). Dynamic Interactomics by Cross-Linking Mass Spectrometry: Mapping the Daily Cell Life in Postgenomic Era . OMICS: A Journal of Integrative Biology . Т. 26, № 12. с. 633—649. doi :10.1089/omi.2022.0137 . Процитовано 11 червня 2023 . ↑ Nkera-Gutabara, C. K.; Kerr, R.; Scholefield, J.; Hazelhurst, S.; Naidoo, J. (2022). Microbiomics: The Next Pillar of Precision Medicine and Its Role in African Healthcare . Frontiers in Genetics . Т. 13. doi :10.3389/fgene.2022.869610 . ISSN 1664-8021 . PMC 9037082 PMID 35480328 . Процитовано 11 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Vecherskii, M. V.; Semenov, M. V.; Lisenkova, A. A.; Stepankov, A. A. (1 грудня 2021). Metagenomics: A New Direction in Ecology . Biology Bulletin (англ.) . Т. 48, № 3. с. S107—S117. doi :10.1134/S1062359022010150 . ISSN 1608-3059 . Процитовано 11 червня 2023 . ↑ Breitwieser, Florian P; Lu, Jennifer; Salzberg, Steven L (19 липня 2019). A review of methods and databases for metagenomic classification and assembly . Briefings in Bioinformatics (англ.) . Т. 20, № 4. с. 1125—1136. doi :10.1093/bib/bbx120 . ISSN 1467-5463 . PMC 6781581 PMID 29028872 . Процитовано 11 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Handelsman, Jo (2004-12). Metagenomics: Application of Genomics to Uncultured Microorganisms . Microbiology and Molecular Biology Reviews (англ.) . Т. 68, № 4. с. 669—685. doi :10.1128/MMBR.68.4.669-685.2004 . ISSN 1092-2172 . PMC 539003 PMID 15590779 . Процитовано 11 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Chiu, Charles Y.; Miller, Steven A. (2019-06). Clinical metagenomics . Nature Reviews Genetics (англ.) . Т. 20, № 6. с. 341—355. doi :10.1038/s41576-019-0113-7 . ISSN 1471-0064 . PMC 6858796 PMID 30918369 . Процитовано 11 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Shakya, Migun; Lo, Chien-Chi; Chain, Patrick S. G. (25 вересня 2019). Advances and Challenges in Metatranscriptomic Analysis . Frontiers in Genetics . Т. 10. doi :10.3389/fgene.2019.00904 . ISSN 1664-8021 . PMC 6774269 PMID 31608125 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Terrón-Camero, Laura C.; Gordillo-González, Fernando; Salas-Espejo, Eduardo; Andrés-León, Eduardo (2022-12). Comparison of Metagenomics and Metatranscriptomics Tools: A Guide to Making the Right Choice . Genes (англ.) . Т. 13, № 12. с. 2280. doi :10.3390/genes13122280 . ISSN 2073-4425 . PMC 9777648 PMID 36553546 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Van Den Bossche, Tim; Arntzen, Magnus Ø.; Becher, Dörte; Benndorf, Dirk; Eijsink, Vincent G. H.; Henry, Céline; Jagtap, Pratik D.; Jehmlich, Nico; Juste, Catherine (2021-12). The Metaproteomics Initiative: a coordinated approach for propelling the functional characterization of microbiomes . Microbiome (англ.) . Т. 9, № 1. doi :10.1186/s40168-021-01176-w . ISSN 2049-2618 . PMC 8690404 PMID 34930457 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Stamboulian, Moses; Canderan, Jamie; Ye, Yuzhen (18 бер. 2022 р.). Metaproteomics as a tool for studying the protein landscape of human-gut bacterial species . PLOS Computational Biology (англ.) . Т. 18, № 3. с. e1009397. doi :10.1371/journal.pcbi.1009397 . ISSN 1553-7358 . PMC 8967034 PMID 35302987 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Henry, Céline; Bassignani, Ariane; Berland, Magali; Langella, Olivier; Sokol, Harry; Juste, Catherine (2022-01). Modern Metaproteomics: A Unique Tool to Characterize the Active Microbiome in Health and Diseases, and Pave the Road towards New Biomarkers—Example of Crohn’s Disease and Ulcerative Colitis Flare-Ups . Cells (англ.) . Т. 11, № 8. с. 1340. doi :10.3390/cells11081340 . ISSN 2073-4409 . PMC 9028112 PMID 35456018 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Wang, Xinkun (6 липня 2023). Next-Generation Sequencing Data Analysis doi :10.1201/9780429329180 . ISBN 978-0-429-32918-0 ↑ Mardis, Elaine R. (1 вересня 2008). Next-Generation DNA Sequencing Methods . Annual Review of Genomics and Human Genetics (англ.) . Т. 9, № 1. с. 387—402. doi :10.1146/annurev.genom.9.081307.164359 . ISSN 1527-8204 . Процитовано 12 червня 2023 . ↑ Shendure, Jay; Ji, Hanlee (2008-10). Next-generation DNA sequencing . Nature Biotechnology (англ.) . Т. 26, № 10. с. 1135—1145. doi :10.1038/nbt1486 . ISSN 1546-1696 . Процитовано 12 червня 2023 . ↑ Next-Generation Sequencing (NGS) | Explore the technology . www.illumina.com (англ.) . Процитовано 12 червня 2023 .↑ An Overview of Next-Generation Sequencing . Genomics Research from Technology Networks (англ.) . Процитовано 12 червня 2023 .↑ Cheng, Chu; Fei, Zhongjie; Xiao, Pengfeng (2023). Methods to improve the accuracy of next-generation sequencing . Frontiers in Bioengineering and Biotechnology . Т. 11. doi :10.3389/fbioe.2023.982111 . ISSN 2296-4185 . PMC 9895957 PMID 36741756 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Pervez, Muhammad Tariq; Hasnain, Mirza Jawad ul; Abbas, Syed Hassan; Moustafa, Mahmoud F.; Aslam, Naeem; Shah, Syed Shah Muhammad (29 вересня 2022). A Comprehensive Review of Performance of Next-Generation Sequencing Platforms . BioMed Research International (англ.) . Т. 2022. с. e3457806. doi :10.1155/2022/3457806 . ISSN 2314-6133 . PMC 9537002 PMID 36212714 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Schena, Mark; Shalon, Dari; Davis, Ronald W.; Brown, Patrick O. (20 жовтня 1995). Quantitative Monitoring of Gene Expression Patterns with a Complementary DNA Microarray . Science (англ.) . Т. 270, № 5235. с. 467—470. doi :10.1126/science.270.5235.467 . ISSN 0036-8075 . Процитовано 12 червня 2023 . ↑ Lehrach, Hans (9 серпня 2013). Faculty Opinions recommendation of RNA-Seq: a revolutionary tool for transcriptomics . Faculty Opinions – Post-Publication Peer Review of the Biomedical Literature . doi :10.3410/f.1128830.793481779 . Процитовано 12 червня 2023 . ↑ Bersanelli, Matteo; Mosca, Ettore; Remondini, Daniel; Giampieri, Enrico; Sala, Claudia; Castellani, Gastone; Milanesi, Luciano (20 січня 2016). Methods for the integration of multi-omics data: mathematical aspects . BMC Bioinformatics . Т. 17, № 2. с. S15. doi :10.1186/s12859-015-0857-9 . ISSN 1471-2105 . PMC 4959355 PMID 26821531 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Vasaikar, Suhas V.; Savage, Adam K.; Gong, Qiuyu; Swanson, Elliott; Talla, Aarthi; Lord, Cara; Heubeck, Alexander T.; Reading, Julian; Graybuck, Lucas T. (27 березня 2023). A comprehensive platform for analyzing longitudinal multi-omics data . Nature Communications (англ.) . Т. 14, № 1. с. 1684. doi :10.1038/s41467-023-37432-w . ISSN 2041-1723 . Процитовано 12 червня 2023 . ↑ Zhou, Guangyan; Pang, Zhiqiang; Lu, Yao; Ewald, Jessica; Xia, Jianguo (5 липня 2022). OmicsNet 2.0: a web-based platform for multi-omics integration and network visual analytics . Nucleic Acids Research (англ.) . Т. 50, № W1. с. W527—W533. doi :10.1093/nar/gkac376 . ISSN 0305-1048 . PMC 9252810 PMID 35639733 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Conesa, Ana; Beck, Stephan (31 жовтня 2019). Making multi-omics data accessible to researchers . Scientific Data (англ.) . Т. 6, № 1. с. 251. doi :10.1038/s41597-019-0258-4 . ISSN 2052-4463 . Процитовано 12 червня 2023 . ↑ Arjmand, Babak; Hamidpour, Shayesteh Kokabi; Tayanloo-Beik, Akram; Goodarzi, Parisa; Aghayan, Hamid Reza; Adibi, Hossein; Larijani, Bagher (2022). Machine Learning: A New Prospect in Multi-Omics Data Analysis of Cancer . Frontiers in Genetics . Т. 13. doi :10.3389/fgene.2022.824451 . ISSN 1664-8021 . PMC 8829119 PMID 35154283 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Garmire, Lana X.; Chaudhary, Kumardeep; Huang, Sijia (2017). More Is Better: Recent Progress in Multi-Omics Data Integration Methods . Frontiers in Genetics (English) . 8 : 84. doi :10.3389/fgene.2017.00084 ISSN 1664-8021 . PMC 5472696 PMID 28670325 . ↑ Tagkopoulos, Ilias; Kim, Minseung (2018). Data integration and predictive modeling methods for multi-omics datasets. Molecular Omics . 14 (1): 8—25. doi :10.1039/C7MO00051K . PMID 29725673 . ↑ Lin, Eugene; Lane, Hsien-Yuan (20 січня 2017). Machine learning and systems genomics approaches for multi-omics data . Biomarker Research . 5 (1): 2. doi :10.1186/s40364-017-0082-y . ISSN 2050-7771 . PMC 5251341 PMID 28127429 . {{cite journal }}: Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ а б Rohart, Florian; Gautier, Benoît; Singh, Amrit; Lê Cao, Kim-Anh (14 лютого 2017). mixOmics: an R package for 'omics feature selection and multiple data integration . PLOS Computational Biology . 13 (11). doi :10.1371/journal.pcbi.1005752 . PMC 5687754 PMID 29099853 . {{cite journal }}: Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Han, Jing-Dong Jackie (5 вересня 2018). Faculty of 1000 evaluation for Single-Cell Multiomics: Multiple Measurements from Single Cells . Trends in Genetics . 33 (2). doi :10.3410/f.727213649.793550351 . ↑ Hu, Youjin; An, Qin; Sheu, Katherine; Trejo, Brandon; Fan, Shuxin; Guo, Ying (20 квітня 2018). Single Cell Multi-Omics Technology: Methodology and Application . Frontiers in Cell and Developmental Biology . 6 : 28. doi :10.3389/fcell.2018.00028 ISSN 2296-634X . PMC 5919954 PMID 29732369 . ↑ A focus on single-cell omics . Nature Reviews Genetics (англ.) . Т. 24, № 8. 2023-08. с. 485—485. doi :10.1038/s41576-023-00628-3 . ISSN 1471-0064 . Процитовано 6 серпня 2023 .↑ Baysoy, Alev; Bai, Zhiliang; Satija, Rahul; Fan, Rong (2023-10). The technological landscape and applications of single-cell multi-omics . Nature Reviews Molecular Cell Biology (англ.) . Т. 24, № 10. с. 695—713. doi :10.1038/s41580-023-00615-w . ISSN 1471-0080 . Процитовано 8 грудня 2023 . ↑ Vandereyken, Katy; Sifrim, Alejandro; Thienpont, Bernard; Voet, Thierry (2 березня 2023). Methods and applications for single-cell and spatial multi-omics . Nature Reviews Genetics (англ.) . с. 1—22. doi :10.1038/s41576-023-00580-2 . ISSN 1471-0064 . Процитовано 11 червня 2023 . ↑ Kester, Lennart Spanjaard, Bastiaan Bienko, Magda van Oudenaarden, Alexander Dey, Siddharth S (2015). Integrated genome and transcriptome sequencing of the same cell . Nature Biotechnology . 33 (3): 285—289. doi :10.1038/nbt.3129 . OCLC 931063996 . PMC 4374170 PMID 25599178 . ↑ Macaulay, Iain C; Teng, Mabel J; Haerty, Wilfried; Kumar, Parveen; Ponting, Chris P; Voet, Thierry (29 вересня 2016). Separation and parallel sequencing of the genomes and transcriptomes of single cells using G&T-seq . Nature Protocols . 11 (11): 2081—2103. doi :10.1038/nprot.2016.138 . ISSN 1754-2189 . PMID 27685099 . ↑ Tang, Fuchou; Wen, Lu; Li, Xianlong; Wu, Xinglong; Zhu, Ping; Guo, Hongshan (1 грудня 2013). Single-cell methylome landscapes of mouse embryonic stem cells and early embryos analyzed using reduced representation bisulfite sequencing . Genome Research . 23 (12): 2126—2135. doi :10.1101/gr.161679.113 . ISSN 1088-9051 . PMC 3847781 PMID 24179143 . ↑ Kelsey, Gavin; Reik, Wolf; Stegle, Oliver; Andrews, Simon R.; Julian Peat; Saadeh, Heba; Krueger, Felix; Angermueller, Christof; Lee, Heather J. (August 2014). Single-cell genome-wide bisulfite sequencing for assessing epigenetic heterogeneity . Nature Methods . 11 (8): 817—820. doi :10.1038/nmeth.3035 . ISSN 1548-7105 . PMC 4117646 PMID 25042786 . ↑ Angermueller, Christof; Clark, Stephen J; Lee, Heather J; Macaulay, Iain C; Teng, Mabel J; Hu, Tim Xiaoming; Krueger, Felix; Smallwood, Sébastien A; Ponting, Chris P (11 січня 2016). Parallel single-cell sequencing links transcriptional and epigenetic heterogeneity . Nature Methods . 13 (3): 229—232. doi :10.1038/nmeth.3728 . ISSN 1548-7091 . PMC 4770512 PMID 26752769 . ↑ Greenleaf, William J.; Chang, Howard Y.; Snyder, Michael P.; Michael L. Gonzales; Ruff, Dave; Litzenburger, Ulrike M.; Wu, Beijing; Buenrostro, Jason D. (July 2015). Single-cell chromatin accessibility reveals principles of regulatory variation . Nature . 523 (7561): 486—490. Bibcode :2015Natur.523..486B . doi :10.1038/nature14590 . ISSN 1476-4687 . PMC 4685948 PMID 26083756 . ↑ Fraser, Peter; Tanay, Amos; Laue, Ernest D.; Dean, Wendy; Yaffe, Eitan; Schoenfelder, Stefan; Stevens, Tim J.; Lubling, Yaniv; Nagano, Takashi (October 2013). Single-cell Hi-C reveals cell-to-cell variability in chromosome structure . Nature . 502 (7469): 59—64. Bibcode :2013Natur.502...59N . doi :10.1038/nature12593 . ISSN 1476-4687 . PMC 3869051 PMID 24067610 . ↑ а б Darmanis, Spyros; Gallant, Caroline Julie; Marinescu, Voichita Dana; Niklasson, Mia; Segerman, Anna; Flamourakis, Georgios; Fredriksson, Simon; Assarsson, Erika; Lundberg, Martin (12 січня 2016). Simultaneous Multiplexed Measurement of RNA and Proteins in Single Cells . Cell Reports . 14 (2): 380—389. doi :10.1016/j.celrep.2015.12.021 . ISSN 2211-1247 . PMC 4713867 PMID 26748716 . ↑ а б Gherardini, Pier Federico; Nolan, Garry P.; Chen, Shih-Yu; Hsieh, Elena W. Y.; Zunder, Eli R.; Bava, Felice-Alessio; Frei, Andreas P. (March 2016). Highly multiplexed simultaneous detection of RNAs and proteins in single cells . Nature Methods . 13 (3): 269—275. doi :10.1038/nmeth.3742 . ISSN 1548-7105 . PMC 4767631 PMID 26808670 . ↑ Assarsson, Erika; Lundberg, Martin; Holmquist, Göran; Björkesten, Johan; Bucht Thorsen, Stine; Ekman, Daniel; Eriksson, Anna; Rennel Dickens, Emma; Ohlsson, Sandra (22 квітня 2014). Homogenous 96-Plex PEA Immunoassay Exhibiting High Sensitivity, Specificity, and Excellent Scalability . PLOS ONE . 9 (4): e95192. Bibcode :2014PLoSO...995192A . doi :10.1371/journal.pone.0095192 ISSN 1932-6203 . PMC 3995906 PMID 24755770 . ↑ Razzaq, Muhammad Khuram; Aleem, Muqadas; Mansoor, Shahid; Khan, Mueen Alam; Rauf, Saeed; Iqbal, Shahid; Siddique, Kadambot H. M. (2021-01). Omics and CRISPR-Cas9 Approaches for Molecular Insight, Functional Gene Analysis, and Stress Tolerance Development in Crops . International Journal of Molecular Sciences (англ.) . Т. 22, № 3. с. 1292. doi :10.3390/ijms22031292 . ISSN 1422-0067 . PMC 7866018 PMID 33525517 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Hilton, Isaac B.; D'Ippolito, Anthony M.; Vockley, Christopher M.; Thakore, Pratiksha I.; Crawford, Gregory E.; Reddy, Timothy E.; Gersbach, Charles A. (2015-05). Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers . Nature Biotechnology (англ.) . Т. 33, № 5. с. 510—517. doi :10.1038/nbt.3199 . ISSN 1546-1696 . PMC 4430400 PMID 25849900 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Shin, Hosub; Choi, Woo Lee; Lim, Joo Young; Huh, Jin Hoe (1 березня 2022). Epigenome editing: targeted manipulation of epigenetic modifications in plants . Genes & Genomics (англ.) . Т. 44, № 3. с. 307—315. doi :10.1007/s13258-021-01199-5 . ISSN 2092-9293 . Процитовано 12 червня 2023 . ↑ а б в г Athieniti, Efi; Spyrou, George M. (1 січня 2023). A guide to multi-omics data collection and integration for translational medicine . Computational and Structural Biotechnology Journal (англ.) . Т. 21. с. 134—149. doi :10.1016/j.csbj.2022.11.050 . ISSN 2001-0370 . PMC 9747357 PMID 36544480 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Cao, Zhi-Jie; Gao, Ge (2022-10). Multi-omics single-cell data integration and regulatory inference with graph-linked embedding . Nature Biotechnology (англ.) . Т. 40, № 10. с. 1458—1466. doi :10.1038/s41587-022-01284-4 . ISSN 1546-1696 . Процитовано 12 червня 2023 . ↑ а б Vahabi, Nasim; Michailidis, George (2022). Unsupervised Multi-Omics Data Integration Methods: A Comprehensive Review . Frontiers in Genetics . Т. 13. doi :10.3389/fgene.2022.854752 . ISSN 1664-8021 . PMC 8981526 PMID 35391796 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ а б Flores, Javier E.; Claborne, Daniel M.; Weller, Zachary D.; Webb-Robertson, Bobbie-Jo M.; Waters, Katrina M.; Bramer, Lisa M. (2023). Missing data in multi-omics integration: Recent advances through artificial intelligence . Frontiers in Artificial Intelligence . Т. 6. doi :10.3389/frai.2023.1098308 . ISSN 2624-8212 . PMC 9949722 PMID 36844425 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Lazar, C.; Meganck, S.; Taminau, J.; Steenhoff, D.; Coletta, A.; Molter, C.; Weiss-Solis, D. Y.; Duque, R.; Bersini, H. (1 липня 2013). Batch effect removal methods for microarray gene expression data integration: a survey . Briefings in Bioinformatics (англ.) . Т. 14, № 4. с. 469—490. doi :10.1093/bib/bbs037 . ISSN 1467-5463 . Процитовано 12 червня 2023 . ↑ Hutchison, William J; Keyes, Timothy J; Crowell, Helena L; Soneson, Charlotte; Mu, Wancen; Park, Ji-Eun; Davis, Eric S; Nahid, Abdullah A; Tang, Ming (13 вересня 2023). The tidyomics ecosystem: Enhancing omic data analyses (англ.) . doi :10.1101/2023.09.10.557072 . Процитовано 15 вересня 2023 . ↑ Donati, Stefano; Mattanovich, Matthias; Hjort, Pernille; Jacobsen, Simo Abdessamad Baallal; Blomquist, Sarah Dina; Mangaard, Drude; Gurdo, Nicolas; Pastor, Felix Pacheco; Maury, Jérôme (19 травня 2023). An automated workflow for multi-omics screening of microbial model organisms . npj Systems Biology and Applications (англ.) . Т. 9, № 1. с. 1—12. doi :10.1038/s41540-023-00277-6 . ISSN 2056-7189 . Процитовано 15 вересня 2023 . ↑ Velten, Britta; Stegle, Oliver (14 вересня 2023). Principles and challenges of modeling temporal and spatial omics data . Nature Methods (англ.) . с. 1—13. doi :10.1038/s41592-023-01992-y . ISSN 1548-7105 . Процитовано 15 вересня 2023 . ↑ Hasin, Yehudit; Seldin, Marcus; Lusis, Aldons (5 травня 2017). Multi-omics approaches to disease . Genome Biology . Т. 18, № 1. с. 83. doi :10.1186/s13059-017-1215-1 . ISSN 1474-760X . PMC 5418815 PMID 28476144 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Khan, Mohd M.; Ernst, Orna; Manes, Nathan P.; Oyler, Benjamin L.; Fraser, Iain D. C.; Goodlett, David R.; Nita-Lazar, Aleksandra (11 березня 2019). Multi-Omics Strategies Uncover Host–Pathogen Interactions. ACS Infectious Diseases . 5 (4): 493—505. doi :10.1021/acsinfecdis.9b00080 . ISSN 2373-8227 . PMID 30857388 . ↑ Aderem, Alan; Adkins, Joshua N.; Ansong, Charles; Galagan, James; Kaiser, Shari; Korth, Marcus J.; Law, G. Lynn; McDermott, Jason G.; Proll, Sean C. (1 лютого 2011). A Systems Biology Approach to Infectious Disease Research: Innovating the Pathogen-Host Research Paradigm . mBio . 2 (1): e00325-10. doi :10.1128/mbio.00325-10 . ISSN 2150-7511 . PMC 3034460 PMID 21285433 . ↑ Mouchtouris, N; Smit, RD; Piper, K; Prashant, G; Evans, JJ; Karsy, M (4 березня 2022). A review of multiomics platforms in pituitary adenoma pathogenesis. Frontiers in Bioscience (Landmark Edition) . 27 (3): 77. doi :10.31083/j.fbl2703077 . PMID 35345309 . ↑ Yan, Jingwen; Risacher, Shannon L; Shen, Li; Saykin, Andrew J. (30 червня 2017). Network approaches to systems biology analysis of complex disease: integrative methods for multi-omics data . Briefings in Bioinformatics . 19 (6): 1370—1381. doi :10.1093/bib/bbx066 . ISSN 1467-5463 . PMC 6454489 PMID 28679163 . ↑ Yoon, Jong; Villa, Chiara, ред. (13 грудня 2021). Multi-Omics for the Understanding of Brain Diseases doi :10.3390/books978-3-0365-2603-4 . ISBN 978-3-0365-2602-7 {{cite book }}: Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ а б Mavromatis, Lucas A.; Rosoff, Daniel B.; Bell, Andrew S.; Jung, Jeesun; Wagner, Josephin; Lohoff, Falk W. (19 квітня 2023). Multi-omic underpinnings of epigenetic aging and human longevity . Nature Communications (англ.) . Т. 14, № 1. с. 2236. doi :10.1038/s41467-023-37729-w . ISSN 2041-1723 . PMC 10115892 PMID 37076473 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Zhong, Yating; Peng, Yuzhong; Lin, Yanmei; Chen, Dingjia; Zhang, Hao; Zheng, Wen; Chen, Yuanyuan; Wu, Changliang (5 травня 2023). MODILM: towards better complex diseases classification using a novel multi-omics data integration learning model . BMC Medical Informatics and Decision Making (англ.) . Т. 23, № 1. doi :10.1186/s12911-023-02173-9 . ISSN 1472-6947 . PMC 10161645 PMID 37147619 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Dar, Mashooq Ahmad; Arafah, Azher; Bhat, Kaisar Ahmad; Khan, Andleeb; Khan, Mosin Saleem; Ali, Aarif; Ahmad, Syed Mudasir; Rashid, Shahzada Mudasir; Rehman, Muneeb U (13 квітня 2023). Multiomics technologies: role in disease biomarker discoveries and therapeutics . Briefings in Functional Genomics (англ.) . Т. 22, № 2. с. 76—96. doi :10.1093/bfgp/elac017 . ISSN 2041-2649 . Процитовано 12 червня 2023 . ↑ Babu, Mohan; Snyder, Michael (2023-06). Multi-Omics Profiling for Health . Molecular & Cellular Proteomics . Т. 22, № 6. с. 100561. doi :10.1016/j.mcpro.2023.100561 . ISSN 1535-9476 . PMC 10220275 PMID 37119971 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Paananen, Jussi; Fortino, Vittorio (1 грудня 2020). An omics perspective on drug target discovery platforms . Briefings in Bioinformatics (англ.) . Т. 21, № 6. с. 1937—1953. doi :10.1093/bib/bbz122 . ISSN 1467-5463 . PMC 7711264 PMID 31774113 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Guo, Shicheng; Zhang, Dake; Wang, Hu; An, Qin; Yu, Guangchuang; Han, Junwei; Jiang, Chunjie; Huang, Jianfeng (21 лютого 2023). Editorial: Computational and systematic analysis of multi-omics data for drug discovery and development . Frontiers in Medicine . Т. 10. doi :10.3389/fmed.2023.1146896 . ISSN 2296-858X . PMC 9989304 PMID 36895719 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Liu, Xiao-Ying; Mei, Xin-Yue (2023). Prediction of drug sensitivity based on multi-omics data using deep learning and similarity network fusion approaches . Frontiers in Bioengineering and Biotechnology . Т. 11. doi :10.3389/fbioe.2023.1156372 . ISSN 2296-4185 . PMC 10150883 PMID 37139048 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Wang, Conghao; Lye, Xintong; Kaalia, Rama; Kumar, Parvin; Rajapakse, Jagath C. (28 листопада 2022). Deep learning and multi-omics approach to predict drug responses in cancer . BMC Bioinformatics (англ.) . Т. 22, № S10. doi :10.1186/s12859-022-04964-9 . ISSN 1471-2105 . PMC 9703655 PMID 36443676 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Proctor, Lita M.; Creasy, Heather H.; Fettweis, Jennifer M.; Lloyd-Price, Jason; Mahurkar, Anup; Zhou, Wenyu; Buck, Gregory A.; Snyder, Michael P.; Strauss, Jerome F. (May 2019). The Integrative Human Microbiome Project . Nature . 569 (7758): 641—648. Bibcode :2019Natur.569..641I . doi :10.1038/s41586-019-1238-8 . ISSN 1476-4687 . PMC 6784865 PMID 31142853 . ↑ After the Integrative Human Microbiome Project, what's next for the microbiome community? . Nature . 569 (7758): 599. 29 травня 2019. Bibcode :2019Natur.569Q.599. . doi :10.1038/d41586-019-01674-w PMID 31142868 .↑ Snyder, Michael; Weinstock, George M.; Sodergren, Erica; McLaughlin, Tracey; Tse, David; Rost, Hannes; Piening, Brian; Kukurba, Kim; Rose, Sophia Miryam Schüssler-Fiorenza (May 2019). Longitudinal multi-omics of host–microbe dynamics in prediabetes . Nature . 569 (7758): 663—671. Bibcode :2019Natur.569..663Z . doi :10.1038/s41586-019-1236-x . ISSN 1476-4687 . PMC 6666404 PMID 31142858 . ↑ Huttenhower, Curtis; Xavier, Ramnik J.; Vlamakis, Hera; Franzosa, Eric A.; Clish, Clary B.; Winter, Harland S.; Stappenbeck, Thaddeus S.; Petrosino, Joseph F.; McGovern, Dermot P. B. (May 2019). Multi-omics of the gut microbial ecosystem in inflammatory bowel diseases . Nature . 569 (7758): 655—662. Bibcode :2019Natur.569..655L . doi :10.1038/s41586-019-1237-9 . ISSN 1476-4687 . PMC 6650278 PMID 31142855 . ↑ Buck, Gregory A.; Strauss, Jerome F.; Jefferson, Kimberly K.; Hendricks-Muñoz, Karen D.; Wijesooriya, N. Romesh; Rubens, Craig E.; Gravett, Michael G.; Sexton, Amber L.; Chaffin, Donald O. (June 2019). The vaginal microbiome and preterm birth . Nature Medicine . 25 (6): 1012—1021. doi :10.1038/s41591-019-0450-2 . ISSN 1546-170X . PMC 6750801 PMID 31142849 . ↑ Chu, Xiaojing; Zhang, Bowen; Koeken, Valerie A. C. M.; Gupta, Manoj Kumar; Li, Yang (2021). Multi-Omics Approaches in Immunological Research . Frontiers in Immunology . Т. 12. doi :10.3389/fimmu.2021.668045 . ISSN 1664-3224 . PMC 8226116 PMID 34177908 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Bonaguro, Lorenzo; Schulte-Schrepping, Jonas; Ulas, Thomas; Aschenbrenner, Anna C.; Beyer, Marc; Schultze, Joachim L. (2022-10). A guide to systems-level immunomics . Nature Immunology (англ.) . Т. 23, № 10. с. 1412—1423. doi :10.1038/s41590-022-01309-9 . ISSN 1529-2916 . Процитовано 12 червня 2023 . ↑ Ota, Mineto; Fujio, Keishi (2021-12). Multi-omics approach to precision medicine for immune-mediated diseases . Inflammation and Regeneration (англ.) . Т. 41, № 1. doi :10.1186/s41232-021-00173-8 . ISSN 1880-8190 . PMC 8325815 PMID 34332645 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Kidd, Brian A; Peters, Lauren A; Schadt, Eric E; Dudley, Joel T (21 січня 2014). Unifying immunology with informatics and multiscale biology . Nature Immunology . 15 (2): 118—127. doi :10.1038/ni.2787 . ISSN 1529-2908 . PMC 4345400 PMID 24448569 . ↑ Harris, Eva; Kasarskis, Andrew; Wolinsky, Steven M.; Suaréz‐Fariñas, Mayte; Zhu, Jun; Wang, Li; Balmaseda, Angel; Thomas, Guajira P.; Stewart, Michael G. (1 серпня 2018). Comprehensive innate immune profiling of chikungunya virus infection in pediatric cases . Molecular Systems Biology . 14 (8): e7862. doi :10.15252/msb.20177862 . ISSN 1744-4292 . PMC 6110311 PMID 30150281 . ↑ Firestein, Gary S.; Wang, Wei; Gay, Steffen; Ball, Scott T.; Bartok, Beatrix; Boyle, David L.; Whitaker, John W. (22 квітня 2015). Integrative Omics Analysis of Rheumatoid Arthritis Identifies Non-Obvious Therapeutic Targets . PLOS ONE . 10 (4): e0124254. Bibcode :2015PLoSO..1024254W . doi :10.1371/journal.pone.0124254 ISSN 1932-6203 . PMC 4406750 PMID 25901943 . ↑ Pulendran, Bali; Li, Shuzhao; Nakaya, Helder I. (29 жовтня 2010). Systems Vaccinology . Immunity . 33 (4): 516—529. doi :10.1016/j.immuni.2010.10.006 . ISSN 1074-7613 . PMC 3001343 PMID 21029962 . ↑ Li, Shuzhao; Sullivan, Nicole L.; Rouphael, Nadine; Yu, Tianwei; Banton, Sophia; Maddur, Mohan S.; McCausland, Megan; Chiu, Christopher; Canniff, Jennifer (18 травня 2017). Metabolic Phenotypes of Response to Vaccination in Humans . Cell . 169 (5): 862—877.e17. doi :10.1016/j.cell.2017.04.026 . ISSN 0092-8674 . PMC 5711477 PMID 28502771 . ↑ Aging Atlas Consortium; Liu, Guang-Hui; Bao, Yiming; Qu, Jing; Zhang, Weiqi; Zhang, Tao; Kang, Wang; Yang, Fei; Ji, Qianzhao (8 січня 2021). Aging Atlas: a multi-omics database for aging biology . Nucleic Acids Research (англ.) . Т. 49, № D1. с. D825—D830. doi :10.1093/nar/gkaa894 . ISSN 0305-1048 . PMC 7779027 PMID 33119753 . Процитовано 14 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Zhang, Bohan; Lee, David E.; Trapp, Alexandre; Tyshkovskiy, Alexander; Lu, Ake T.; Bareja, Akshay; Kerepesi, Csaba; McKay, Lauren K.; Shindyapina, Anastasia V. (27 липня 2023). Multi-omic rejuvenation and life span extension on exposure to youthful circulation . Nature Aging (англ.) . с. 1—17. doi :10.1038/s43587-023-00451-9 . ISSN 2662-8465 . Процитовано 5 серпня 2023 . ↑ Mavromatis, Lucas A.; Rosoff, Daniel B.; Bell, Andrew S.; Jung, Jeesun; Wagner, Josephin; Lohoff, Falk W. (19 квітня 2023). Multi-omic underpinnings of epigenetic aging and human longevity . Nature Communications (англ.) . Т. 14, № 1. с. 2236. doi :10.1038/s41467-023-37729-w . ISSN 2041-1723 . PMC 10115892 PMID 37076473 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Mahmood, Umer; Li, Xiaodong; Fan, Yonghai; Chang, Wei; Niu, Yue; Li, Jiana; Qu, Cunmin; Lu, Kun (2022). Multi-omics revolution to promote plant breeding efficiency . Frontiers in Plant Science . Т. 13. doi :10.3389/fpls.2022.1062952 . ISSN 1664-462X . PMC 9773847 PMID 36570904 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Yang, Yaodong; Saand, Mumtaz Ali; Huang, Liyun; Abdelaal, Walid Badawy; Zhang, Jun; Wu, Yi; Li, Jing; Sirohi, Muzafar Hussain; Wang, Fuyou (2021). Applications of Multi-Omics Technologies for Crop Improvement . Frontiers in Plant Science . Т. 12. doi :10.3389/fpls.2021.563953 . ISSN 1664-462X . PMC 8446515 PMID 34539683 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Sirangelo, Tiziana Maria; Ludlow, Richard Andrew; Chenet, Tatiana; Pasti, Luisa; Spadafora, Natasha Damiana (2023-04). Multi-Omics and Genome Editing Studies on Plant Cell Walls to Improve Biomass Quality . Agriculture (англ.) . Т. 13, № 4. с. 752. doi :10.3390/agriculture13040752 . ISSN 2077-0472 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Ichihashi, Yasunori; Date, Yasuhiro; Shino, Amiu; Shimizu, Tomoko; Shibata, Arisa; Kumaishi, Kie; Funahashi, Fumiaki; Wakayama, Kenji; Yamazaki, Kohei (23 червня 2020). Multi-omics analysis on an agroecosystem reveals the significant role of organic nitrogen to increase agricultural crop yield . Proceedings of the National Academy of Sciences (англ.) . Т. 117, № 25. с. 14552—14560. doi :10.1073/pnas.1917259117 . ISSN 0027-8424 . PMC 7321985 PMID 32513689 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Aguiar-Pulido, Vanessa; Huang, Wenrui; Suarez-Ulloa, Victoria; Cickovski, Trevor; Mathee, Kalai; Narasimhan, Giri (2016-01). Metagenomics, Metatranscriptomics, and Metabolomics Approaches for Microbiome Analysis: Supplementary Issue: Bioinformatics Methods and Applications for Big Metagenomics Data . Evolutionary Bioinformatics (англ.) . Т. 12s1. с. EBO.S36436. doi :10.4137/EBO.S36436 . ISSN 1176-9343 . PMC 4869604 PMID 27199545 . Процитовано 12 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання )↑ Joshi, Samiksha; Gangola, Saurabh; Rani, Anju; Sahgal, Manvika; Tewari, Samarth; Bhandari, Narendra Singh; Jaggi, Vandana; Prakash, Om (1 січня 2023). Gangola, Saurabh; Kumar, Saurabh; Joshi, Samiksha; Bhatt, Pankaj (ред.). Chapter 1 - Recent molecular and omics approaches to study rhizosphere functioning (англ.) . Academic Press. с. 1—13. doi :10.1016/b978-0-323-95090-9.00009-1 . ISBN 978-0-323-95090-9 ↑ Herold, Malte; Martínez Arbas, Susana; Narayanasamy, Shaman; Sheik, Abdul R.; Kleine-Borgmann, Luise A. K.; Lebrun, Laura A.; Kunath, Benoît J.; Roume, Hugo; Bessarab, Irina (19 жовтня 2020). Integration of time-series meta-omics data reveals how microbial ecosystems respond to disturbance . Nature Communications (англ.) . Т. 11, № 1. с. 5281. doi :10.1038/s41467-020-19006-2 . ISSN 2041-1723 . Процитовано 12 червня 2023 . ↑ McDaniel, Elizabeth A.; Wahl, Sebastian Aljoscha; Ishii, Shun'ichi; Pinto, Ameet; Ziels, Ryan; Nielsen, Per Halkjær; McMahon, Katherine D.; Williams, Rohan B. H. (15 жовтня 2021). Prospects for multi-omics in the microbial ecology of water engineering . Water Research (англ.) . Т. 205. с. 117608. doi :10.1016/j.watres.2021.117608 . ISSN 0043-1354 . Процитовано 12 червня 2023 . ↑ Meng, Chen; Kuster, Bernhard; Culhane, Aedín C; Gholami, Amin (2014). A multivariate approach to the integration of multi-omics datasets . BMC Bioinformatics . 15 (1): 162. doi :10.1186/1471-2105-15-162 . ISSN 1471-2105 . PMC 4053266 PMID 24884486 . {{cite journal }}: Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Ramos, Marcel; Schiffer, Lucas; Re, Angela; Azhar, Rimsha; Basunia, Azfar; Rodriguez, Carmen; Chan, Tiffany; Chapman, Phil; Davis, Sean R. (1 листопада 2017). Software for the Integration of Multiomics Experiments in Bioconductor . Cancer Research . 77 (21): e39—e42. doi :10.1158/0008-5472.CAN-17-0344 . PMC 5679241 PMID 29092936 . ↑ Seonggyun Han, Younghee Lee (2017), IMAS doi :10.18129/b9.bioc.imas , процитовано 28 червня 2019 ↑ Karim Mezhoud [Aut, Cre] (2017), bioCancer doi :10.18129/b9.bioc.biocancer , процитовано 28 червня 2019 ↑ Hernandez-Ferrer, Carles; Ruiz-Arenas, Carlos; Beltran-Gomila, Alba; González, Juan R. (17 січня 2017). MultiDataSet: an R package for encapsulating multiple data sets with application to omic data integration . BMC Bioinformatics . 18 (1): 36. doi :10.1186/s12859-016-1455-1 . ISSN 1471-2105 . PMC 5240259 PMID 28095799 . {{cite journal }}: Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Reap the rewards of a biological insight engine . omicX . Процитовано 26 червня 2019 .↑ Conesa, Ana; Dopazo, Joaquín; García-López, Federico; García-Alcalde, Fernando (1 січня 2011). Paintomics: a web based tool for the joint visualization of transcriptomics and metabolomics data . Bioinformatics . 27 (1): 137—139. doi :10.1093/bioinformatics/btq594 . ISSN 1367-4803 . PMC 3008637 PMID 21098431 . ↑ Conesa, Ana; Pappas, Georgios J.; Furió-Tarí, Pedro; Balzano-Nogueira, Leandro; Martínez-Mira, Carlos; Tarazona, Sonia; Hernández-de-Diego, Rafael (2 липня 2018). PaintOmics 3: a web resource for the pathway analysis and visualization of multi-omics data . Nucleic Acids Research . 46 (W1): W503—W509. doi :10.1093/nar/gky466 . ISSN 0305-1048 . PMC 6030972 PMID 29800320 . ↑ Chari, Raj; Coe, Bradley P.; Wedseltoft, Craig; Benetti, Marie; Wilson, Ian M.; Vucic, Emily A.; MacAulay, Calum; Ng, Raymond T.; Lam, Wan L. (7 жовтня 2008). SIGMA2: A system for the integrative genomic multi-dimensional analysis of cancer genomes, epigenomes, and transcriptomes . BMC Bioinformatics . 9 (1): 422. doi :10.1186/1471-2105-9-422 . ISSN 1471-2105 . PMC 2571113 PMID 18840289 . {{cite journal }}: Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )↑ Choi, Hyungwon; Ewing, Rob; Choi, Kwok Pui; Fermin, Damian; Koh, Hiromi W. L. (23 липня 2018). iOmicsPASS: a novel method for integration of multi-omics data over biological networks and discovery of predictive subnetworks . bioRxiv : 374520. doi :10.1101/374520 . ↑ Kanai, Masahiro; Maeda, Yuichi; Okada, Yukinori (19 червня 2018). Grimon: graphical interface to visualize multi-omics networks . Bioinformatics . 34 (22): 3934—3936. doi :10.1093/bioinformatics/bty488 . ISSN 1367-4803 . PMC 6223372 PMID 29931190 . ↑ Su, Andrew I.; Loguercio, Salvatore; Carland, Tristan M.; Ducom, Jean-Christophe; Gioia, Louis; Meißner, Tobias; Fisch, Kathleen M. (1 червня 2015). Omics Pipe: a community-based framework for reproducible multi-omics data analysis . Bioinformatics . 31 (11): 1724—1728. doi :10.1093/bioinformatics/btv061 . ISSN 1367-4803 . PMC 4443682 PMID 25637560 . ↑ Montague, Elizabeth; Stanberry, Larissa; Higdon, Roger; Janko, Imre; Lee, Elaine; Anderson, Nathaniel; Choiniere, John; Stewart, Elizabeth; Yandl, Gregory (June 2014). MOPED 2.5—An Integrated Multi-Omics Resource: Multi-Omics Profiling Expression Database Now Includes Transcriptomics Data . OMICS: A Journal of Integrative Biology . 18 (6): 335—343. doi :10.1089/omi.2014.0061 . ISSN 1536-2310 . PMC 4048574 PMID 24910945 . ↑ Zhang, Bing; Wang, Jing; Straub, Peter; Vasaikar, Suhas V. (4 січня 2018). LinkedOmics: analyzing multi-omics data within and across 32 cancer types . Nucleic Acids Research . 46 (D1): D956—D963. doi :10.1093/nar/gkx1090 . ISSN 0305-1048 . PMC 5753188 PMID 29136207 . ↑ LinkedOmics :: Login . www.linkedomics.org . Процитовано 26 червня 2019 .↑ Kan, Zhengyan; Rejto, Paul A.; Roberts, Peter; Ding, Ying; AChing, Keith; Wang, Kai; Deng, Shibing; Schefzick, Sabine; Estrella, Heather (January 2016). OASIS: web-based platform for exploring cancer multi-omics data. Nature Methods . 13 (1): 9—10. doi :10.1038/nmeth.3692 . ISSN 1548-7105 . PMID 26716558 . ↑ Wu, Jiaqi; Hu, Shuofeng; Chen, Yaowen; Li, Zongcheng; Zhang, Jian; Yuan, Hanyu; Shi, Qiang; Shao, Ningsheng; Ying, Xiaomin (May 2017). BCIP: a gene-centered platform for identifying potential regulatory genes in breast cancer . Scientific Reports . 7 (1): 45235. Bibcode :2017NatSR...745235W . doi :10.1038/srep45235 . ISSN 2045-2322 . PMC 5361122 PMID 28327601 . ↑ Husi, Holger; Patel, Alisha; Fernandes, Marco (12 листопада 2018). C/VDdb: A multi-omics expression profiling database for a knowledge-driven approach in cardiovascular disease (CVD) . PLOS ONE . 13 (11): e0207371. Bibcode :2018PLoSO..1307371F . doi :10.1371/journal.pone.0207371 ISSN 1932-6203 . PMC 6231654 PMID 30419069 . ↑ Gupta, Amit Kumar; Kaur, Karambir; Rajput, Akanksha; Dhanda, Sandeep Kumar; Sehgal, Manika; Khan, Md. Shoaib; Monga, Isha; Dar, Showkat Ahmad; Singh, Sandeep (16 вересня 2016). ZikaVR: An Integrated Zika Virus Resource for Genomics, Proteomics, Phylogenetic and Therapeutic Analysis . Scientific Reports . 6 (1): 32713. Bibcode :2016NatSR...632713G . doi :10.1038/srep32713 . ISSN 2045-2322 . PMC 5025660 PMID 27633273 . ↑ Tagkopoulos, Ilias; Violeta Zorraquino; Rai, Navneet; Kim, Minseung (7 жовтня 2016). Multi-omics integration accurately predicts cellular state in unexplored conditions for Escherichia coli . Nature Communications . 7 : 13090. Bibcode :2016NatCo...713090K . doi :10.1038/ncomms13090 . ISSN 2041-1723 . PMC 5059772 PMID 27713404 . ↑ Li, Guojing; Lu, Zhongfu; Lin, Jiandong; Hu, Yaowen; Yunping Huang; Wang, Baogen; Wu, Xinyi; Wu, Xiaohua; Xu, Pei (26 лютого 2018). GourdBase: a genome-centered multi-omics database for the bottle gourd ( Lagenaria siceraria ), an economically important cucurbit crop . Scientific Reports . 8 (1): 3604. Bibcode :2018NatSR...8.3604W . doi :10.1038/s41598-018-22007-3 . ISSN 2045-2322 . PMC 5827520 PMID 29483591 . ↑ Liu, Haijun; Wang, Fan; Xiao, Yingjie; Tian, Zonglin; Wen, Weiwei; Zhang, Xuehai; Chen, Xi; Liu, Nannan; Li, Wenqiang (2016). MODEM: multi-omics data envelopment and mining in maize . Database . 2016 : baw117. doi :10.1093/database/baw117 . ISSN 1758-0463 . PMC 4976297 PMID 27504011 . ↑ Xu, Dong; Nguyen, Henry T.; Stacey, Gary; Gaudiello, Eric C.; Endacott, Ryan Z.; Zhang, Hongxin; Liu, Yang; Chen, Shiyuan; Fitzpatrick, Michael R. (1 січня 2014). Soybean knowledge base (SoyKB): a web resource for integration of soybean translational genomics and molecular breeding . Nucleic Acids Research . 42 (D1): D1245—D1252. doi :10.1093/nar/gkt905 . ISSN 0305-1048 . PMC 3965117 PMID 24136998 . ↑ Samaras, Patroklos; Schmidt, Tobias; Frejno, Martin; Gessulat, Siegfried; Reinecke, Maria; Jarzab, Anna; Zecha, Jana; Mergner, Julia; Giansanti, Piero (8 січня 2020). ProteomicsDB: a multi-omics and multi-organism resource for life science research . Nucleic Acids Research (англ.) . 48 (D1): D1153—D1163. doi :10.1093/nar/gkz974 . ISSN 0305-1048 . PMC 7145565 PMID 31665479 . ↑ Le, Hannah. Latch Registry: An integrated database for multi-omics . blog.latch.bio (англ.) . Процитовано 8 грудня 2023 . ↑ Mubeen, Sarah; Hoyt, Charles Tapley; Gemünd, André; Hofmann-Apitius, Martin; Fröhlich, Holger; Domingo-Fernández, Daniel (2019). The Impact of Pathway Database Choice on Statistical Enrichment Analysis and Predictive Modeling . Frontiers in Genetics . Т. 10. doi :10.3389/fgene.2019.01203 . ISSN 1664-8021 . PMC 6883970 PMID 31824580 . Процитовано 14 червня 2023 . {{cite news }}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання ) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання )