直鎖状ジユビキチンアルデヒド(緑)と共有結合的に連結されたUSP21(青)。ユビキチンのC末端がUSP21の活性部位を通って伸びている(右下)。 幅広いDUBの蛍光基質となるUb-AMC (英語版 ) 脱ユビキチン化酵素 (だつユビキチンかこうそ、英 : deubiquitinating enzyme 、略称: DUB )は、タンパク質 からユビキチン を切断除去するプロテアーゼ のグループである[ 1] [ 2] プロテアソーム とリソソーム を介したタンパク質分解 の調節や、タンパク質の細胞内局在の調整、タンパク質の活性化や不活性化、タンパク質間相互作用の調節のためにタンパク質に付加される[ 3] [ 4] [ 5] ペプチド結合 またはイソペプチド結合 を切断することで、ユビキチン化による効果を逆転させる。ヒトには約100種類のDUBの遺伝子が存在し、システインプロテアーゼ とメタロプロテアーゼ という2つの主要なクラスに分類される。システインプロテアーゼグループのDUBは、USP(ubiquitin-specific protease)、UCH(ubiquitin C-terminal hydrolase)、MJD(Machado-Josephin domain protease)、OTU(ovarian tumour protease)ファミリーなどからなる。メタロプロテアーゼグループのDUBは、JAMM(Jab1/Mov34/Mpr1 Pad1 N-terminal+)ドメインプロテアーゼのみが含まれる[ 2]
ヒトには95個のDUB遺伝子があると推定され、システインプロテアーゼとメタロプロテアーゼの2つの主要なクラスに分類される。58種類がUSP、4種類がUCH、5種類がMJD、14種類がOTU、そして14種類がJAMMドメインを含んでいる。そのうち90種類が発現しているが、11種類には活性がなく、残りの79種類が機能的な酵素であると考えられている[ 6]
システインプロテアーゼ型DUBには4つの主要なスーパーファミリーが存在する[ 7]
USPスーパーファミリー (USP1 (英語版 ) USP2 (英語版 ) USP3 , USP4 (英語版 ) USP5 (英語版 ) USP6 (英語版 ) USP7 , USP8 (英語版 ) USP9X (英語版 ) USP9Y (英語版 ) USP10 (英語版 ) USP11 (英語版 ) USP12 , USP13 (英語版 ) USP14 (英語版 ) USP15 (英語版 ) USP16 (英語版 ) USP17 , USP17L2 , USP17L3 , USP17L4 , USP17L5 , USP17L7 , USP17L8 , USP18 (英語版 ) USP19 (英語版 ) USP20 (英語版 ) USP21 , USP22 (英語版 ) USP23 , USP24 (英語版 ) USP25 (英語版 ) USP26 (英語版 ) USP27X (英語版 ) USP28 , USP29 , USP30 , USP31 , USP32 , USP33 (英語版 ) USP34 (英語版 ) USP35 , USP36 (英語版 ) USP37 (英語版 ) USP38 , USP39 (英語版 ) USP40 (英語版 ) USP41 , USP42 (英語版 ) USP43 , USP44 (英語版 ) USP45 , USP46 )
OTUスーパーファミリー (OTUB1 (英語版 ) OTUB2 (英語版 )
MJDスーパーファミリー (ATXN3 (英語版 ) ATXN3L )
UCHスーパーファミリー (BAP1 (英語版 ) UCHL1 , UCHL3 (英語版 ) UCHL5 (英語版 ) また、近年新たなファミリーが発見されている。
さらに、PPPDE(permuted papain fold peptidases of dsDNA viruses and eukaryotes)スーパーファミリーと呼ばれる、理解は進んでいないもののDUBと推定されるグループも存在し、実際にDUBであることが示されれば7番目のクラスとなる[ 10]
JAMMドメインスーパーファミリーのタンパク質は、亜鉛 を結合するメタロプロテアーゼである[ 7]
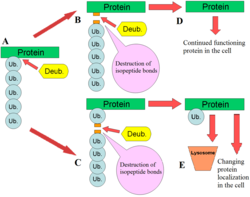
DUBの機能の模式図 DUBはユビキチン経路でいくつかの役割を果たす。DUBの最もよく特徴づけられている機能は、タンパク質からのモノユビキチンとポリユビキチン鎖の除去である。ユビキチン化は翻訳後修飾 (タンパク質の合成後に付加されたもの)であり、基質タンパク質のリジン 残基に1つのユビキチンまたはユビキチンの鎖が付加される。ユビキチンは、ユビキチン活性化酵素 (E1)、ユビキチン結合酵素 (E2)、ユビキチンリガーゼ (E3)からなるユビキチン化装置によってタンパク質に付加され、最終的にはリジン残基にイソペプチド結合で付加されたユビキチンとなる[ 11] [ 3] [ 4] [ 5] [ 2] SUMO やNEDD8 などのユビキチン様タンパク質 の除去にも関与しており、一部のDUBはこれらのタンパク質と基質タンパク質の間のイソペプチド結合を切断する[ 12]
DUBは、不活性型として発現するユビキチンをタンパク質分解によって活性化する。哺乳類ではユビキチンは、UBA52 (英語版 ) RPS27A (英語版 ) UBB (英語版 ) UBC (英語版 ) UBA52 、RPS27A 遺伝子はリボソーム タンパク質に融合した形でユビキチンを産生し、UBB 、UBC 遺伝子はポリユビキチン(C末端 とN末端 で連結されたユビキチンの鎖)を産生する[ 13] [ 14] [ 2]
DUBは、意図せずC末端テールが細胞内の求核性 低分子と結合したモノユビキチンタンパク質の切断も行う[ 2] アミド やユビキチン-チオエステル は標準的なE1-E2-E3カスケードによるユビキチン化反応でも形成される場合があり、グルタチオン やポリアミン はユビキチンとこれらの酵素の間のチオエステル結合を攻撃する可能性がある。ユビキチンC末端ヒドロラーゼ(UCH)は、こうした意図しない結合を広い特異性で加水分解するDUBの例である[ 12] [ 15]
またDUBは、遊離ポリユビキチン鎖を切断してモノユビキチンを産生する。こうしたポリユビキチン鎖は、基質タンパク質がない場合でもE1-E2-E3装置によって形成される場合がある。遊離ポリユビキチン鎖の他の発生源は、ユビキチン化基質の切断産物である。DUBがタンパク質に付加されたポリユビキチン鎖をその基部で切断した場合にはポリユビキチン鎖が遊離し、DUBによるモノユビキチンへの再生が必要となる[ 2]
USP7の触媒ドメイン。USPの触媒ドメインの構造は、fingers、palm、thumbという領域に分けられる。ユビキチンのC末端はthumbとpalmの間を通る。 DUBは多くの場合、触媒ドメインが1つまたは複数の付属ドメインに囲まれた構成をしており、それらの一部は標的の認識に寄与している。こうした付加的ドメインには、DUSP(domain present in ubiquitin-specific proteases)ドメイン、UBL(ubiquitin-like)ドメイン、MATH(meprin and TRAF homology)ドメイン、ZnF-UBP(zinc-finger ubiquitin-specific protease)ドメイン、ZnF-MYND(zinc-finger myeloid, nervy and DEAF1)ドメイン、UBA(ubiquitin-associated)ドメイン、CS(CHORD-SGT1)ドメイン、MIT(microtubule-interacting and trafficking)ドメイン、ロダネーゼ様ドメイン、TBC/RABGAPドメイン、B-boxドメインなどがある[ 6] [ 16]
DUBの触媒ドメインは、 USP、OTU、MJD、UCH、MPN+/JAMMなどのグループに分類される。最初の4つのグループはシステインプロテアーゼであり、MPN+/JAMMは亜鉛メタロプロテアーゼである。システインプロテアーゼ型のDUBはパパイン に類似しており、同様の作用機構を有する。ユビキチンと基質の間のアミド結合 の加水分解 の触媒には、触媒二残基または触媒三残基 のいずれかが利用される。システインプロテアーゼ型のDUBの触媒活性に寄与する活性部位の残基は、システイン 、ヒスチジン 、そしてアスパラギン酸 またはアスパラギン (触媒三残基のみ)である。ヒスチジンは、触媒三残基ではアスパラギン酸またはアスパラギンによって、触媒二残基では他の方法で極性化される。この極性化はシステインのpKa を低下させ、ユビキチンのC末端と基質のリジンの間のイソペプチド結合への求核攻撃を可能にする。メタロプロテアーゼ型DUBでは亜鉛イオンがヒスチジン、アスパラギン酸、セリン 残基に配位しており、水分子を活性化してイソペプチド結合への攻撃を可能にする[ 17] [ 18]
UBLドメインは、末端のグリシン 残基を欠いていることを除いて、ユビキチンと同様の構造を持つ。18種類のUSPがUBLドメインを持つと考えられており、USP以外でUBLを持つのはOTUB1とVCPIP1 (英語版 ) [ 16] [ 19] [ 20] [ 21] [ 22]
6種類のUSPには、約120残基からなるDUSPドメインが1つ、または複数個がタンデムに並んで存在している。DUSPドメインの機能は現在のところ不明であるが、タンパク質間相互作用、特にDUBの基質の認識に関与している可能性がある。この予測は、USP15のDUSPドメインには疎水的な溝が存在すること、そしてDUSP含有USPで観察されるタンパク質相互作用の一部はDUSPドメインがなくなることで生じなくなることに基づいている。DUSPドメインは3本のαヘリックス と3本のストランドからなる逆平行βシート から構成され、三脚のような形へフォールディングする。αヘリックスが脚の部分、βシートが座面に相当する。USPのDUSPドメイン内にはP G PI モチーフと呼ばれる保存されたアミノ酸配列が存在し、3ヘリックスバンドルに対してパッキングすることで高度に安定した構造となる[ 6] [ 23]
疾患におけるDUBの役割は完全には解明されていない。しかし、がん や神経疾患 などに関与する生理的過程での役割が知られていることから、疾患に関与していることが予測されている[ 24]
USP28は結腸がん や肺がん など、さまざまなタイプのがんで過剰発現している。さらに、USP28はc-Myc 、Notch 1 (英語版 ) c-jun 、ΔNp63 などの重要ながんタンパク質 を脱ユビキチン化して安定化する[ 25] [ 26] 扁平上皮癌 では、USP28はΔNp63-ファンコニ貧血経路を介してDNA修復 を調節し、化学療法 抵抗性を調節する[ 27] [ 28]
^ Wilkinson KD (December 1997). “Regulation of ubiquitin-dependent processes by deubiquitinating enzymes”. FASEB J. 11 (14): 1245–56. doi :10.1096/fasebj.11.14.9409543 . PMID 9409543 . ^ a b c d e f “Regulation and cellular roles of ubiquitin-specific deubiquitinating enzymes” . Annu. Rev. Biochem. 78 : 363–97. (2009). doi :10.1146/annurev.biochem.78.082307.091526 . PMC 2734102 . PMID 19489724 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2734102/ .
^ a b “The ubiquitin-proteasome proteolytic pathway: destruction for the sake of construction”. Physiol. Rev. 82 (2): 373–428. (April 2002). doi :10.1152/physrev.00027.2001 . PMID 11917093 .
^ a b “Proteasome-independent functions of ubiquitin in endocytosis and signaling”. Science 315 (5809): 201–5. (January 2007). Bibcode : 2007Sci...315..201M . doi :10.1126/science.1127085 . PMID 17218518 .
^ a b “Non-traditional functions of ubiquitin and ubiquitin-binding proteins”. J. Biol. Chem. 278 (38): 35857–60. (September 2003). doi :10.1074/jbc.R300018200 . PMID 12860974 .
^ a b c “A genomic and functional inventory of deubiquitinating enzymes”. Cell 123 (5): 773–86. (December 2005). doi :10.1016/j.cell.2005.11.007 . hdl :1874/20959 PMID 16325574 .
^ a b “Mechanism and function of deubiquitinating enzymes”. Biochim. Biophys. Acta 1695 (1–3): 189–207. (November 2004). doi :10.1016/j.bbamcr.2004.10.003 . PMID 15571815 .
^ Abdul Rehman, Syed Arif; Kristariyanto, Yosua Adi; Choi, Soo-Youn; Nkosi, Pedro Junior; Weidlich, Simone; Labib, Karim; Hofmann, Kay; Kulathu, Yogesh (2016-07-07). “MINDY-1 Is a Member of an Evolutionarily Conserved and Structurally Distinct New Family of Deubiquitinating Enzymes” . Molecular Cell 63 (1): 146–155. doi :10.1016/j.molcel.2016.05.009 . ISSN 1097-2765 . PMC 4942677 . PMID 27292798 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4942677/ . ^ Kwasna, Dominika; Abdul Rehman, Syed Arif; Natarajan, Jayaprakash; Matthews, Stephen; Madden, Ross; De Cesare, Virginia; Weidlich, Simone; Virdee, Satpal et al. (2018-04-05). “Discovery and Characterization of ZUFSP/ZUP1, a Distinct Deubiquitinase Class Important for Genome Stability” (英語). Molecular Cell 70 (1): 150–164.e6. doi :10.1016/j.molcel.2018.02.023 . ISSN 1097-2765 . PMC 5896202 . PMID 29576527 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5896202/ . ^ “Novel predicted peptidases with a potential role in the ubiquitin signaling pathway”. Cell Cycle 3 (11): 1440–50. (November 2004). doi :10.4161/cc.3.11.1206 . PMID 15483401 . ^ “Modification of proteins by ubiquitin and ubiquitin-like proteins”. Annu. Rev. Cell Dev. Biol. 22 : 159–80. (2006). doi :10.1146/annurev.cellbio.22.010605.093503 . PMID 16753028 . ^ a b Wing SS (May 2003). “Deubiquitinating enzymes--the importance of driving in reverse along the ubiquitin-proteasome pathway”. Int. J. Biochem. Cell Biol. 35 (5): 590–605. doi :10.1016/s1357-2725(02)00392-8 . PMID 12672452 .
^ “Regulatory mechanisms involved in the control of ubiquitin homeostasis”. J Biochem 147 (6): 793–8. (2010). doi :10.1093/jb/mvq044 . PMID 20418328 . ^ “The yeast ubiquitin genes: a family of natural gene fusions” . EMBO J. 6 (5): 1429–39. (May 1987). doi :10.1002/j.1460-2075.1987.tb02384.x . PMC 553949 . PMID 3038523 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC553949/ . ^ “Ubiquitin carboxyl-terminal hydrolase acts on ubiquitin carboxyl-terminal amides”. J. Biol. Chem. 260 (13): 7903–10. (July 1985). PMID 2989266 . ^ a b “Breaking the chains: structure and function of the deubiquitinases”. Nat. Rev. Mol. Cell Biol. 10 (8): 550–63. (August 2009). doi :10.1038/nrm2731 . PMID 19626045 .
^ Komander D (2010). “Mechanism, specificity and structure of the deubiquitinases”. Conjugation and Deconjugation of Ubiquitin Family Modifiers . Subcellular Biochemistry. 54 . 69–87. doi :10.1007/978-1-4419-6676-6_6 . ISBN 978-1-4419-6675-9 . PMID 21222274 ^ “Emerging roles for cysteine proteases in human biology”. Annu. Rev. Physiol. 59 : 63–88. (1997). doi :10.1146/annurev.physiol.59.1.63 . PMID 9074757 . ^ “The role of UBL domains in ubiquitin-specific proteases”. Biochem. Soc. Trans. 40 (3): 539–45. (June 2012). doi :10.1042/BST20120004 . PMID 22616864 . ^ “Dissection of USP catalytic domains reveals five common insertion points”. Mol Biosyst 5 (12): 1797–808. (December 2009). doi :10.1039/b907669g . PMID 19734957 . ^ “Structural variability of the ubiquitin specific protease DUSP-UBL double domains”. FEBS Lett. 585 (21): 3385–90. (November 2011). doi :10.1016/j.febslet.2011.09.040 . PMID 22001210 . ^ “Structure of the USP15 N-terminal domains: a β-hairpin mediates close association between the DUSP and UBL domains”. Biochemistry 50 (37): 7995–8004. (September 2011). doi :10.1021/bi200726e . PMID 21848306 . ^ “Solution structure of the human ubiquitin-specific protease 15 DUSP domain”. J. Biol. Chem. 281 (8): 5026–31. (February 2006). doi :10.1074/jbc.M510993200 . PMID 16298993 . ^ “Deubiquitylating enzymes and disease” . BMC Biochem. 9 (Suppl 1): S3. (2008). doi :10.1186/1471-2091-9-S1-S3 . PMC 2582804 . PMID 19007433 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2582804/ . ^ E. Diefenbacher, Markus; Popov, Nikita; al., Et (June 2014). “The deubiquitinase USP28 controls intestinal homeostasis and promotes colorectal cancer” . J. Clin. Invest. 124 (8): 3407–18. doi :10.1172/JCI73733 . PMC 4109555 . PMID 24960159 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4109555/ . ^ Prieto-Garcia, Cristian; Hartmann, Oliver; Reissland, Michaela; Braun, Fabian; Fischer, Thomas; Walz, Susanne; Schülein-Völk, Christina; Eilers, Ursula et al. (2020-04-07). “Maintaining protein stability of ∆Np63 via USP28 is required by squamous cancer cells” . EMBO molecular medicine 12 (4): e11101. doi :10.15252/emmm.201911101 . ISSN 1757-4684 . PMC 7136964 . PMID 32128997 . https://pubmed.ncbi.nlm.nih.gov/32128997 . ^ Prieto-Garcia, C.; Hartmann, O; Diefenbacher, M.; et, al. (September 2020). “Inhibition of USP28 overcomes Cisplatin-Resistance of Squamous Tumors by Suppression of the Fanconi Anemia Pathway”. bioRxiv 10.1101/2020.09.10.291278 ^ “The potential role of ubiquitin c-terminal hydrolases in oncogenesis”. Biochim. Biophys. Acta 1806 (1): 1–6. (August 2010). doi :10.1016/j.bbcan.2010.03.001 . PMID 20302916 .