|
ウイルス定量ウイルスの定量は、ウイルス濃度を測定するために特定の容量内のウイルスの数を数えること。産業・学術などでの研究開発(R&D)のみならず、製造でもさまざまな工程においてウイルスの量が重要になることから必要になる。たとえば、ウイルスワクチンや、ウイルスベクターを使用した組換えタンパク質、ウイルス抗原の生産では、生産収量を最適化し、常に変化する需要や用途に対応するため、プロセスを継続的に適応・監視する目的でウイルス定量を必要とする。既知のウイルスを定量化する必要がある例として、クローンスクリーニング、感染多重度(MOI)の最適化、細胞培養法の改良などがある。この項では、液体サンプル中のウイルスを定量化するために現在使用されているさまざまな手法について説明する。手法は従来の手法と最新の手法の2つのカテゴリに分けられる。従来の手法は、何十年にもわたって使用されてきた業界標準の手法であるが、一般的に時間がかかり、労力を必要とする。最新の手法は、定量化時間を大幅に短縮する比較的新しい市販の製品やキットである。ただし、すべての手法の網羅的なレビューではなく、従来の手法と新しい市販の手法の代表例を示す。ウイルスの定量化には他にも公開された方法が存在する可能性があるが、ここでは非商用の手法については説明しない。 従来法プラークアッセイ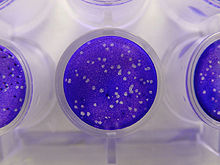 プラークベースのアッセイは、感染量からウイルス濃度を測定する標準的な方法である。プラークアッセイは、ウイルス量の1つの尺度であるウイルスサンプル中のプラーク形成単位(pfu)の数を測定する。この測定法は、ペトリ皿またはマルチウェルプレートで実施される微生物学的手法であり、具体的には、コンフルエントな単層の宿主細胞に、さまざまな希釈率でウイルスを感染させ、寒天やカルボキシメチルセルロースなどの半固体培地で覆って、ウイルス感染が際限なく広がるのを防ぐ。ウイルスプラークは、ウイルスが固定細胞単層内の細胞に感染すると形成される[1]。ウイルスに感染した細胞は溶解し、隣接する細胞に感染を広げ、感染から溶解までのサイクルが繰り返される。感染した細胞領域は、光学顕微鏡または視覚的に見ることができるプラーク(感染していない細胞に囲まれた感染領域)を形成する。操作の具体例としては、重層した培地を洗い流し、細胞質が着色するまでクリスタルバイオレット溶液を添加し15分間静置する。余分な染色液を水で静かに取り除くと、死んだ細胞の位置が無着色で示される[2]。プラークの形成には、分析するウイルスによって異なるが、3〜14日かかる場合がある。プラークは通常手動でカウントされ、その結果は、プレートの調製に使用された希釈倍率を乗じてサンプル単位体積あたりのプラーク形成単位の数(pfu/mL)を計算する。 pfu/mLの結果は、サンプル内の感染性粒子の数を表し、形成された各プラークが1つの感染性ウイルス粒子を表すという仮定に基づいた数である[3][4]。 フォーカス形成アッセイ(FFA)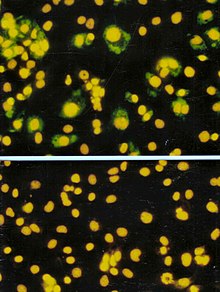 フォーカス形成アッセイ(FFA)はプラークアッセイの一部であるが、FFAはウイルス抗原に特異的な蛍光標識抗体を使用して、感染した宿主細胞とプラークが形成される前の感染ウイルス粒子を検出する免疫染色技術を用いる。 FFAは、プラーク形成と異なり細胞溶解を必要としないことから、細胞膜を溶解しないプラークアッセイに適さないウイルス種の定量に特に用いられる。プラークアッセイと同様に、単層の宿主細胞に段階希釈したウイルスサンプルを感染させ、感染性ウイルスの拡散を制限する半固体重層培地の下で比較的短い培養時間(24〜72時間など)インキュベートし、局所的な感染細胞のクラスター(フォーカス、病巣)を形成する。続いてプレートをウイルス抗原に対する蛍光標識抗体で染色し、蛍光顕微鏡を使用してフォーカスの数を数え、定量する。 FFA法では、通常、プラークや50%組織培養感染量(TCID50)アッセイよりも短時間で結果が得られるが、必要な試薬や機器の点で高価になることがある。測定時間は、測定者が数える領域のサイズにも依存する。より大きな領域はより多くの時間を必要とするが、より正確な値となる。 FFAの結果は、ミリリットルあたりのフォーカス形成単位(FFU/mL)として表される。 限界希釈法50%組織培養感染量(50% Tissue Culture Infective Dose: TCID50)は、感染性のウイルス力価の尺度である。TCID50は接種された組織培養細胞の50%を殺す、あるいは細胞変性効果を生むのに必要なウイルスの量を定量する。TCID50は臨床研究などにおいてウイルスの致死量を決定する必要がある場合や、ウイルスがプラークを形成しない場合によく用いられる。組織培養での操作例として、宿主細胞を播種し、段階希釈したウイルスを添加、培養後、死滅した細胞(感染細胞)の割合を手動で観察する。各ウイルス希釈での割合を記録し、結果から数学的にTCID50を計算する[5]。測定方法や測定原理の違いから、TCID50やPFU/ml、他の感染性アッセイの結果は等価ではない。この手法は、細胞の感染が必要であり、最大1週間かかる場合がある[6]。 TCID50計算には、一般的に以下の2つの計算方法が使用される(EC50、IC50、LD50などの他のタイプの50%エンドポイントの計算にも使用される) 数学的には、TCID50で陰性だったウェルは0個のプラーク形成単位を表し、陽性だったウェルはそれぞれ1個以上のプラーク形成単位を表すので、予想されるPFUはTCID50の2分の1よりも若干大きくなる。ポアソン分布に基づくと、より正確な推定値が得られ、約0.69 PFU = 1 TCID50である[8]。ポアソン分布は、一定の空間において、既知の平均発生確率で発生するランダムなイベントの発生回数を表す確率分布である。観測された陰性のウェルは感染イベントが0回発生したとみなされることから、P(0)は陰性のウェルの比率であり、mは体積あたりの感染単位の平均数(PFU/ml)であることから、P(0) = e-mである。TCID50として表される力価の場合、P(0) = 0.5であるから、e-m = 0.5、すなわちm = -ln 0.5であり、これは約0.69と計算される。ただし、実際には、この関係は同じウイルスと細胞の組み合わせでも当てはまらないことがある。これは、2種類の測定法の測定条件が異なることや、ウイルスの感染力は細胞年齢、重層培地等に非常に影響を受けやすいことによる。しかし、次の文献では、この関係を異なる形で定義している。同じ細胞系を使用し、ウイルスがそれらの細胞上でプラークを形成し、プラーク形成を阻害するような処置を加えないと仮定すると、1 mlのウイルスストックはTCID50の約半分の数のプラーク形成単位(PFU)を持つことが予想される。これは推定に過ぎないが、添加した細胞層の50%に感染する限界希釈液では、感染した細胞層に最初に1つのプラークができると予想されることが多いという根拠に基づいている。場合によっては、偶然にも2つ以上のプラークが形成されることもあるので、実際のPFU数は実験的に決定されるべきである。(ATCC - Converting TCID50 to plaque forming units PFU-124を参照) タンパク質定量タンパク質ベースのウイルス定量法にはいくつかの種類がある。一般に、タンパク質定量では、感染した細胞やウイルス粒子の数ではなく、サンプル中の総タンパク質量か、特定のウイルスタンパク質の量のいずれかを定量する。一般的には蛍光検出によって定量することが最も多い。サンプル中のタンパク質を直接定量する方法もあるが、タンパク質定量の前に宿主細胞に感染させ、培養してウイルスの増殖する場合もある。サンプル中のタンパク質(すなわちウイルス)の量と測定法自体の感度に応じて使用する測定法が選ばれる。ウイルスの増幅が必要な場合、分析前に細胞やウイルスの溶解・分解が行われることが多い。ほとんどのタンパク質ベースの測定法は比較的迅速で感度が高いが、正確な定量には標準品を必要とし、実際のウイルス粒子濃度ではなくタンパク質を定量している。以下は、広く使用されているタンパク質ベースの定量法の具体例である。 血球凝集アッセイ→詳細は「赤血球凝集アッセイ」を参照
血球凝集アッセイ(HA)は、インフルエンザウイルスの定量に代表される一般的な非蛍光タンパク質定量法である。インフルエンザウイルスの表面タンパク質である血球凝集素が赤血球を凝集させる反応により定量している。この定量法では、インフルエンザサンプルの希釈液を1%赤血球溶液と1時間培養し、凝集が最初に発生するウイルス希釈液を目視で確認する。この定量法の結果は赤血球凝集単位(hemagglutination units: HAU)の形で得られ、通常、PFUに対する比率は106の範囲である [9] [10]。この定量法は測定に約1〜2時間かかり、測定結果は操作者の技術や経験によって大きく異なることがある。 血球凝集阻害アッセイ(HI)は、血清中のインフルエンザ特異的抗体濃度の測定によく用いられるHAアッセイのうちの一手法である。血清中にインフルエンザウイルスに対する抗体が十分な濃度で存在する場合、ウイルスの赤血球への付着が妨害され、血球凝集反応が阻害されることを利用した測定法である[11]。 ビシンコニン酸アッセイ→詳細は「ビシンコニン酸アッセイ」を参照
ビシンコニン酸アッセイ(BCA)は、単純な比色測定による最も一般的なタンパク質定量分析法である。 BCAはLowry法やBradford法に類似しており、現在Thermo Fisher Scientific社となっているPierce社によって最初に市販化された。 BCAアッセイでは、タンパク質のペプチド結合がCu2+をCu1+に定量的に還元し、薄い青色を呈する。BCAはCu1+を2:1の比率でキレートし、562 nmに吸収を持つより濃い色の錯体を形成する。分光光度計またはプレートリーダーで562 nmでの試料の吸光度を測定し、既知の標準曲線と比較することで、試料中の総タンパク質濃度を測定することができる[12]。分析の所要時間は30分〜1時間ほどである。この分析法は汎用的で高速だが、すべてのタンパク質を定量するため特異性に欠け、定量するウイルス試料中の宿主細胞由来タンパク質の混入が非常に低濃度である必要がある。 一元放射状免疫拡散法一元放射状免疫拡散法(single radial immunodiffusion method;SRID法)は、マンチーニ法とも呼ばれ、半固形培地(寒天など)上での拡散によって特定のウイルス抗原の量を検出するタンパク質定量分析である。目的の抗原に特異的な抗血清を含む培地の上に、抗原を含む円筒状のディスクを中央に置く。抗原は培地に放射状に拡散し、平衡に達するまで成長する免疫沈降のリングが形成される。測定時間は、抗原と抗体の平衡時間に応じて、10時間から数日の範囲である。リングの直径は、タンパク質濃度の対数と直線関係にあり、定量には既知のタンパク質標準の直径と比較する[13]。いくつかのキットと血清がこの分析用に市販されている(例:バインディングサイト株式会社)。 透過型電子顕微鏡透過型電子顕微鏡(Transmission Electron Microscope; TEM)は、磁場で集束した電子線をサンプルにあて画像化する電子顕微鏡の一種であり、光学顕微鏡の1000倍(0.2 nm)の空間分解能を持つ[14]。極薄くしたネガティブ染色のサンプルが必要であり、サンプルの準備として、コーティングしたTEMグリッドと呼ばれる金属板の上への標本を載せ、電子不透過性の液体によりネガティブ染色を行う[15]。 組織に埋没しているサンプルは、薄く切片化すれば検査可能である。サンプルの準備はプロトコルと実験者によって異なるが、通常、数時間かかる。 TEM画像は個々のウイルス粒子を観察でき、定量的画像分析によりウイルス濃度の測定も可能である。TEMによる高解像度画像は、他の方法では難しい粒子の形態情報を得ることができる。感染力の有無に関係なく、すべての粒子の数を1 mLあたりのウイルス様粒子数(vlp/mL)として定量するため、定量的TEMの結果は他の分析結果よりも大きくなることが多い。定量的TEMは一般に106粒子/mLを超えるウイルス濃度に適用できる。機器のコストが高く、必要なスペースとサポート施設が必要なため、TEMが利用できる施設は限られている。 最新の分析法調整可能抵抗パルスセンシング(TRPS)→詳細は「調整可能抵抗パルスセンシング」を参照
調整可能抵抗性パルスセンシング(Tunable resistive pulse sensing; TRPS)は、個々のウイルス粒子がサイズ調整可能なナノポアを通過するところを1つずつ測定することにより、高スループットの単一粒子測定が可能な測定技術である[16]。溶液中のウイルス粒子のサイズと濃度を高解像度で同時に測定できるという利点があり、サンプルの安定性や凝集体評価、総ウイルス粒子濃度(vp/mL)測定に使用できる[17]。 TRPSの測定はイオン性緩衝液中で行われ、分析前にサンプルを前染色する必要がないため、この手法は蛍光色素による前処理を必要とする手法よりも迅速であり、総調製時間と測定時間はサンプルあたり10分以下である。TRPSによるウイルス分析はqViro-Xシステムとして市販されており、測定後にオートクレーブ処理による失活が可能である。 フローサイトメトリーほとんどのフローサイトメーターはウイルス定量には十分な感度を持たないが、ウイルス定量可能なフローサイトメーターはいくつか市販されている。ウイルスカウンターは、共に局在するタンパク質と核酸を蛍光により検出することで、サンプル中の完全ウイルス粒子数を定量する。サンプルは、タンパク質に特異的な色素と核酸に特異的な色素の2つの色素で染色され、流路中を流れる粒子をレーザーで分析する。ウイルス粒子の濃度(vp/mL)は、2つの異なる蛍光チャネルで同時に計測された粒子数と、測定されたサンプルの流量から測定される[18]。結果は一般的にTEMと近い結果が得られる。この分析法では105–109 VP/mLでの範囲で直線性があり、分析時間は約10分でサンプル調製時間も短い。 定量ポリメラーゼ連鎖反応(qPCR)→詳細は「定量PCR」を参照
定量PCR(Quantitative polymerase chain reaction; qPCR)は、ポリメラーゼ連鎖反応を利用してウイルスDNAまたはRNAを増幅し、蛍光により検出と定量を行う分析法。一般に、qPCRでは、未知のサンプルと既知の濃度の標準の段階希釈を検量線として並行して比較分析することで定量する。定量的検出は、配列特異的プローブやSYBR Greenなどの非特異的蛍光色素など、さまざまな蛍光検出法により行う[19] 。TaqMan(Applied Biosystemsが開発)、Molecular Beacons、またはScorpionなどの配列特異的プローブは、反応中に生成された適切な配列のDNAにのみ結合し蛍光を発する。 SYBR Green色素は、反応中に生成されたすべての二本鎖DNA[20]に結合し蛍光を発する。SYBR Greenは利用しやすいが、特異性がなく感度が低いため、配列特異的プローブのqPCR検出法が使用されることが多い。内部標準を含む複数のサンプルからのCt値(増幅産物が統計的に有意な増加を示したPCRサイクル数)の比較を通じて相対定量を行う比較しきい値法などqPCRには多くのバリエーションがある。PCRは、完全な感染性ウイルス粒子(ビリオン)、壊れたウイルス粒子、溶液中の遊離核酸を含む、すべての標的核酸を増幅する。このため、qPCRの結果(ゲノムコピー/mLで表される)は、TEMの結果よりも通常大きな値になる。ウイルスの定量化では、核酸のコピー数に対するビリオンの比率が1対1になることは稀である。これは、ウイルス複製中に、核酸とウイルスタンパク質が常に1:1の比率で生成されるとは限らず、ウイルスの組み立てプロセスにより、完全なビリオン、空のキャプシドや、過剰な遊離ウイルスゲノムが生じることによる。口蹄疫ウイルスの例では、活発に複製している宿主細胞内の全ビリオンとRNAコピーの比率は約1:1000である[21]。qPCRベースのウイルス定量用の製品は、多くの企業から市販されている(例:Invitrogen、Roche、Qiagenなど)。 qPCRによる定量の利点には、迅速な分析時間(1〜4時間)と感度(他の方法よりもはるかに低い濃度のウイルスを検出できる)などがある。 酵素免疫測定法(ELISA) ELISAは、酵素に結合した特定の抗体を利用して、サンプル中の未知の量の抗原(すなわちウイルス)の存在を検出するタンパク質検出法のより現代的な手法である。サンプル中の抗原の濃度に応じて、酵素が試薬を検出可能な信号に変換することで、抗原抗体結合反応を検出・定量化する[22]。西洋ワサビペルオキシダーゼ(HRP)は、シグナルを増幅し、分析法の感度を高めることができることから、ELISAでよく利用される酵素である。 ELISA法には多くの手法があるが、一般に直接法、間接法、競合法、サンドイッチ、逆ELISA法のいずれかに分類できる[23]。ELISAキットは多くの企業(例:Invitrogen、Santa Cruz Biotechnology Inc.)から市販されており、定量化は一般的に発色レポーターや蛍光による。この手法は、従来の方法よりもはるかに労力がかからず、抗体のインキュベーション時間に応じ4〜24時間ほどかかる。 参考文献
|

