|
Influenzavirus A
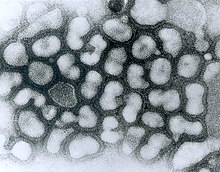
L'Influenzavirus A o virus dell'influenza A è una specie di virus appartenente alla famiglia degli Orthomyxoviridae, in grado di infettare alcune specie di uccelli e mammiferi. È l'unica specie appartenente al genere Alphainfluenzavirus.[1] I sottotipi appartenenti all'influenzavirus A sono stati identificati in molti tipi di uccelli selvatici, benché lo sviluppo dell'influenza sia molto raro in questi animali. L'influenzavirus A è in grado di sostenere gravi entità sindromiche nel pollame e, molto raramente, negli esseri umani[2] Occasionalmente, il virus può trasmettersi da uccelli d'acqua al pollame domestico, con rischio di infezione e malattia per l'essere umano. In tali condizioni, è possibile lo sviluppo di epidemie e pandemie.[3][4] Struttura del virusIl virus dell'influenza A è un virus a RNA segmentato, a singolo filamento e a polarità negativa (per essere tradotto in proteina è necessaria una RNA polimerasi RNA dipendente che crei il filamento a polarità positiva). Esistono diversi sottotipi, in accordo con le possibili combinazioni tra le diverse forme di emoagglutinina (H) e di neuraminidasi (N). Esistono 18 differenti tipi di emoagglutinina (da H1 a H18) e 11 differenti tipi di neuroaminidasi (da N1 a N11)[5]. Il tipo H16 è il più nuovo ed è stato isolato nel gabbiano dalla testa nera in Svezia e Paesi Bassi nel 1999 e riportato nella letteratura nel 2005.[6] I diversi sottotipi di virus hanno un diverso profilo patogenetico e clinico. Inoltre i diversi sottotipi virali possiedono capacità diverse di contagiare specie animali differenti. La struttura di tutti gli 'Influenzavirus A è simile, con caratteristica forma sferica o filamentosa.[7] Il genomaIl genoma è composto da 13.588 basi[8] ed è diviso in 8 segmenti che codificano per 11 diverse proteine (HA, NA, NP, M1, M2, NS1, NEP, PA, PB1, PB1-F2, PB2). La natura segmentata del materiale genomico consente lo scambio dei frammenti tra i diversi sottotipi virali durante la co-infezione di uno stesso organismo. Gli otto frammenti di RNA sono:
I segmenti genomici hanno sequenze terminali comuni e i vari filamenti di RNA sono parzialmente legati attraverso legami ad idrogeno. In seguito alla trascrizione da RNA a polarità negativa a RNA a polarità positiva viene aggiunto il cap in posizione 5' grazie all'opera della proteina NS1 che utilizza molecole di preRNA di origine cellulare. Il clivaggio del cap è operato da PB2. I frammenti positivi così creati sono utilizzati per la sintesi proteica virale e come stampo per l'RNA a polarità negativa. La sintesi di RNA e l'assemblaggio delle nucleo proteine avviene nel nucleo, mentre la sintesi proteica avviene nel citoplasma. Il core virale lascia il nucleo per dirigersi verso la membrana cellulare. La liberazione delle particelle virali avviene in seguito al rivestimento del core nucleare con la membrana cellulare contenente le proteine HA e NA. Sottotipi noti
Patologie nell'essere umanoI sintomi sono:
L'infiammazione delle mucose aeree a causa dell'influenza (soprattutto negli anziani, nei bambini, negli immunodepressi e nelle persone che hanno patologie) rende suscettibili a infezioni opportunistiche batteriche: polmonite (sia bilaterale che lobale), broncopolmonite e raramente alla sindrome di Reye. Tuttavia alcuni ceppi di virus influenzali danno una polmonite estremamente grave in soggetti peraltro sani. Generalmente l'influenza ha una incubazione di 2-3 giorni e risolve poi spontaneamente anche se alcuni sintomi come la tosse possono protrarsi anche alcune settimane. La percentuale delle vittime correlate al virus H1N1 sarebbe dello 0,0038% dei malati[10], contro lo 0,2% dei decessi causati dalla normale influenza stagionale.[11][12] Note
Voci correlateAltri progetti
|
||||||||||||||||||||||||||