|
Estrutura comunitáriaEm ciência das redes, uma rede tem estrutura comunitária se os nós dessa rede podem ser divididos em conjuntos onde os nós são densamente conectados internamente. Esses conjuntos podem ter sobreposição ou serem disjuntos. No caso disjunto, é assumido que os nós são esparsamente conectados com os nós de outros conjuntos. De forma geral, é mais provável um nó em uma rede com estrutura comunitária tenha uma aresta com outro nó de sua(s) comunidade(s) do que outros nós na rede, em contraste com redes aleatórias onde todo par de nós tem a mesma chance de ter uma ligação. Definição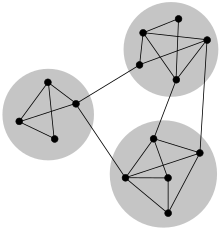 Comunidades disjuntasNessa seção, vamos considerar apenas estruturas de comunidade disjuntas. Os primeiros trabalhos sobre estrutura comunitária disjunta assumiam que as comunidades eram cliques[1]. De fato, cliques são subgrafos densos na rede, isso constitui uma comunidade. Por outro lado, definir comunidades somente como cliques é muito restritivo porque geralmente em redes complexas é raro cliques de número de nós superiores a três e podemos deixar de classificar comunidades legítimas[2]. Por exemplo, na Fig. 1 à direita, nenhuma das comunidades destacadas são cliques. Para relaxar a definição de comunidades através de cliques, foram propostas duas definições: comunidades fortes e comunidades fracas[3]. Dada uma rede representada por um grafo , é denotado como o grau do vértice . Seja um subgrafo tal que , vamos chamar de o número de arestas conectado o nó com os nós em . E similarmente, o número de arestas conectando o nó com os nós não pertencentes a . Comunidades fortesO subgrafo é uma comunidade forte se Em outras palavras, todo nó pertencente a uma comunidade forte tem mais ligações internas em relação a comunidade do que externas. Comunidades fracasO subgrafo é uma comunidade fraca se Nas comunidades fracas, a soma de todos os graus internos de é maior que a soma dos graus externos. Note que todo clique é uma comunidade forte e toda comunidade forte também é uma comunidade fraca. Por outro lado, comunidades fracas nem sempre são comunidades fortes. É possível observar que há diversas definições para comunidades, algumas mais restritivas como definir comunidades como cliques e outras menos, como comunidades fortes e fracas. Adicionalmente, há diversas outras formas de definir comunidades que podem ser mais apropriadas dependendo do contexto[4]. ModularidadeEm redes aleatórias, não é possível observar nenhum tipo de estrutura comunitária, pois é esperado que as chances de ligações ocorram de forma uniforme por todos os nós. Através dessa observação, é possível criar uma métrica de modularidade calculando a diferença do padrão de ligações com de uma rede aleatória. Dessa forma, é possível medir quantitativamente se uma partição tem estrutura de comunitária. Vamos supor uma rede representada pelo grafo com a matriz de adjacência e uma partição da rede em comunidades . Note que é um subgrafo, vamos denotar como o número de links internos da comunidade e a soma do graus dos nós em em relação a . Para cada comunidade , podemos definir a diferença entre suas ligações em e o número esperado de ligações entre dois nós e caso a rede fosse aleatória: Em uma rede aleatória, a probabilidade de dois nós e estarem conectados é então, a modularidade da rede toda é onde é caso os nós e pertencem a mesma comunidade e caso o contrário. Dessa forma, apenas os nós que estão na mesma comunidade serão considerados, então podemos reescrever a modularidade como Quanto maior for a modularidade, melhor representa uma partição de em comunidades. Também, é sempre menor que 1[5][6]. Algoritmos para encontrar comunidadesEncontrar comunidades em uma rede arbitrária pode ser difícil computacionalmente. Normalmente, o número de comunidades é desconhecido a priori e as comunidades tem diferentes tamanhos e densidade de ligações. Apesar dessas dificuldades, algoritmos foram desenvolvidos com essa finalidade[7]. Método por corte mínimoO método por corte mínimo, a rede é dividida em um número pré-determinado de comunidades, onde essas comunidades normalmente tem tamanhos iguais. Essas comunidades são escolhidas de tal forma que o número de arestas entre os grupos é minimizado. Esse método funciona bem para várias aplicações mas não tem resultados satisfatórios para redes no geral porque o método encontrará comunidades sem levar em consideração a estrutura implícita, pois o número de comunidades é sempre fixo[8]. Hierarchical clusteringOutro método para encontrar estruturas comunitárias é agrupamento hierárquico. Nesse método, é necessário uma medida de similidade quantificando algum tipo de similidade (normalmente topológica) entre par de nós. Algumas medidas utilizadas são similaridade por cosseno, índice de Jaccard e Distância de Hamming entre as linhas da matriz de adjacência. Então, são formadas as comunidades com nós similares levando em consideração essa métrica. Há diversos métodos para realizar o agrupamento desses nós, sendo um deles o clustering de ligação única, no qual dois grupos são considerados separados em comunidades se e somente se todos os pares de nós em grupos diferentes tem a similaridade maior que um limiar. Outra abordagem que obteve resultados melhores é o uso de várias métricas de similaridades e dissimilaridades, combinadas através de uma combinação convexa[9]. Algoritmo de Girvan–NewmanOutra abordagem para detecção de comunidades é o algoritmo de Girvan-Newman [10]. Esse algoritmo identifica as arestas em uma rede que estão na fronteira de comunidades e as remove, deixando somente as comunidades isoladas. A identificação é feita utilizando intermediação, que é um número associado a cada aresta onde esse valor é maior se a aresta está "entre" muitos pares de nós. O algoritmo de Girvan-Newman retorna resultados razoáveis em relação a qualidade mas sua popularidade advém do fato que esse método é implementado em diversos pacotes de software. Em contrapartida, sua execução é lenta levando tempo proporcional a em uma rede de vértices e arestas, por essa razão é impraticável para redes de alguns milhares de nós[10]. Maximização da modularidadeApesar de suas desvantagens, um dos métodos mais utilizados para detecção de comunidades é a maximização da modularidade. A maximização da modularidade detecta comunidades realizando uma busca entre candidatos de partições e seleciona a partição (ou partições) com maior modularidade. Devido ao fato que uma busca por todas as possíveis partições é algo intratável, algoritmos baseados em aproximação são utilizados como algoritmos gulosos, simulated annealing, otimização espectral. Essas propostas oferecem diferentes resultados referente a velocidade e acurácia[5][11]. Um método popular de maximização da modularidade é o método de Louvain, que iterativamente realiza a otimização de comunidades locais até que a modularidade global não seja mais melhorada através de perturbações na partição de comunidades[12][13]. Atualmente, o melhor algoritmo para a otimização de modularidade utiliza o método RenELL que é um exemplo de aprendizado ensemble extremo[14] [15]. A modularidade é questionada como métrica para otimização porque já foi mostrado que sua maximização falha em detectar clusters menores que certa escala que depende do tamanho da rede[6]. Referências
|











![{\displaystyle A=\left[a_{ij}\right]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/9d811d5a7227a39043a1cd37979acd1f711799ac)

















![{\displaystyle {\begin{aligned}M&={\frac {1}{2|E|}}\sum _{i,j=1}^{N}A_{ij}\delta _{ij}-{\frac {1}{2|E|}}{\frac {\deg(i)\deg(j)}{2|E|}}\delta _{ij}\\&=\sum _{c=1}^{m}{\frac {1}{2|E|}}\sum _{i,j\in S_{c}}A_{ij}-\sum _{c=1}^{m}{\frac {1}{(2|E|)^{2}}}\sum _{i,j\in S_{c}}\deg(i)\deg(j)\\&=\sum _{c=1}^{m}{\frac {L(S_{c})}{|E|}}-\sum _{c=1}^{m}{\frac {\deg(S_{c})^{2}}{4|E|^{2}}}\\&=\sum _{c=1}^{m}\left[{\frac {L(S_{c})}{|E|}}-\left({\frac {\deg(S_{c})}{2|E|}}\right)^{2}\right].\end{aligned}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/585b676538b69741b45e3ab6ac68a0ec074716a1)




