|
Ribozima de cabeza de martillo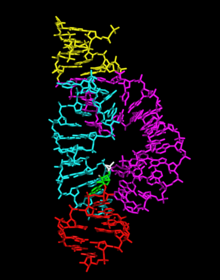 La ribozima de cabeza de martillo o hammerhead es un motivo de ARN que cataliza reacciones de corte y ligación reversibles en un sitio específico dentro de la molécula de ARN. Es uno de los distintos tipos de pequeños ARNs catalíticos ( ribozimas ) descritos en la naturaleza. Desde su descubrimiento, se ha empleado como sistema modelo para la investigación sobre la estructura y las propiedades del ARN y las ribozimas en general, y se utiliza para experimentos de escisión dirigida de diferentes ARNs, algunos de los cuales tienen posibles aplicaciones biotecnológicas y terapéuticas. Las ribozimas del cabeza de martillo, llamadas así por la semejanza de los primeros diagramas de estructura secundaria con un la cabeza de un tiburón martillo, [1] se descubrieron originalmente en dos clases de ARN circulares infecciosos de plantas: los ARN satélites y los viroides . También se conocen ribozimas de esta clase en algunas familias de retrotransposones de plantas y animales, incluidas las retrozimas . [2] El motivo del ribozima cabeza de martillo se ha reportado de manera ubicua en linajes a lo largo del árbol de la vida. [3] [4] Las reacciones de autocorte, descritas por primera vez en 1986, [5] [6] son parte de un mecanismo de replicación de círculo rodante . La secuencia de cabeza de martillo es suficiente para la autoescisión [7] y actúa formando una estructura terciaria tridimensional conservada. En su estado natural, un ARN de cabeza de martillo es una hebra de ARN de simple cadena. Aunque la escisión se produce en ausencia de enzimas proteicas, el ARN de cabeza de martillo en la naturaleza no actúa como catalizador, ya que es consumido por la reacción, es decir, realiza el autocorte una vez y no puede catalizar múltiples recambios. Las construcciones de cabeza de martillo actuando en trans se pueden diseñar de tal manera que consistan en dos cadenas de ARN que interactúan, con una cadena que compone una ribozima de cabeza de martillo que escinde la otra cadena. [8] La cadena que se corta se puede suministrar en exceso, y se puede demostrar que la actividad en múltiple recambio obedece a la cinética de Michaelis-Menten, [8][cita requerida]</link> típico de la cinética enzimática de proteínas . Estos constructos se emplean normalmente para experimentos in vitro, y el término "ARN cabeza de martillo" se ha convertido en la práctica en sinónimo del término más frecuentemente utilizado " ribozima cabeza de martillo". La secuencia mínima de ribozima de cabeza de martillo que actúa en trans y que es catalíticamente activa consta de tres tallos de doble hebra que flanquean un núcleo central de 15 nucleótidos conservados esenciales para la actividad catalítica de la ribozima. Estas construcciones de ribozimas de cabeza de martillo exhiben in vitro una tasa de recambio (k cat) de aproximadamente 1 molécula/minuto y una Km del orden de 10 nanomolar. Gracias a su pequeño tamaño, su química de corte minuciosamente investigada, su estructura cristalina conocida y su relevancia biológica hacen que la ribozima cabeza de martillo sea la pequeña ribozima mejor caracterizada.. Las ribozimas de cabeza de martillo pueden llegar a utilizarse en el futuro como agente terapéuticos, como tanto detectando secuencias de ARN definidas y degradándolos, como biosensores o para aplicaciones en genómica funcional y descubrimiento de genes. [9] Distribución de especiesEn 1986 se descubrieron las primeras ribozimas de cabeza de martillo en patógenos de ARN de plantas como viroides y satélites virales. [5] [6] Un año más tarde, también se detectó la existencia de ribozimas de cabeza de martillo en el ADN satélite de los genomas del tritón y salamandras. [10] Posteriormente se encontraron nuevos ejemplos de estas ribozimas en los genomas de ADN de organismos tan alejados evolutivamente como los esquistosomas, [11] los grillos de cueva, [12] la planta Arabidopsis thaliana [13] y algunos mamíferos como roedores o el ornitorrinco. [14] En 2010, se descubrió que las ribozimas de cabeza de martillo se encuentran en realidad en una amplia variedad de genomas que iban desde bacterias y fagos a todo tipo de eucariotas, [15] e incluso en el propio genoma humano. [16] Informes similares confirmaron y ampliaron estas observaciones, [17] [18] [19] revelando que la ribozima cabeza de martillo es un ARN catalítico ubicuo en todos los reinos de la vida. [20] La mayoría de las ribozimas de cabeza de martillo encontradas en genomas de ADN eucariótico de plantas y animales están relacionadas con un tipo de retroelementos (SINE) llamados retrozimas, y que se expresan activamente como pequeños ARNs circulares en plantas y animales. [21] Sin embargo, en los genomas de todos los amniotas se puede encontrar también un grupo excepcional de ribozimas de cabeza martillo sorprendentemente conservados. [16] Estas ribozimas cabeza de martillo (las llamadas HH9 [22] y HH10 [23]) están en los intrones de unos pocos genes específicos y apuntan a un papel biológico preservado durante la biosíntesis de pre-ARNm. [24] En 2021 se descubrió que novedosos genomas de ARN circular encontrados en diversos animales, pero muy similares al virus de la hepatitis D humano, codificaban ribozimas de cabeza de martillo muy similares a las de los viroides y los satélites virales de plantas. [25] Búsquedas bioinformáticas masivas de agentes tipo deltavirus descubrieron cientos de nuevos genomas de ARN circular conteniendo tanto ribozimas de cabeza de martillo como de tipo deltavirus, lo que indica que no solo esta ribozima sino también los pequeños ARN circulares con ribozimas en general son moléculas omnipresentes en la biosfera. [26] La ribozima cabeza de martillo lleva a cabo una reacción química muy simple que produce la rotura de la cadena de sustrato del ARN, específicamente en la posición C17 de la ribozima, el nucleótido del sitio de escisión. Aunque la escisión del ARN a menudo se denomina hidrólisis, el mecanismo empleado en realidad no implica la adición de agua . Más bien, la reacción de escisión es simplemente una isomerización que consiste en la reorganización del enlace fosfodiéster de enlace. Es la misma reacción, químicamente, que ocurre con la degradación del ARN mediada por bases aleatorias, excepto que es altamente específica del sitio y la velocidad se acelera 10.000 veces o más.  La reacción de escisión es una reacción de isomerización de fosfodiéster que se inicia mediante la abstracción del protón 2'-hidroxilo de la ribosa del sitio de escisión del 2'-oxígeno, que luego se convierte en el nucleófilo atacante en una reacción "en línea" o similar a S N 2(P), aunque no se sabe si este protón se elimina antes o durante el paso químico de la reacción de escisión de cabeza de martillo. (La reacción de escisión técnicamente no es bimolecular, pero se comporta de la misma manera que una reacción SN2(P); sufre una inversión de configuración posterior a la formación de un estado de transición que consiste en un oxifosfrano pentacoordinado). Los oxígenos del grupo atacante y saliente ocuparán las dos posiciones axiales en la estructura de estado de transición bipiramidal trigonal como se requiere para un mecanismo de reacción tipo S N 2 . El producto 5', como resultado de este mecanismo de reacción de escisión, posee un extremo fosfato cíclico 2',3', y el producto 3' posee un extremo 5'-OH, como ocurre con la escisión alcalina no enzimática del ARN. Por lo tanto, la reacción es reversible, ya que el fosfato escindible sigue siendo un fosfodiéster y puede en principio actuar como sustrato para la ligación mediada por el ARN cabeza de martillo sin necesidad de ATP o una fuente de energía exógena similar. [27] La reacción catalizada por el ribozima cabeza de martillo, a diferencia de la escisión alcalina no enzimática formalmente idéntica del ARN, es una reacción de escisión altamente específica de la secuencia con una tasa de recambio típica de aproximadamente 1 molécula de sustrato por molécula de enzima por minuto a pH 7,5 en 10 mM de Mg 2+ (las denominadas "condiciones de reacción estándar" para la secuencia mínima del ARN cabeza de martillo), dependiendo de la secuencia del constructo particular del ribozima cabeza de martillo medido. Esto representa una mejora de la tasa de aproximadamente 10.000 veces con respecto a la escisión no enzimática del ARN. Originalmente se pensaba que todas las ribozimas eran metaloenzimas . Se asumió que los iones metálicos divalentes como Mg 2+ tenían dos funciones: promover el plegamiento adecuado del ARN y formar el núcleo catalítico. [28] Como el ARN en sí no contenía suficiente variación en los grupos funcionales, se pensó que los iones metálicos desempeñaban un papel en el sitio activo, como se sabía acerca de las proteínas. El mecanismo propuesto para el ion Mg2+ fue: la desprotonación del grupo 2'-OH por un complejo Magnesium.agua.hidroxi unido por el oxígeno pro-R en el sitio de escisión del fosfato, seguido por un ataque nucleofílico del 2'-alcaóxido resultante sobre el fosfato escindible formando un intermedio de fosfato pentacoordinado. El último paso es la salida del grupo saliente 5', produciendo un fosfato 2',3'-cíclico con una configuración invertida. [29] Se asumió que los iones de magnesio hexahidratados, que existen en equilibrio con el hidróxido de magnesio, podrían desempeñar los papeles de ácido general y base general, de una manera análoga a los desempeñados por dos histidinas en la ARNasa A. También se ha propuesto un papel adicional para los iones metálicos divalentes en forma de estabilización electrostática del estado de transición . No es una metaloenzimaEn 1998 se descubrió [30] que la ribozima en forma de cabeza de martillo, así como la ribozima VS y la ribozima en horquilla, no requieren la presencia de iones metálicos para la catálisis, siempre que haya una concentración suficientemente alta de catión monovalente para permitir que el ARN se pliegue. Este descubrimiento sugirió que el ARN mismo, en lugar de servir como un andamio inerte y pasivo para la unión de iones metálicos divalentes químicamente activos, está íntimamente involucrado en la química de la catálisis. Los últimos resultados estructurales, descritos posteriormente confirman que dos nucleótidos invariantes, G12 y G8, están posicionados de manera consistente con sus roles como base general y ácido general en la reacción de escisión de la cabeza de martillo. Ribozima mínimaLa secuencia mínima de cabeza de martillo que se requiere para la reacción de autocorte incluye aproximadamente 13 nucleótidos "centrales" conservados o invariantes, la mayoría de los cuales no participan en la formación de pares de bases Watson-Crick canónicos. La región central está flanqueada por los tallos I, II y III, que en general están formados por pares de bases Watson-Crick canónicos, pero que por lo demás no están restringidos con respecto a la secuencia. La tasa de recambio catalítico de las ribozimas mínimas de cabeza de martillo es de ~ 1/min (comúnmente se observa un rango de 0,1/min a 10/min, dependiendo de las secuencias no conservadas y las longitudes de los tres tallos helicoidales) en condiciones de reacción estándar de alto Mg 2+ (~10 mM), pH 7,5 y 25 °C. Gran parte del trabajo experimental realizado sobre las ribozimas del pez martillo ha utilizado una construcción mínima. ARNs de cabeza de martillo tipo I, tipo II y tipo IIIEstructuralmente, la ribozima cabeza de martillo está compuesta por tres hélices con pares de bases, separadas por enlaces cortos de secuencias conservadas. Estas hélices se llaman I, II y III. Las ribozimas cabeza de martillo se pueden clasificar en tres tipos según en qué hélice se encuentran los extremos 5' y 3'. Si los extremos 5' y 3' de la secuencia contribuyen al tallo I, entonces es un ribozima cabeza de martillo tipo I, al tallo II es un tipo II y al tallo III entonces es una ribozima cabeza de martillo tipo III. De los tres tipos topológicos posibles, el tipo I se puede encontrar en los genomas de procariotas, eucariotas y en menor medida, algunos patógenos de plantas de ARN, mientras que el tipo II solo se ha descrito en procariotas [18] [19] y el tipo III se encuentra principalmente en genomas de plantas, patógenos de RNA de plantas, animales y hongos, así como en procariotas. [20] [24] La ribozima cabeza de martillo de longitud completa consta de elementos de secuencia adicionales en los bucles de los tallos I y II que permiten la formación de contactos terciarios adicionales. Las interacciones terciarias estabilizan la conformación activa de la ribozima, lo que da como resultado tasas de escisión hasta 1000 veces mayores que las de las secuencias de cabeza de martillo mínimas correspondientes. [31] [32]  La ribozima de cabeza de martillo mínima ha sido estudiada exhaustivamente por bioquímicos y enzimólogos, así como por cristalógrafos de rayos X, espectroscopistas de RMN y otros profesionales de técnicas biofísicas. La primera estructura tridimensional de una ribozima cabeza de martillo se publicó en 1994 en forma de una estructura cristalina de rayos X de una ribozima cabeza de martillo unida a un análogo de sustrato de ADN, publicada en Nature por Pley, Flaherty y McKay. [33] Posteriormente, Scott, Finch y Klug publicaron una estructura mínima de ribozima de cabeza de martillo completamente de ARN en Cell a principios de 1995. [34] La ribozima cabeza de martillo mínima está compuesta por tres hélices con pares de bases, separadas por enlaces cortos de secuencia conservada como se muestra en la estructura cristalina . [34] Estas hélices se llaman I, II y III. El giro de uridina conservado une las hélices I con la II, y generalmente contiene la secuencia CUGA. Las hélices II y III están unidas por una secuencia GAAA. La reacción de escisión ocurre entre las hélices III y I, y normalmente es una C. La estructura de una ribozima de cabeza de martillo de longitud completa muestra que existen interacciones extensas entre el bucle del tallo II y el tallo I. [35]
Por estas razones, los dos conjuntos de experimentos (bioquímico vs. cristalográfico) parecían no sólo estar en desacuerdo, sino ser completamente irreconciliables, generando una discordia en el campo. Nunca se presentó con éxito evidencia convincente para descartar ninguno de los dos conjuntos de resultados experimentales, haciéndose muchas afirmaciones en contrario y a favor de cada uno de ellos.  En 2006 se obtuvo una estructura cristalina de resolución de 2,2 Å del ribozima cabeza de martillo de longitud completa. Esta nueva estructura (mostrada a la derecha) parece resolver la más preocupante de las discrepancias previas. En particular, C17 ahora está posicionado para el ataque en línea, y los residuos invariantes C3, G5, G8 y G12 parecen estar involucrados en interacciones clave para la catálisis. Además, se observa que los fosfatos A9 y escindibles están separados por 4,3 Å, lo que concuerda con la idea de que, cuando se modifican, estos fosfatos podrían unirse a un solo ion metálico tiófilo. La estructura también revela cómo dos residuos invariantes, G-12 y G-8, se posicionan dentro del sitio activo, en consonancia con su papel previamente propuesto en la catálisis ácido/base. G12 está dentro de la distancia de enlace de hidrógeno al 2'–O de C17, el nucleófilo en la reacción de escisión, y la ribosa de G8 se enlaza con el hidrógeno al grupo saliente 5'-O. (ver abajo), mientras que la base de nucleótidos de G8 forma un par Watson-Crick con el invariante C3. Esta disposición sugiere que G12 es la base general en la reacción de corte de la ribozima y que G8 actuaría como el ácido general, en consonancia con observaciones bioquímicas anteriores. De este modo, la estructura cristalina del ribozima cabeza de martillo de longitud completa resuelve claramente todas las discrepancias que parecían irreconciliables con las estructuras cristalinas anteriores del ribozima cabeza de martillo mínimo. Estructura y catálisis Las interacciones terciarias en el ribozima cabeza de martillo de longitud completa estabilizan lo que parece ser la conformación activa. El nucleófilo, el oxígeno 2' del nucleótido del sitio de escisión, C17, está alineado casi perfectamente para un ataque en línea (la reacción S N 2(P)). G12 está ubicado dentro de la distancia de enlace de hidrógeno de este nucleófilo y, por lo tanto, sería capaz de abstraer un protón del oxígeno 2' si el propio G12 se desprotona. El 2'-OH de G8 forma un enlace de hidrógeno con el oxígeno del grupo saliente 5' y, por lo tanto, potencialmente puede suministrar un protón a medida que se acumula carga negativa en el oxígeno 5' de la ribosa de A1.1. La explicación más probable es entonces que G12, en la forma desprotonada, es la base general y la ribosa de G8 es el ácido general. El pKa cinético aparente El pKa de la ribozima cabeza de martillo es 8,5, mientras que el pKa de la guanosina es aproximadamente 9,5. Es posible que el pKa de la posición G12 se altere de 9,5 a 8,5 en el núcleo catalítico de cabeza de martillo.  Si la G8 invariante se cambia a C8, se elimina la catálisis de cabeza de martillo. Sin embargo, un mutante doble G8C+C3G que mantiene el par de bases G8-C3 que se encuentra en la ribozima de cabeza de martillo de longitud completa restaura la mayor parte de la actividad catalítica. También se ha observado que la posición 2'-OH de G8 es esencial para la catálisis, lo que sugiere que es te hidroxilo es crucial para el mecanismo catalítico.
Aplicaciones terapéuticasSe están probando ribozimas de cabeza de martillo modificadas como agentes terapéuticos. [36] Se están estudiando ARNs sintéticos que contienen secuencias complementarias al ARNm SOD1 mutante y secuencias necesarias para formar la estructura catalítica en forma de cabeza de martillo como posible terapia para la esclerosis lateral amiotrófica. Se ha demostrado que los adenovirus con ribozimas cabeza de martillo son potentes en el tratamiento del cáncer tanto in vitro como in vivo. [37] El uso terapéutico de las ribozimas de cabeza de martillo que se escinden trans se ha visto mermado por su baja actividad in vivo . El potencial catalítico de las ribozimas de cabeza de martillo que escinden en trans se puede recuperar in vivo. Ya existen algunas ribozimas de cabeza de martillo que están próximas a tener aplicación clínica. [9] Referencias
|