|
Raza y genética
Los investigadores han investigado la relación entre raza y genética como parte de los esfuerzos para comprender cómo la biología puede contribuir o no a la categorización racial humana. Muchas construcciones de raza están asociadas con rasgos fenotípicos y ascendencia geográfica, y estudiosos como Carlos Linneo han propuesto modelos científicos para la organización de la raza desde al menos el siglo XVIII. Tras el descubrimiento de la genética mendeliana y el mapeo del genoma humano, las preguntas sobre la biología de la raza a menudo se han enmarcado en términos de genética.[1] Se ha empleado una amplia gama de métodos de investigación para examinar los patrones de variación humana y sus relaciones con la ascendencia y los grupos raciales, incluidos estudios de rasgos individuales,[2] estudios de grandes poblaciones y grupos genéticos[3] y estudios de factores genéticos de riesgo de enfermedad.[4] La investigación sobre la raza y la genética también ha sido criticada por surgir o contribuir al racismo científico. Algunos han interpretado los estudios genéticos de rasgos y poblaciones como evidencia para justificar las desigualdades sociales asociadas con la raza,[5] a pesar de que se ha demostrado que los patrones de variación humana son principalmente clinales, siendo el código genético humano aproximadamente un 99,9% idéntico entre individuos y sin límites claros entre los grupos.[6][1] Existe un debate científico en curso sobre la definición y el significado de raza en la investigación genética y biomédica. Algunos investigadores sostienen que la raza puede actuar como un "proxy" de la ascendencia genética porque los individuos de la misma categoría racial pueden compartir una ascendencia común, pero otros abogan por distinguir entre la biología y los factores sociales, políticos, culturales y económicos que contribuyen a la raza, como se entiende más comúnmente.[7] Variación genéticaLa variación genética surge de mutaciones, de la selección natural, de la migración entre poblaciones (flujo genético) y de la reorganización de genes a través de la reproducción sexual.[8] Las mutaciones conducen a un cambio en la estructura del ADN, ya que se reorganiza el orden de las bases. Como resultado, se codifican diferentes proteínas polipeptídicas. Algunas mutaciones pueden ser positivas y pueden ayudar al individuo a sobrevivir de manera más eficaz en su entorno. La variación se contrarresta mediante la selección natural y la deriva genética; nótese también el efecto fundador, cuando un pequeño número de fundadores iniciales establece una población que, por tanto, comienza con un grado correspondientemente pequeño de variación genética.[9] La herencia epigenética implica cambios hereditarios en el fenotipo (apariencia) o la expresión génica, causados por mecanismos distintos a los cambios en la secuencia del ADN.[10] Los fenotipos humanos son altamente poligénicos (dependen de la interacción de muchos genes) y están influenciados tanto por el medio ambiente como por la genética. La diversidad de nucleótidos se basa en mutaciones únicas, polimorfismos de nucleótido único (SNP). La diversidad de nucleótidos entre humanos es de aproximadamente 0,1 % (una diferencia por mil nucleótidos entre dos humanos elegidos al azar). Esto equivale a aproximadamente tres millones de SNP (ya que el genoma humano tiene alrededor de tres mil millones de nucleótidos). Se estima que hay diez millones de SNP en la población humana.[11] La investigación ha demostrado que la variación no SNP (estructural) explica más variación genética humana que la diversidad de un solo nucleótido. La variación estructural incluye la variación del número de copias y los resultados de eliminaciones, inversiones, inserciones y duplicaciones. Se estima que aproximadamente del 0,4 al 0,6 por ciento de los genomas de personas no relacionadas difieren.[12][13] ¿Existe una base genética para la raza?Gran parte de la investigación científica se ha organizado en torno a la cuestión de si existe o no una base genética para la raza. Según Luigi Luca Cavalli-Sforza,
Métodos de búsquedaLos científicos que investigan la variación humana han utilizado una serie de métodos para caracterizar cómo varían las diferentes poblaciones. Estudios iniciales de rasgos, proteínas y genesVéase también: Raza (clasificación de los seres humanos)
Los primeros intentos de clasificación racial midieron los rasgos de la superficie, particularmente el color de la piel, el color y la textura del cabello, el color de los ojos y el tamaño y la forma de la cabeza. (Las mediciones de este último a través de la craneometría fueron desacreditadas repetidamente a fines del siglo XIX y mediados del XX debido a la falta de correlación de los rasgos fenotípicos con la categorización racial.[20]) En realidad, la adaptación biológica juega el papel más importante en estas características corporales y tipo de piel. Un puñado relativo de genes explica los factores heredados que dan forma a la apariencia de una persona.[21][22] Se estima que los seres humanos tienen entre 19 000 y 20 000 genes codificadores de proteínas humanas.[23] Richard Sturm y David Duffy describen 11 genes que afectan la pigmentación de la piel y explican la mayoría de las variaciones en el color de la piel humana, las más importantes de las cuales son MC1R, ASIP, OCA2 y TYR.[24] Existe evidencia de que hasta 16 genes diferentes podrían ser responsables del color de ojos en humanos; sin embargo, los dos genes principales asociados con la variación del color de los ojos son OCA2 y HERC2, y ambos están localizados en el cromosoma 15.[25] Análisis de proteínas sanguíneas y genética entre grupos  Antes del descubrimiento del ADN, los científicos usaban proteínas sanguíneas (los sistemas de grupos sanguíneos humanos) para estudiar la variación genética humana. La investigación de Ludwik y Hanka Herschfeld durante la Primera Guerra Mundial encontró que la incidencia de los grupos sanguíneos A y B difería según la región; por ejemplo, entre los europeos, el 15 por ciento eran del grupo B y el 40 por ciento del grupo A. Los europeos del este y los rusos tenían una mayor incidencia del grupo B; la gente de la India tuvo la mayor incidencia. Los Herschfelds concluyeron que los humanos comprenden dos "razas bioquímicas", que se originan por separado. Se planteó la hipótesis de que estas dos razas se mezclaron más tarde, lo que resultó en los patrones de los grupos A y B. Esta fue una de las primeras teorías de las diferencias raciales en incluir la idea de que la variación humana no se correlaciona con la variación genética. Se esperaba que los grupos con proporciones similares de grupos sanguíneos estuvieran más estrechamente relacionados, pero en cambio, a menudo se encontró que los grupos separados por grandes distancias (como los de Madagascar y Rusia), tenían incidencias similares.[26] Más tarde se descubrió que el sistema de grupos sanguíneos ABO no solo es común a los humanos, sino que se comparte con otros primates,[27] y probablemente es anterior a todos los grupos humanos.[28] En 1972, Richard Lewontin realizó un análisis estadístico FST utilizando 17 marcadores (incluidas las proteínas del grupo sanguíneo). Encontró que la mayoría de las diferencias genéticas entre humanos (85,4 por ciento) se encontraron dentro de una población, el 8,3 por ciento se encontró entre poblaciones dentro de una raza y el 6,3 por ciento se encontró para diferenciar razas (caucásicos, africanos, mongoloides, aborígenes del sur de Asia, amerindios, aborígenes australianos y provenientes de Oceanía en su estudio). Desde entonces, otros análisis han encontrado valores de FST del 6 al 10 por ciento entre grupos humanos continentales, del 5 al 15 por ciento entre diferentes poblaciones en el mismo continente y del 75 al 85 por ciento dentro de las poblaciones.[29][30][31][32][33] Este punto de vista ha sido confirmado por la Asociación Americana de Antropología y la Asociación Americana de Antropólogos Físicos desde entonces.[34] Críticas al análisis de proteínas en sangreSi bien reconoce la observación de Lewontin de que los humanos son genéticamente homogéneos, A. W. F. Edwards en su artículo de 2003 Human Genetic Diversity: Lewontin's Falacy argumentó que la información que distingue a las poblaciones entre sí está oculta en la estructura de correlación de las frecuencias alélicas, lo que hace posible clasificar a los individuos utilizando métodos matemáticos. Edwards argumentó que incluso si la probabilidad de clasificar erróneamente a un individuo en función de un solo marcador genético es tan alta como el 30 por ciento (como informó Lewontin en 1972), la probabilidad de clasificación errónea se acerca a cero si se estudian simultáneamente suficientes marcadores genéticos. Edwards vio el argumento de Lewontin como basado en una postura política, negando las diferencias biológicas para defender la igualdad social.[35] El artículo de Edwards se reimprime, comentado por expertos como Noah Rosenberg y se le da más contexto en una entrevista con el filósofo de la ciencia Rasmus Grønfeldt Winther en una antología reciente.[36] Edwards criticó el artículo de Lewontin, ya que tomó 17 rasgos diferentes y los analizó de forma independiente, sin mirarlos junto con ninguna otra proteína. Por lo tanto, hubiera sido bastante conveniente para Lewontin llegar a la conclusión de que el naturalismo racial no es sostenible, según su argumento.[37] Sesardic también fortaleció la visión de Edwards, ya que usó una ilustración que se refiere a cuadrados y triángulos, y mostró que si miras un rasgo de forma aislada, lo más probable es que sea un mal predicador de a qué grupo pertenece el individuo.[38] En contraste, en un artículo de 2014, reimpreso en el volumen de Edwards Cambridge University Press de 2018, Rasmus Grønfeldt Winther sostiene que "La falacia de Lewontin" es efectivamente un nombre inapropiado, ya que en realidad hay dos conjuntos diferentes de métodos y preguntas en juego al estudiar la estructura poblacional genómica de nuestra especie: "partición de varianza" y "análisis de agrupamiento". Según Winther, son "las dos caras de la misma moneda matemática" y ninguna "implica necesariamente nada sobre la realidad de los grupos humanos".[39]  Estudios actuales de genética de poblacionesLos investigadores actualmente usan pruebas genéticas, que pueden involucrar cientos (o miles) de marcadores genéticos o todo el genoma. EstructuraExisten varios métodos para examinar y cuantificar subgrupos genéticos, incluido el análisis de conglomerados y el análisis de componentes principales. Los marcadores genéticos de los individuos se examinan para encontrar la estructura genética de una población. Si bien los subgrupos se superponen cuando se examinan variantes de un solo marcador, cuando se examinan varios marcadores, los diferentes subgrupos tienen una estructura genética promedio diferente. Un individuo puede describirse como perteneciente a varios subgrupos. Estos subgrupos pueden ser más o menos distintos, dependiendo de cuánta superposición haya con otros subgrupos.[40] En el análisis de conglomerados, el número de conglomerados para buscar K se determina de antemano; lo distintos que son los grupos varía. Los resultados obtenidos de los análisis de conglomerados dependen de varios factores:
Se han publicado estudios recientes utilizando un número creciente de marcadores genéticos.[42][45][46][47][48][49] DistanciaLa distancia genética es la divergencia genética entre especies o poblaciones de una especie. Puede comparar la similitud genética de especies relacionadas, como humanos y chimpancés. Dentro de una especie, la distancia genética mide la divergencia entre subgrupos. La distancia genética se correlaciona significativamente con la distancia geográfica entre poblaciones, un fenómeno conocido a veces como "aislamiento por distancia".[50] La distancia genética puede ser el resultado de límites físicos que restringen el flujo de genes, como islas, desiertos, montañas o bosques. La distancia genética se mide mediante el índice de fijación (FST). El FST es la correlación de alelos elegidos al azar en un subgrupo con una población más grande. A menudo se expresa como una proporción de la diversidad genética. Esta comparación de la variabilidad genética dentro (y entre) poblaciones se utiliza en genética de poblaciones. Los valores van de 0 a 1; cero indica que las dos poblaciones se cruzan libremente y uno indicaría que dos poblaciones están separadas. Muchos estudios sitúan la distancia FST media entre razas humanas en aproximadamente 0,125. Henry Harpending argumentó que este valor implica a escala mundial un "parentesco entre dos individuos de la misma población humana es equivalente al parentesco entre abuelos y nietos o entre medio hermanos". De hecho, las fórmulas derivadas del artículo de Harpending implican que dos individuos no emparentados de la misma raza tienen un coeficiente de parentesco más alto (0.125) que un individuo y su medio hermano de raza mixta (0.109).[51] Críticas a FSTAunque se reconoce que el FST sigue siendo útil, varios científicos han escrito sobre otros enfoques para caracterizar la variación genética humana.[52][53][54] Long y Kittles afirmaron que el FST no logró identificar variaciones importantes y que cuando el análisis incluye solo humanos, FST = 0.119, pero agregar chimpancés lo aumenta solo a FST = 0.183. Se ha argumentado que una estimación de FST de 0,10-0,15 no descarta una base genética para las diferencias fenotípicas entre grupos y que una estimación de FST baja implica poco sobre el grado en que los genes contribuyen a las diferencias entre grupos. Pearse y Crandall escribieron que las cifras de FST no pueden distinguir entre una situación de alta migración entre poblaciones con un tiempo de divergencia prolongado y una de historia compartida relativamente reciente pero sin flujo de genes en curso. En su artículo de 2015, Keith Hunley, Graciela Cabana y Jeffrey Long (que habían criticado previamente la metodología estadística de Lewontin con Rick Kittles[34]) recalculan la distribución de la diversidad humana utilizando un modelo más complejo que Lewontin y sus sucesores.
Los antropólogos (como C. Loring Brace),[56] filósofo Jonathan Kaplan y el genetista Joseph L. Graves[57] han argumentado que si bien es posible encontrar variaciones biológicas y genéticas que se correspondan aproximadamente con la raza, esto es cierto para casi todas las poblaciones geográficamente distinta: la estructura de grupos de datos genéticos depende de las hipótesis iniciales del investigador y de las poblaciones muestreadas. Cuando se toman muestras de grupos continentales, los racimos se vuelven continentales; con otros patrones de muestreo, los conglomerados serían diferentes. Weiss y Fullerton señalan que si se muestrearan solo islandeses, mayas y maoríes, se formarían tres grupos distintos; todas las demás poblaciones estarían compuestas por mezclas genéticas de material maorí, islandés y maya.[58] Por lo tanto, Kaplan concluye que, si bien las diferencias en frecuencias alélicas particulares se pueden usar para identificar poblaciones que se corresponden libremente con las categorías raciales comunes en el discurso social occidental, las diferencias no tienen más importancia biológica que las diferencias encontradas entre cualquier población humana (por ejemplo, el Español y portugués).[59] Análisis histórico y geográficoLa estructura genética de la población actual no implica que los diferentes grupos o componentes indiquen solo un hogar ancestral por grupo; por ejemplo, un grupo genético en los Estados Unidos comprende hispanos con ascendencia europea, nativa americana y africana.[41] Los análisis geográficos intentan identificar los lugares de origen, su importancia relativa y las posibles causas de variación genética en un área. Los resultados se pueden presentar como mapas que muestran la variación genética. Se ha argumentado que si se investigan las variaciones genéticas, a menudo corresponden a migraciones de población debido a nuevas fuentes de alimentos, transporte mejorado o cambios en el poder político. Por ejemplo, en Europa, la dirección más significativa de variación genética corresponde a la expansión de la agricultura desde el Medio Oriente a Europa entre hace 10,000 y 6,000 años.[60] Este análisis geográfico funciona mejor en ausencia de migraciones rápidas y a gran escala recientes. Los análisis históricos utilizan las diferencias en la variación genética (medidas por distancia genética) como un reloj molecular que indica la relación evolutiva de especies o grupos, y se pueden utilizar para crear árboles evolutivos que reconstruyen las separaciones de poblaciones.[60] Los resultados de la investigación de la ascendencia genética se apoyan si están de acuerdo con los resultados de la investigación de otros campos, como la lingüística o la arqueología.[60] También se ha argumentado que existe una correspondencia entre las familias de lenguas encontradas en la investigación lingüística y el árbol de población. Por lo general, existen distancias genéticas más cortas entre poblaciones que utilizan lenguas de la misma familia lingüística. También se encuentran excepciones a esta regla, por ejemplo Sami, que están genéticamente asociados con poblaciones que hablan idiomas de otras familias lingüísticas. Los Sami hablan una lengua urálica, pero genéticamente son principalmente europeos. Se argumenta que esto es el resultado de la migración (y el mestizaje) con los europeos mientras se conserva su idioma original. También existe un acuerdo entre las fechas de investigación en arqueología y las calculadas utilizando la distancia genética.[42] Estudios de autoidentificaciónSe ha encontrado que si bien los grupos de marcadores genéticos estaban correlacionados con algunos conceptos tradicionales de raza, las correlaciones eran imperfectas e imprecisas debido a la naturaleza continua y superpuesta de la variación genética, y señalaron que la ascendencia, que puede determinarse con precisión, no es equivalente al concepto de raza.[61] Un estudio de 2005 utilizó 326 marcadores genéticos para determinar los grupos genéticos. Los 3.636 sujetos, de Estados Unidos y Taiwán, se autoidentificaron como pertenecientes a grupos étnicos blancos, afroamericanos, asiáticos orientales o hispanos. El estudio encontró "una correspondencia casi perfecta entre el grupo genético y SIRE para los principales grupos étnicos que viven en los Estados Unidos, con una tasa de discrepancia de solo el 0,14 por ciento".[41] Se encontró un acuerdo "esencialmente perfecto" entre 51 poblaciones de origen autoidentificadas y la estructura genética de la población, utilizando 650.000 marcadores genéticos. La selección de marcadores genéticos informativos permitió una reducción a menos de 650, manteniendo una precisión casi total.[62] La correspondencia entre los grupos genéticos en una población (como la población actual de EE. UU.) y la raza o los grupos étnicos autoidentificados no significa que dicho grupo (o grupo) corresponda a un solo grupo étnico. Los afroamericanos tienen una mezcla genética europea estimada del 20 al 25 por ciento; los hispanos tienen ascendencia europea, nativa americana y africana.[41] En Brasil ha habido una gran mezcla entre europeos, amerindios y africanos. Como resultado, las diferencias en el color de la piel dentro de la población no son graduales y existen asociaciones relativamente débiles entre la raza autoinformada y la ascendencia africana.[63][64] La autoclasificación etnoracial en los brasileños ciertamente no es aleatoria con respecto a la ascendencia individual del genoma, pero la fuerza de la asociación entre el fenotipo y la proporción mediana de ascendencia africana varía en gran medida entre la población.[65] Crítica de los estudios y conglomerados de la distancia genética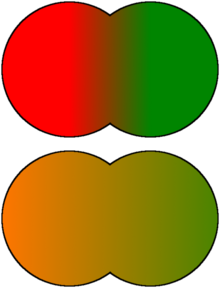 Las distancias genéticas generalmente aumentan continuamente con la distancia geográfica, lo que hace que una línea divisoria sea arbitraria. Cualesquiera dos asentamientos vecinos exhibirán alguna diferencia genética entre sí, lo que podría definirse como una raza. Por tanto, los intentos de clasificar las razas imponen una discontinuidad artificial a un fenómeno natural. Esto explica por qué los estudios sobre la estructura genética de la población arrojan resultados variables, según la metodología. Rosenberg ha argumentado, basándose en el análisis de conglomerados de las 52 poblaciones en el Panel de diversidad genética humana, que las poblaciones no siempre varían continuamente y que la estructura genética de una población es consistente si se incluyen suficientes marcadores genéticos (y sujetos).
También escribieron, con respecto a un modelo con cinco grupos correspondientes a África, Eurasia (Europa, Medio Oriente y Asia Central /Meridional), Asia Oriental, Oceanía y América:
Esto se aplica a las poblaciones en sus hogares ancestrales cuando las migraciones y el flujo de genes fueron lentos; las migraciones grandes y rápidas exhiben características diferentes. Afirmando,
 El análisis de conglomerados ha sido criticado porque el número de conglomerados a buscar se decide de antemano, con diferentes valores posibles (aunque con diferentes grados de probabilidad).[66] El análisis de componentes principales no decide de antemano cuántos componentes buscar.[67] El estudio de 2002 de Noah Rosenberg ejemplifica por qué los significados de estos agrupamientos son discutibles. El estudio muestra que en el análisis de conglomerados K = 5, los conglomerados genéticos se mapean aproximadamente en cada una de las cinco regiones geográficas principales. En 2005 se obtuvieron resultados similares en estudios adicionales[68] Sin embargo, además de los cinco conglomerados supuestamente geográficos principales, un sexto grupo, los Kalash, un grupo étnico minoritario en Pakistán, comenzó a aparecer a partir de K = 6. El naturalista racial Nicholas Wade considera que los resultados "no tienen sentido genético o geográfico". Por lo tanto, se omiten en su libro A Troublesome Inheritance a favor del análisis de conglomerados K = 5. Este sesgo, sin embargo, refleja cómo la investigación es inherentemente defectuosa. La población de muestra se elige teniendo en cuenta la representación geográfica y los conceptos populares de raza, en lugar de tener en cuenta la diversidad genética dentro de las diferentes regiones geográficas. Los Kalash no encajaban en el patrón general porque había sido una población genéticamente aislada que se reflejó en este estudio. Potencialmente numerosos grupos genéticamente modificados, como los Sentineleses no contactados, no están representados en el estudio. Crítica de los marcadores informativos de ascendenciaLos marcadores informativos de ascendencia (AIM) son una tecnología de rastreo de genealogía que ha sido objeto de muchas críticas debido a su dependencia de las poblaciones de referencia. Se ha descrito cómo la tecnología contemporánea permite rastrear el linaje ancestral, pero solo a lo largo de las líneas de una línea materna y una línea paterna. Es decir, de 64 tatarabuelos en total, solo se identifica uno de cada padre, lo que implica que los otros 62 antepasados se ignoran en los esfuerzos de rastreo.[69] Además, las "poblaciones de referencia" utilizadas como marcadores para la pertenencia a un grupo particular se designan de forma arbitraria y contemporánea. El uso de poblaciones que actualmente residen en lugares determinados como referencias para ciertas razas y grupos étnicos no es confiable debido a los cambios demográficos que se han producido durante muchos siglos en esos lugares. Además, los marcadores informativos de ascendencia se comparten ampliamente entre toda la población humana, lo que se prueba es su frecuencia, no su mera ausencia/presencia. Por lo tanto, debe establecerse un umbral de frecuencia relativa. Los criterios para establecer dichos umbrales son un secreto comercial de las empresas que comercializan las pruebas. Por tanto, no se puede decir nada concluyente sobre si son apropiados. Los resultados de los AIM son extremadamente sensibles a dónde se establece esta barra.[70] Dado que muchos rasgos genéticos se encuentran muy similares entre muchas poblaciones diferentes, la tasa de frecuencia que se considera suficiente para formar parte de una población de referencia es muy importante. Esto también puede conducir a errores, dado que muchas poblaciones pueden compartir los mismos patrones, si no exactamente los mismos genes.
Esto sucede porque los AIM se basan en una suposición de '100% de pureza' de las poblaciones de referencia. Es decir, asumen que un patrón de rasgos sería idealmente condición necesaria y suficiente para asignar un individuo a una población ancestral de referencia. Raza, genética y medicinaExisten ciertas diferencias estadísticas entre grupos raciales en cuanto a la susceptibilidad a ciertas enfermedades.[71] Los genes cambian en respuesta a enfermedades locales; por ejemplo, las personas que son negativas tienden a tener una mayor resistencia a la malaria. El fenotipo negativo es muy frecuente en África central y la frecuencia disminuye con la distancia a África central, con frecuencias más altas en poblaciones globales con altos grados de inmigración africana reciente. Esto sugiere que el genotipo negativo evolucionó en África subsahariana y posteriormente fue seleccionado positivamente para la zona endémica de malaria. Varias condiciones genéticas prevalentes en áreas endémicas de malaria pueden proporcionar resistencia genética a la malaria, incluida la anemia de células falciformes, talasemias y glucosa-6-fosfato deshidrogenasa. La fibrosis quística es la enfermedad autosómica recesiva limitante de la vida más común entre las personas de ascendencia europea; se ha cuestionado una hipotética ventaja heterocigótica, que proporciona resistencia a enfermedades que antes eran comunes en Europa.[72] Los científicos Michael Yudell, Dorothy Roberts, Rob DeSalle y Sarah Tishkoff argumentaron que el uso de estas asociaciones en la práctica de la medicina ha llevado a los médicos a pasar por alto o identificar erróneamente la enfermedad.
La información sobre la población de origen de una persona puede ayudar en el diagnóstico y las respuestas adversas a los medicamentos pueden variar según el grupo.[42] Debido a la correlación entre la raza autoidentificada y los grupos genéticos, los tratamientos médicos influenciados por la genética tienen diferentes tasas de éxito entre los grupos raciales autodefinidos.[74] Por esta razón, algunos médicos consideran la raza del paciente al elegir el tratamiento más eficaz,[75] y algunos medicamentos se comercializan con instrucciones específicas de la raza.[76] Se ha argumentado que debido a la variación genética dentro de los grupos raciales, cuando
Sin embargo, la raza sigue siendo un factor al examinar grupos (como la investigación epidemiológica).[61] Algunos médicos y científicos, como el genetista Neil Risch, argumentan que el uso de la raza autoidentificada como un sustituto de la ascendencia es necesario para poder obtener una muestra suficientemente amplia de diferentes poblaciones ancestrales y, a su vez, para poder brindar una atención médica personalizada a las necesidades de los grupos minoritarios.[77] Uso en revistas científicasAlgunas revistas científicas han abordado errores metodológicos anteriores al exigir un escrutinio más riguroso de las variables de población. Desde 2000, Nature Genetics exige a sus autores que "expliquen por qué hacen uso de grupos étnicos o poblaciones particulares y cómo se logró la clasificación". Los editores de Nature Genetics dicen que "[ellos] esperan que esto genere conciencia e inspire diseños más rigurosos de estudios genéticos y epidemiológicos".[78] Interacciones gen-ambienteSe ha argumentado que la raza autoidentificada es de mayor uso en medicina, ya que se correlaciona fuertemente con exposomas relacionados con el riesgo que son potencialmente heredables cuando se incorporan al epigenoma. Resumen la evidencia del vínculo entre la discriminación racial y los resultados de salud debido a la peor calidad de los alimentos, el acceso a la atención médica, las condiciones de vivienda, la educación, el acceso a la información, la exposición a agentes infecciosos y sustancias tóxicas y la escasez de materiales. También citan evidencia de que este proceso puede funcionar positivamente; por ejemplo, la ventaja psicológica de percibirse a sí mismo en la cima de una jerarquía social está relacionada con una mejor salud. Sin embargo, advierten que los efectos de la discriminación no ofrecen una explicación completa de las tasas diferenciales de enfermedad y factores de riesgo entre grupos raciales, y el empleo de personas de raza autoidentificada tiene el potencial de reforzar las desigualdades raciales.[79] Objeciones al naturalismo racialEl naturalismo racial es la opinión de que las clasificaciones raciales se basan en patrones objetivos de similitudes y diferencias genéticas. Los defensores de este punto de vista lo han justificado utilizando la evidencia científica descrita anteriormente. Sin embargo, este punto de vista es controvertido y los filósofos de la raza le han presentado cuatro objeciones principales.[80] Las objeciones semánticas, como la objeción de la discreción, argumentan que las poblaciones humanas seleccionadas en la investigación genética de poblaciones no son razas y no corresponden a lo que significa "raza" en los Estados Unidos.
Por lo tanto, se podría argumentar que la investigación científica no se trata realmente de raza. Las dos siguientes objeciones son objeciones metafísicas que argumentan que incluso si las objeciones semánticas fallan, los resultados del agrupamiento genético humano no apoyan la realidad biológica de la raza. La "objeción muy importante" estipula que las razas en la definición estadounidense no son importantes para la biología, en el sentido de que las poblaciones continentales no forman subespecies biológicas. La 'objeción objetivamente real' establece que
Los naturalistas raciales han respondido a cada una de estas objeciones con argumentos en contra. También hay críticos metodológicos que rechazan el naturalismo racial debido a preocupaciones relacionadas con el diseño experimental, la ejecución o la interpretación de la investigación genética de poblaciones relevante.[83] Otra objeción semántica es la objeción de visibilidad que refuta la afirmación de que existen grupos raciales estadounidenses en las estructuras de la población humana. Filósofos como Joshua Glasgow y Naomi Zack creen que los grupos raciales estadounidenses no pueden definirse por rasgos visibles, como el color de la piel y los atributos físicos:
Una objeción diferente establece que los grupos raciales estadounidenses no son biológicamente reales porque no son objetivamente reales en el sentido de existir independientemente de algún estado mental de los seres humanos.[84][85] Se sostiene que una entidad puede ser tanto biológicamente real como socialmente construida y para capturar con precisión las entidades biológicas reales, también se deben considerar los factores sociales. Se ha argumentado que el conocimiento de la raza de una persona tiene un valor limitado, ya que las personas de la misma raza varían entre sí.[61] David J. Witherspoon y sus colegas han argumentado que cuando se asignan individuos a grupos de población, dos individuos elegidos al azar de diferentes poblaciones pueden parecerse más entre sí que un miembro elegido al azar de su propio grupo. Descubrieron que se tenían que usar muchos miles de marcadores genéticos para la respuesta a "¿Con qué frecuencia un par de individuos de una población es genéticamente más diferente que dos individuos elegidos de dos poblaciones diferentes?" ser "nunca". Esto asumió tres grupos de población, separados por grandes distancias geográficas (Europa, África y Asia Oriental). La población humana mundial es más compleja y estudiar una gran cantidad de grupos requeriría un mayor número de marcadores para la misma respuesta. Llegan a la conclusión de que "se debe tener precaución al utilizar la ascendencia geográfica o genética para hacer inferencias sobre fenotipos individuales".[86]
Esto es similar a la conclusión a la que llegó el antropólogo Norman Sauer en un artículo de 1992 sobre la capacidad de los antropólogos forenses para asignar "raza" a un esqueleto, basándose en las características craneofaciales y la morfología de las extremidades.
Críticas a los medicamentos basados en la razaTroy Duster señala que la genética a menudo no es el determinante predominante de la susceptibilidad a las enfermedades, aunque podrían correlacionarse con categorías específicas definidas socialmente. Esto se debe a que esta investigación a menudo carece de control sobre una multiplicidad de factores socioeconómicos. Cita datos recopilados que indican cómo las diferencias dietéticas desempeñan un papel importante en la explicación de las variaciones de la prevalencia de la diabetes entre las poblaciones. Duster elabora presentando el ejemplo de los pima de Arizona, una población que padece tasas desproporcionadamente altas de diabetes mellitus. La razón de esto, argumenta, no fue necesariamente el resultado de la prevalencia del gen FABP2, que está asociado con la resistencia a la insulina. Más bien, sostiene que los científicos a menudo descartan las implicaciones del estilo de vida en contextos sociohistóricos específicos. Por ejemplo, a finales del siglo XIX, la economía pima se basaba predominantemente en la agricultura. Sin embargo, a medida que la población estadounidense de origen europeo se instala en territorio tradicionalmente pima, los estilos de vida de los pima se occidentalizaron en gran medida. En tres décadas, la incidencia de diabetes aumentó en múltiples pliegues. La provisión gubernamental de alimentos gratuitos para aliviar la prevalencia de la pobreza en la población, que tenía un contenido relativamente alto de grasas, se señala como una explicación de este fenómeno.[89] Se ha argumentado en contra de la suposición de que "la raza autoidentificada es un buen indicador de una ascendencia genética específica"[79] sobre la base de que la raza autoidentificada es compleja: depende de una variedad de factores psicológicos, culturales y sociales, y, por lo tanto, "no es un proxy sólido de la ascendencia genética".[90] Además, explican que la raza autoidentificada de un individuo se compone de otros factores colectivamente arbitrarios: opiniones personales sobre qué es la raza y hasta qué punto debe tenerse en cuenta en la vida cotidiana. Además, los individuos que comparten una ascendencia genética pueden diferir en su autoidentificación racial en contextos históricos o socioeconómicos. A partir de esto, se concluye que la precisión en la predicción de la ascendencia genética sobre la base de la autoidentificación es baja, específicamente en poblaciones mezcladas racialmente nacidas de historias ancestrales complejas. Véase tambiénReferencias
Otras lecturas
|
||||||