|
Small interfering RNA Small interfering RNA, abgekürzt siRNA, (englisch für kleine eingreifende RNA) sind kurze, einzel- oder doppelsträngige Ribonukleinsäure-Moleküle von 20 bis 25 Basenpaaren Länge. Sie codieren keine Proteine, sondern verbinden sich mit komplementären einzelsträngigen Ribonukleinsäure-Molekülen, wodurch sie deren normale Funktion unterbinden. Dieser Vorgang wird RNA-Interferenz genannt und spielt eine wichtige Rolle in den Zellen von Lebewesen mit einem Zellkern, den Eukaryoten. So unterdrückt RNA-Interferenz die Expression von Genen und stoppt die Replikation von Viren. StrukturEine siRNA ist ein etwa 19 bis 23 Basenpaare langer RNA-Doppelstrang, dessen Einzelstränge jeweils am 3'-Ende um zwei Nukleotide überhängen. Das 5'-Ende jedes Stranges ist phosphoryliert, während die 3'-Enden freie Hydroxygruppen tragen. BiosyntheseDie siRNA wird durch eine Spaltung eines großen doppelsträngigen RNA-Moleküls gebildet. Diese Vorläufer-RNA kann mehrere Hunderte bis Tausende Basenpaare groß sein und fällt beispielsweise bei der Vervielfältigung viraler RNA an. An der Spaltung ist insbesondere das Enzym Dicer, eine sogenannte RNase vom Typ III, beteiligt. Funktion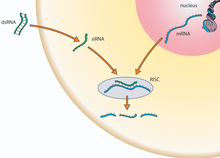 Die siRNA wird in vielen Zellen als eine Folge der Infektion mit einem RNA-Virus gebildet und spielt insbesondere bei Pflanzen bei der Verteidigung gegen fremde RNA eine wichtige Rolle. Sie fällt als ein Spaltprodukt bei der Vervielfältigung (Replikation) der Virus-RNA an und dient zugleich der Zelle zur Erkennung und Zerstörung dieser fremden RNA. Ähnliche Mechanismen konnten auch bei Pilzen, Fadenwürmern und Insekten gefunden werden. Zahlreiche Viren versuchen ihrerseits über eine Hemmung der an der RNA-Interferenz beteiligten Proteine diesem Abwehrmechanismus zu entgehen.[1] Auch bei der Genregulation über das Posttranskriptionelle Gen-Silencing kann die siRNA eine zentrale Rolle spielen, indem sie die Selektivität bestimmt. Zusammen mit speziellen Proteinkomponenten bildet sie den RNA-induced silencing complex (RISC). Nur ein Strang der siRNA, der in diesem Zusammenhang ‚Leitstrang‘ genannt wird, wird dabei übernommen. Einzelsträngige RNA, insbesondere also Messenger-RNA, die die zum Leitstrang der siRNA komplementäre Nukleotidsequenz enthält, wird vom RISC zerstört oder die Translation zum Protein auf andere Weise verhindert. Da nur solche mRNA zum Protein übersetzt wird, die dem RISC entkommt, ist die Aktivität des betroffenen Gens reduziert. Erstes Opfer dürfte im Allgemeinen der nicht übernommene Teilstrang der siRNA sein. AnwendungsiRNA wird insbesondere in der Grundlagenforschung zur Aufklärung der noch unbekannten Funktion eines zu untersuchenden bekannten Gens und dessen codierten Proteins mit Hilfe der RNA-Interferenz genutzt. Durch die gezielte Abschaltung des Gens mit Hilfe von siRNA kann die Funktion des von ihm codierten Proteins abgeleitet werden. Auch für die umgekehrte Fragestellung, die Suche nach den für eine bekannte Funktion oder ein bestimmtes Merkmal verantwortlichen Genen oder Proteinen, können siRNA-Bibliotheken genutzt werden. Auch die therapeutische Anwendung von siRNA in der Medizin ist Gegenstand der Forschung. Vier siRNA-basierte Wirkstoffe wurden seit 2018 bereits zugelassen. Weitere siRNA-basierte potenzielle Arzneistoffe befinden sich in Erprobung.
Fitusiran ist ein noch experimentelles, in Phase III befindliches, subkutan verabreichtes Small-Interference-RNA-Therapeutikum, das für die prophylaktische Behandlung von Menschen mit Hämophilie A oder B mit oder ohne Inhibitoren entwickelt wird.[2] Ein weiterer Arzneistoffkandidat ist Olpasiran der Firma Amgen, der in einer Phase II Studie Lipoprotein (a), abgekürzt LP(a) dosisabhängig senkte. LP(a) erhöht wie LDL-Cholesterin in hohen Konzentrationen das Risiko für Arterienverkalkung.[3][4][5] Auch einige bei Affen wirksame Medikamente gegen Filoviren, wie Marburg- und Ebola-Viren, basieren auf siRNA.[6] Involvierte FirmenNach dem Scheitern von Bevasiranib[7] stellten mehrere große pharmazeutische Unternehmen, darunter auch Merck & Co., Novartis und Roche, ihre auf siRNA basierenden Entwicklungsprogramme ein[8], während das Unternehmen Alnylam Pharmaceuticals erfolgreich die o.a. vier RNAi-Therapeutika zur Zulassung brachte. Sanofi und Alnylam Pharmaceuticals vereinbarten in 2012/2014 eine Entwicklungszusammenarbeit für die Medikamente von Alnylam. In 2018 gab Sanofi die Entwicklungs- und Vermarktungsrechte für die RNAi-Therapeutika zur Behandlung von ATTR-Amyloidose an Alnylam zurück. Sanofi ist weiterhin für die Entwicklung von Fitusiran verantwortlich.[9] Im Juli 2022 kündigten Sanofi und das in Shanghai ansässige Biotechnologie Unternehmen Rona Therapeutics eine exklusive Lizenzvereinbarung an, das siRNA-Therapieportfolio seine Technologieplattform von Sanofi an Rona zu übertragen. Rona Therapeutics ist ein RNA-Therapeutik-Plattformunternehmen, das sich der Entdeckung und Entwicklung modularer und programmierbarer RNA-Medikamente widmet.[10] Literatur
Einzelnachweise
Commons: Small interfering RNA – Sammlung von Bildern, Videos und Audiodateien
|
