|
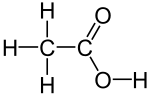
Simplified Molecular Input Line Entry SystemSimplified Molecular Input Line Entry System (SMILES, zuweilen auch für Simplified Molecular Line Entry Specification) ist ein chemischer Strukturcode, bei dem die Strukturen beliebiger Moleküle stark vereinfacht als (ASCII-)Zeichenkette wiedergegeben werden. Mehrere Molekül-Editoren können SMILES-Strings importieren und so zweidimensionale und dreidimensionale Modelle erzeugen. Die ursprüngliche SMILES-Spezifikation wurde von Arthur Weininger und David Weininger in den späten 1980ern entwickelt.[1][2][3] Insbesondere die Daylight Chemical Information Systems Inc. betrieb in den folgenden Jahren die Weiterentwicklung und Modifikation der Spezifikation. Im Jahr 2007 wurde schließlich ein offener Standard namens OpenSMILES von Blue Obelisk, einer chemisch orientierten Open-Source-Community, entwickelt. Da die SMILES-Sprache durch die Firma Daylight kontrolliert wird und einige Probleme mit der Stereochemie und Tautomerie hat, hat die IUPAC eine eigene lineare Molekülrepresentation, InChI, entwickelt, die frei verfügbar ist. Beispiele
KonventionenAtomeEin Chemisches Element wird durch sein Elementsymbol, das in eckigen Klammern eingefasst ist, dargestellt (z. B. Ionen, also elektrisch geladene Atome, werden in der SMILES-Notation durch Angabe der Ladung in den eckigen Klammern beschrieben (z. B. Direkt an das Atom gebundener Wasserstoff kann ebenfalls in der Klammernotation mit angegeben werden, dazu wird hinter dem Elementsymbol ein Um die Notation zu vereinfachen, können die eckigen Klammern bei Elementen der sogenannten „organischen Teilmenge“ weggelassen werden.
Wenn die Klammern weggelassen werden, dann werden die freien Valenzen des Atoms auf die niedrigste Standardvalenz nach der abgebildeten Tabelle mit Wasserstoffatomen aufgefüllt.
So reicht zum Beispiel für die Eingabe von Wasser ein
BindungenUm anzuzeigen, dass zwei Atome durch eine chemische Bindung verbunden sind, wird eines der folgenden Symbole zwischen die Atome gesetzt.
* Nur OpenSMILES Um die Schreibweise noch weiter zu vereinfachen, dürfen die Symbole für Einfachbindungen und aromatische Bindungen weggelassen werden.[5] VerzweigungenAtome mit drei oder mehr Bindungen sind Ausgangspunkt von Verzweigungen. Dabei wird nach dem entsprechenden Atom erst die Seitenkette in runden Klammern gesetzt, bevor die weiteren Bindungen folgen. Die Klammerebenen und damit die Verzweigungen können beliebig tief verschachtelt werden.[6] Beispiele:
Getrennte StrukturenBei Strukturen, die nicht zusammenhängen, wie z. B. Ionenbindungen wird ein Punkt (.) zwischen die getrennten Moleküle gesetzt.[7] Cyclische StrukturenEines der größten Probleme einer solchen Sprache ist es, cyclische Strukturen darzustellen. Bei SMILES geschieht dies dadurch, dass man nach einem Atom, das mit einem anderen Atom verbunden werden soll, welches weiter hinten steht, einen Index schreibt; dies macht man auch bei dem anderen Atom und die beiden sind verbunden.[8] Bei aromatischen Ringen werden die ringbildenden Atome kleingeschrieben. Beispiele:
ReaktionenReaktionen werden in SMILES mithilfe von 2 schließenden spitzen Klammern (>>) dargestellt. Wenn bei einer Reaktion ein weiterer Stoff einfließt, so schreibt man ihn zwischen die Spitzen Klammern. ErweiterungSMARTS ist eine Erweiterung von SMILES, die das Suchen nach molekularen Substrukturen ermöglicht. Hierzu wurde SMILES, um Wildcards oder spezifische Bindungen (z. B. aromatische) anzugeben, modifiziert. Es gilt, dass jeder gültige SMILES-Ausdruck ebenfalls als SMARTS verwendet werden darf. Diese Regel gilt andersherum nicht. SMARTS werden vor allem für Suchanwendungen bei chemischen Datenbanken verwendet. Siehe auch
Weblinks
Einzelnachweise
|