|
Fluoreszenzmarkierung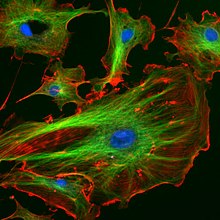 Die Fluoreszenzmarkierung ist eine Methode der Biochemie zur Markierung von Biomolekülen mit Fluorophoren. Prinzip Biomoleküle werden gelegentlich zur erleichterten Verfolgung mit fluoreszenten Molekülen versehen.[2] Durch die Kupplung von Fluorophoren an Proteine oder Kohlenhydrate auf der Zelloberfläche können auch Zellen direkt markiert werden oder mit fluoreszenten Immunkonjugaten indirekt nachgewiesen werden,[3] z. B. per Durchflusszytometrie, Fluoreszenzmikroskopie oder Fluoreszenztomographie. Bei einer Fluoreszenzmarkierung kann das nachzuweisende Molekül direkt mit einem Fluorophor versehen werden oder indirekt durch selektiv bindende fluoreszente Moleküle nachgewiesen werden. Methoden zur Fluoreszenzmarkierung sind:
Proteine Bei Proteinen werden in vivo unter anderem das grün fluoreszierende Protein (GFP) als rekombinantes Fusionsprotein oder als Reportergen verwendet.[4] Inteine können zur C-terminalen Markierung von Proteinen eingesetzt werden. Als fluoreszentes Protein-Tag kann auch das Flash-Tag verwendet werden. Mit Redox-abhängigen Fluoreszenten Proteinen wie roGFP, rxYFP und HyPer kann die Fluoreszenz sauerstoffabhängig verändert werden. Mit spannungsabhängigen Proteinen wie VSFP kann die Fluoreszenz durch Anlegen einer elektrischen Spannung verändert werden.   Proteine können auch teilsynthetisch (bioorthogonal) markiert werden, in dem reaktive Aminosäurederivate in vivo eingebaut werden, die im Anschluss in vitro mit einem Fluorophor gekuppelt werden. Weiterhin werden Proteine mit verschiedenen chemischen Kupplungsreaktionen in vitro mit unterschiedlichen Fluorophoren versehen. Beispielsweise können nukleophile Gruppen (am häufigsten kommen Aminogruppen in Proteinen vor) durch Reaktionen mit Isothiocyanate (z. B. FITC, TRITC), Succinimidylestern (z. B. CFSE) mit unterschiedlichen Fluoreszenzfarbstoffen wie z. B. Fluorescein, Rhodamin und deren Derivate markiert werden. 1-Fluor-2,4-dinitrobenzol und Dansylchlorid reagieren mit Aminogruppen. Cysteine können mit IAEDANS oder Ellmans Reagenz markiert werden. Proteine können auch indirekt durch eine Immunmarkierung mit fluoreszenten Immunkonjugaten nachgewiesen werden, z. B. bei der Fluoreszenzmikroskopie, dem Western Blot und dem Immunoassay. Im Zuge einer Peptidsynthese können Peptide durch Verwendung von fluoreszenten Aminosäurederivaten markiert werden. Durch einen Förster-Resonanzenergietransfer (FRET), Biolumineszenz-Resonanzenergietransfer (BRET), Fluoreszenzkorrelationsspektroskopie (FCS) oder eine Bimolekulare Fluoreszenzkomplementation (BiFC) kann mit Fluorophoren die Nachbarschaft zweier Proteine nachgewiesen werden. NukleinsäurenDNA kann bioorthogonal markiert werden. Dazu kann DNA in vivo oder in vitro mit Nukleosidanaloga markiert werden, welche z. B. anschließend per Staudinger-Reaktion mit einem Fluorophor gekuppelt werden können.[5] DNA kann chemisch mit Fluorophoren versehen werden.[6] Oligonukleotide können durch eine Phosphoramidit-Synthese mit Fluorophoren markiert werden, die z. B. in der QPCR, der DNA-Sequenzierung und der In-situ-Hybridisierung verwendet werden. Daneben kann DNA enzymatisch im Zuge einer Polymerasekettenreaktion mit fluoreszenten Nukleotiden erzeugt oder mit einer Ligase oder einer terminalen Desoxynukleotidyltransferase markiert werden.[7][8][9] DNA kann indirekt durch eine Biotinylierung und fluoreszentem Avidin nachgewiesen werden. Als Fluorophore werden für Kupplungen unter anderem Fluorescein, fluoreszente Lanthanoide,[10][11] Gold-Nanopartikel, Kohlenstoffnanoröhren oder Quantenpunkte eingesetzt.[6] Literatur
Einzelnachweise
|