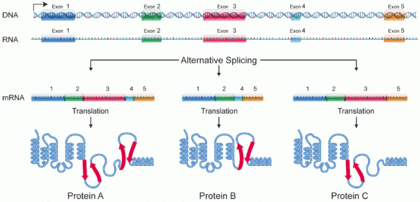
蛋白质A、B和C是由同一基因通过选择性剪接 编码的异构体。 蛋白异构体 (Protein isoform )或蛋白异形体 [ 1] 蛋白变异体 (Protein variant[ 2] 基因 或基因家族 的一组高度相似的蛋白质 ,常具有相同或相似的生物学功能,是遗传差异的结果[ 3]
虽然大多数蛋白异构体功能相同或近似,但某些异构体具有独特的功能。一组蛋白质异构体可以通过单个基因的选择性剪接 、可变启动子 的使用或其他转录后修饰 形成,但通常不包括翻译后修饰 。(参阅蛋白质变体 RNA剪接 机制,mRNA 能够选择基因的不同蛋白质编码片段(外显子 ),甚至从RNA 中选择外显子的不同部分,从而形成不同的mRNA序列。每个独特的序列都会产生特定形式的蛋白质。
随着蛋白质异构体的发现,由人类基因组计划 揭示的蛋白质编码区 数量较少,但生物体中蛋白质多样性却较丰富这一问题得到了解释:由同一基因编码的不同蛋白质可以增加蛋白质组 的多样性。RNA水平的异构体可以很轻松地通过cDNA 转录研究来表征。许多人类基因具有已确认的选择性剪接 异构体。据估计,人类中可以识别出约10万个表达序列标签 (EST)。[ 2] 结构域 或较短环状结构的缺失,通常位于蛋白质表面。[ 4]
单个基因能够产生多种在结构和组成上均不同的蛋白质。[ 5] [ 6] 选择性剪接 所调节。然而,这一过程对人类蛋白质组 多样性的影响程度尚不清楚,因为mRNA转录异构体的丰度并不一定与蛋白质异构体的丰度相关。[ 7] AlphaFold 已经预测了人类蛋白质组中大多数异构体的结构,并在isoform.io (页面存档备份 ,存于互联网档案馆 )上公开发布。[ 8] [ 5] [ 6] 亚基 且每个亚基都具有多个异构体时,确定特异性就变得更加复杂。
例如,单磷酸腺苷激活的蛋白激酶 [ 9]
在人类骨骼肌 中,主要形式是α2β2γ1。[ 9] [ 9]
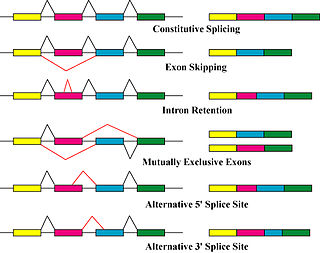
RNA剪接 的不同机制产生蛋白质异构体的主要机制是选择性剪接 和可变启动子 的使用,但由基因变化而引起的改变(如突变 和多态性 )有时也被认为是不同的异构体。[ 10]
选择性剪接是产生mRNA转录异构体的主要转录后修饰 过程,也是可能导致蛋白质多样性的主要分子机制。[ 6] 剪接体 是细胞核内负责RNA切割和连接 的大型核糖核蛋白 分子机器,它会移除非编码蛋白质的片段(内含子)。[ 11]
由于剪接是发生在转录 和翻译 之间的过程,其主要影响主要通过基因组学 技术进行研究。例如,微阵列分析 和RNA测序 已被用于识别选择性剪接转录本并测量其丰度。[ 10] 凝胶电泳 和质谱 的蛋白质组学 实验已证明转录本和蛋白质数量之间的相关性通常很低,并且往往仅一种蛋白质异构体占主导地位。[ 12] [ 13] [ 12]
选择性剪接通常描述一种严格调控的过程,其中剪接机制有意生成选择性转录本。然而,此类转录本也可能通过剪接错误产生,该过程称为“剪切噪声”,并且也可能被翻译成蛋白质异构体。尽管约95%的多外显子基因被认为是选择性剪接的,但一项关于剪切噪声的研究发现,大多数不同的低丰度转录本都是噪声,并预测细胞中大多数选择性转录本和蛋白质异构体并没有功能上的相关性。[ 14]
其他转录和转录后调控步骤也可产生不同的蛋白质异构体。[ 15] RNA聚合酶 、转录因子 和其他酶 )在不同的启动子 (基因附近作为初始结合位点 的DNA区域)开始转录时,会发生可变启动子的使用,从而产生略有不同的转录本和蛋白质异构体。
通常,一种蛋白质异构体被标记为规范序列是基于其普遍性和与其他物种的直系同源 (或功能性同功)的相似性等标准。[ 16] 反式剪接 ),并且可能与规范序列共享少部分甚至不共享外显子。此外,它们可以具有不同的生物学效应,例如在极端情况下,一种异构体的功能可能促进细胞存活,而另一种异构体则可能促进细胞死亡;它们也可能具有相似的基本功能,只是在亚细胞定位 上有所不同。[ 17] [ 18] 体外 细胞上进行的,因此尚不清楚表达的人类蛋白质组中的异构体是否具有这些特征。此外,由于每个异构体的功能通常必须单独确定,因此大多数已识别和预测的异构体的功能仍然是未知的。
糖型 是指仅在附着的聚糖 数量或类型上有所不同的蛋白质异构体。糖蛋白 通常由多种不同的糖型组成,这些糖型在附着的糖 或寡糖 上有所差异。这些变化可能是由于糖基化 过程中的生物合成 差异,或由于糖苷酶 或糖基转移酶 的作用所致。糖型可以通过对分离的糖型进行详细的化学分析来检测,但更便捷的检测方法是通过与凝集素 的差异反应,如凝集素亲和色谱法 和凝集素亲和电泳法 。由糖型组成的糖蛋白的典型例子是血液蛋白 ,如血清类粘蛋白 、抗胰蛋白酶 和结合珠蛋白 。在神经元黏附分子 (NCAM)中涉及聚唾液酸 (PSA)的糖型变异是一种不寻常的情况。
G-肌动蛋白 :尽管其性质保守,但它具有不同数量的异构体(哺乳动物 中至少有六种)。肌酸激酶 :其在血液中的存在可用于帮助诊断心肌梗死 ,以三种异构体的形式存在,。透明质酸合酶 :负责透明质酸 生成的酶,在哺乳动物细胞中有三种异构体。尿苷雙磷酸葡萄糖醛酸轉移酶 :这个酶超家族负责许多药物、环境污染物和有毒内源性化合物的解毒途径,已知有16种异构体编码在人类基因组中。[ 19] G6PDA:在任何组织细胞中,其活性异构体的正常比例与G6PDG相同,为1:1。这正是增生中的正常异构体比例。在肿瘤 形成过程中只发现其中一种异构体。
单胺氧化酶 :这个酶家族是一类催化单胺 氧化的酶,存在两种异构体,分别是MAO-A和MAO-B。[ 20]
^ 蛋白质异形体 . 术语在线 . 全国科学技术名词审定委员会 . (简体中文) ^ 2.0 2.1 Brett D, Pospisil H, Valcárcel J, Reich J, Bork P. Alternative splicing and genome complexity. Nature Genetics. January 2002, 30 (1): 29–30. PMID 11743582 S2CID 2724843 doi:10.1038/ng803 ^ Schlüter H, Apweiler R, Holzhütter HG, Jungblut PR. Finding one's way in proteomics: a protein species nomenclature . Chemistry Central Journal. September 2009, 3 : 11. PMC 2758878 PMID 19740416 doi:10.1186/1752-153X-3-11 ^ Kozlowski, L.; Orlowski, J.; Bujnicki, J. M. Structure Prediction for Alternatively Spliced Proteins. Alternative pre-mRNA Splicing. 2012: 582. ISBN 9783527636778doi:10.1002/9783527636778.ch54 ^ 5.0 5.1 Andreadis A, Gallego ME, Nadal-Ginard B. Generation of protein isoform diversity by alternative splicing: mechanistic and biological implications. Annual Review of Cell Biology. 1987-01-01, 3 (1): 207–42. PMID 2891362 doi:10.1146/annurev.cb.03.110187.001231 ^ 6.0 6.1 6.2 Breitbart RE, Andreadis A, Nadal-Ginard B. Alternative splicing: a ubiquitous mechanism for the generation of multiple protein isoforms from single genes. Annual Review of Biochemistry. 1987-01-01, 56 (1): 467–95. PMID 3304142 doi:10.1146/annurev.bi.56.070187.002343 ^ Liu Y, Beyer A, Aebersold R. On the Dependency of Cellular Protein Levels on mRNA Abundance. Cell. April 2016, 165 (3): 535–50. PMID 27104977 doi:10.1016/j.cell.2016.03.014 hdl:20.500.11850/116226 ^ Sommer, Markus J.; Cha, Sooyoung; Varabyou, Ales; Rincon, Natalia; Park, Sukhwan; Minkin, Ilia; Pertea, Mihaela; Steinegger, Martin; Salzberg, Steven L. Structure-guided isoform identification for the human transcriptome . eLife. 2022-12-15, 11 : e82556. PMC 9812405 PMID 36519529 doi:10.7554/eLife.82556 (英语) . ^ 9.0 9.1 9.2 Dasgupta B, Chhipa RR. Evolving Lessons on the Complex Role of AMPK in Normal Physiology and Cancer . Trends in Pharmacological Sciences. March 2016, 37 (3): 192–206. PMC 4764394 PMID 26711141 doi:10.1016/j.tips.2015.11.007 (English) . ^ 10.0 10.1 Kornblihtt AR, Schor IE, Alló M, Dujardin G, Petrillo E, Muñoz MJ. Alternative splicing: a pivotal step between eukaryotic transcription and translation. Nature Reviews Molecular Cell Biology. March 2013, 14 (3): 153–65. PMID 23385723 S2CID 54560052 doi:10.1038/nrm3525 hdl:11336/21049 (英语) . ^ Lee Y, Rio DC. Mechanisms and Regulation of Alternative Pre-mRNA Splicing . Annual Review of Biochemistry. 2015-01-01, 84 (1): 291–323. PMC 4526142 PMID 25784052 doi:10.1146/annurev-biochem-060614-034316 ^ 12.0 12.1 Tress ML, Abascal F, Valencia A. Alternative Splicing May Not Be the Key to Proteome Complexity . Trends in Biochemical Sciences. February 2017, 42 (2): 98–110. PMC 6526280 PMID 27712956 doi:10.1016/j.tibs.2016.08.008 ^ Battle A, Khan Z, Wang SH, Mitrano A, Ford MJ, Pritchard JK, Gilad Y. Genomic variation. Impact of regulatory variation from RNA to protein . Science. February 2015, 347 (6222): 664–7. PMC 4507520 PMID 25657249 doi:10.1126/science.1260793 ^ Pickrell JK, Pai AA, Gilad Y, Pritchard JK. Noisy splicing drives mRNA isoform diversity in human cells . PLOS Genetics. December 2010, 6 (12): e1001236. PMC 3000347 PMID 21151575 doi:10.1371/journal.pgen.1001236 ^ Smith LM, Kelleher NL. Proteoform: a single term describing protein complexity . Nature Methods. March 2013, 10 (3): 186–7. PMC 4114032 PMID 23443629 doi:10.1038/nmeth.2369 (英语) . ^ Li HD, Menon R, Omenn GS, Guan Y. Revisiting the identification of canonical splice isoforms through integration of functional genomics and proteomics evidence (PDF) . Proteomics. December 2014, 14 (23–24): 2709–18. PMC 4372202 PMID 25265570 doi:10.1002/pmic.201400170 ^ Sundvall M, Veikkolainen V, Kurppa K, Salah Z, Tvorogov D, van Zoelen EJ, Aqeilan R, Elenius K. Cell death or survival promoted by alternative isoforms of ErbB4 . Molecular Biology of the Cell. December 2010, 21 (23): 4275–86. PMC 2993754 PMID 20943952 doi:10.1091/mbc.E10-04-0332 ^ Yang X, Coulombe-Huntington J, Kang S, Sheynkman GM, Hao T, Richardson A, et al. Widespread Expansion of Protein Interaction Capabilities by Alternative Splicing . Cell. February 2016, 164 (4): 805–17. PMC 4882190 PMID 26871637 doi:10.1016/j.cell.2016.01.029 ^ Barre L, Fournel-Gigleux S, Finel M, Netter P, Magdalou J, Ouzzine M. Substrate specificity of the human UDP-glucuronosyltransferase UGT2B4 and UGT2B7. Identification of a critical aromatic amino acid residue at position 33. The FEBS Journal. March 2007, 274 (5): 1256–64. PMID 17263731 doi:10.1111/j.1742-4658.2007.05670.x ^ Pathoma, Fundamentals of Pathology