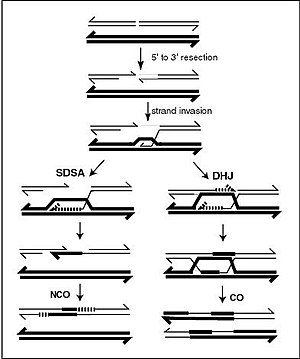
减数分裂重组模型,由双链断裂或缺口开始,然后与同源染色体配对、链侵入,开始重组修复过程。间隙的修复可能导致侧翼区域的交叉(CO)或非交叉(NCO)。CO的重组被认为是通过双霍利迪结(DHJ)模型发生的,如上图右边所示。NCO重组被认为主要是通过合成依赖链退火(SDSA)模型发生的,如上图左所示。大多数重组事件似乎是SDSA类型。 遺傳學 中,基因重組 (英語:genetic recombination/reshuffling )亦稱遗传重組 ,是指DNA 片段斷裂並且轉移位置的过程,会导致基因间或基因内新的连锁关系形成。对于真核生物 ,减数分裂 過程中的基因重組能夠形成一套新的遺傳信息,並從親本 遺傳給子代。多数基因重组是自然发生的,可以分为两种类型:(1)染色体间重组,通过位点在不同染色体上的等位基因的自由组合发生(减数分裂I中同源染色體 的非姐妹染色单体上的基因自由组合);(2)染色体内重组,通过交换发生。[ 1]
在真核生物 减数分裂过程中,遗传重组时同源染色體会配对。随后可能会发生染色体间的遗传信息传递。信息传递可以不需要物理交换(遗传物质的一部分从一条染色体复制到另一条染色体,而来源的染色体并未改变,见图中SDSA路径),也可以通过DNA链断裂并重新连接,形成新的DNA分子(见图中DHJ路径)。
重组也可能在真核生物有絲分裂 时发生,它通常涉及染色体复制后形成的两个姐妹染色体。在这种情况下,由于姐妹染色体通常是相同的,所以不会产生新的等位基因 组合。在减数分裂和有丝分裂中,DNA的相似分子(同源序列 )之间发生重组。减数分裂过程中,非姐妹同源染色体相互配对,使非姐妹同源染色体之间发生典型的重组。在减数分裂和有丝分裂细胞中,同源染色体之间的重组是DNA修復 中常见的机制。无性生殖 的细菌 和古菌 也会发生基因重组和重组DNA修復。
對原核生物 (例如細菌 )來說,個體之間可以通過交接,或是經由病毒 (例如噬菌體 )的傳送,來交換彼此的基因,並且利用基因重組,將這些基因組合到本身原有的遺傳物質中。
對於較複雜的生物來說,重組通常是因為同源染色體配對時發生互換 ,使得同源染色體 上的基因在遺傳到子代時,經常有不完全的連鎖。由於重組現象的存在,科學家可以利用重組率來定出基因之間的相對位置,描繪出基因圖譜 。
使同源序列相同的基因轉換 过程也属于基因重组。
重组可以在实验室(体外)环境中人工诱导发生,产生重組DNA ,用于疫苗 开发等目的。
在減數分裂 期間,聯會 (同源染色體的配對)通常在基因重組之前發生。在一般減數分裂的步驟,先是聯會 、再進行基因重組及分離(gene segregation)[ 2] [ 3] [ 4]
基因重组由多种酶 催化 进行。重组酶 大腸桿菌 内发现的主要重组酶RecA double strand break(s) ,DSB(s) )。在酵母 和其他真核生物中,修复DSB需要两种重组酶。有絲分裂 和减数分裂 重组过程需要RAD51 ,而DNA修复蛋白DMC1 同源 的是RadA。
托马斯·亨特·摩尔根 繪製的染色體互換示意圖真核生物 中,减数分裂 期间的重组是通过染色體互換 (或称交叉互换)进行的。交叉互换过程使后代拥有与亲本不同的基因组合,偶尔还会产生新的嵌合 等位基因 。基因的重组会增加遺傳變異 。它还允许有性繁殖的生物体避免穆勒棘轮效应 无性生殖 种群的情况,随着时间的推移,其基因組 中的有害突变往往会积累得比有益突变或复原突变更多。
染色体互换是指遗传自父母的成对染色体 之间的重组,通常发生在减数分裂期间。在前期I期(粗线期),四条染色单体 彼此紧密排列。而在这种结构中,两条染色单体上的同源 位点可以配对,并可以交换遗传信息。[ 5]
由于染色体的每个位置上都有很小的概率发生重组,两个位置之间重组的频率取决于它们之间的距离(遗传连锁 )。因此,对于同一染色体上相距足够远的基因,交叉互换的次数足以打破等位基因 之间的相关性。
对遗传学家来说,追踪由交叉互换产生的基因的移动已经被证明是非常有用的。因为两个相距较近的基因比相距较远的基因更难分离,所以如果知道交叉互换的频率,遗传学家就可以推断出一条染色体上两个基因之间的大致距离。遗传学家也可以使用这种方法来推断某些基因的存在。若两个基因在重组过程中常保持在一起,则称二者之间存在遗传连锁 。一个连锁对中的一个基因有时可以作为标记来推断另一个基因的存在。这通常用于检测致病基因的存在。[ 6]
观察得到的两个位点 之间的重组频率称为交换值 遗传标记 )之间发生染色體互換 的频率,取决于观察的两个遗传位点之间相隔的距离。对于任何一组固定的遗传和环境条件,连锁结构 (染色体 )的特定区域的重组频率往往是固定的,而用于制作遗传图谱 的交换值亦然。[ 7] [ 8]
在基因轉換 中,遗传物质的一部分从一条染色体复制到另一条染色体,而來源染色體不變。减数分裂过程中,基因转换发生的频率很高。它是一个DNA序列从一个DNA螺旋(不变)复制到另一个DNA螺旋,后者序列被改变的过程。真菌杂交[ 9]
重组可以发生在不同源 的DNA序列之间,其後果是染色體易位 ,有时会导致癌症。
免疫系统 的B细胞 进行的基因重组,称为免疫球蛋白类型转换 。这是一种将抗体 从一类变成另一类的生物机制,例如,从一种叫做IgM的同种型 变成IgG同种型。
基因工程 中,重组也可以指人为地将完全不相关的DNA片段(往往来自不同生物体)重新组合,形成重組DNA 。其中基因靶向 是的对基因重组的一种常见应用,可用来增添、删除或改变一个生物体的基因。该技术在生物医学研究 蛋白质工程
多种外源(如紫外线 、X射线 、化学交联 剂)引起的DNA损伤可通过同源重组修复(homologous recombinational repair ,HRR)得到恢复。[ 10] [ 11] 活性氧 )产生的DNA损伤,也会得到同源重组修复。对人类而言,:减数分裂时同源重组修复所需的基因产物缺乏可能导致不育 。[ 12]
Nowak和Ohtsuki[ 13] 生命起源 也是生物演化 的起源。他们指出,地球上所有已知的生命都是以生物聚合物 为基础的,并提出生命起源的任何理论都必然涉及作为信息载体和催化剂的生物聚合物。Lehman[ 14] [ 15]
^ Daly, M J; Minton, K W. Interchromosomal recombination in the extremely radioresistant bacterium Deinococcus radiodurans. . Journal of Bacteriology. October 1995, 177 (19): 5495–5505. ISSN 0021-9193 PMC 177357 PMID 7559335 doi:10.1128/jb.177.19.5495-5505.1995 ^ Hawley RS, Arbel T.,"Yeast genetics and the fall of the classical view of meiosis " (页面存档备份 ,存于互联网档案馆 ),PubMed - NCBI,1993 Feb 12;72(3):301-3.. PMID 8431941 DOI: 10.1016/0092-8674(93)90108-3
^ Peter B. Moens,"Molecular perspectives of chromosome pairing at meiosis " (页面存档备份 ,存于互联网档案馆 ),BioEssays - Wiley Online Library,Volume16,Issue2,February 1994,Pages 101-106.
^ 趙紹惠[1] 和 大衛摩亞[2] (页面存档备份 ,存于互联网档案馆 )(Siu-Wai Chiu & David Moore),"解讀真菌的形態發育--第六章(Deciphering Fungal Morphogenesis--Chapter 6)"[3] (页面存档备份 ,存于互联网档案馆 ),香港中文大學 生命科學學院,2003.
^ Alberts B. Molecular Biology of the Cell, Fourth Edition. New York: Garland Science. 2002. ISBN 978-0-8153-3218-3 ^ Access Excellence . Crossing-over: Genetic Recombination. The National Health Museum Resource Center. [February 23, 2011] . (原始内容存档 于2013-07-31). ^ Rieger R, Michaelis A, Green MM. Glossary of genetics and cytogenetics: Classical and molecular ISBN 978-3-540-07668-1 ^ King RC, Stransfield WD. Dictionary of Genetics.. New York, Oxford: Oxford University Press. 1998. ISBN 0-19-509442-5 ^ Stacey KA. Recombination. Kendrew J, Lawrence E (编). The Encyclopedia of Molecular Biology . Oxford: Blackwell Science. 1994: 945 –950. ^ Baker BS, Boyd JB, Carpenter AT, Green MM, Nguyen TD, Ripoll P, Smith PD. Genetic controls of meiotic recombination and somatic DNA metabolism in Drosophila melanogaster . Proceedings of the National Academy of Sciences of the United States of America. November 1976, 73 (11): 4140–4. Bibcode:1976PNAS...73.4140B PMC 431359 PMID 825857 doi:10.1073/pnas.73.11.4140 ^ Boyd JB. DNA repair in Drosophila . Hanawalt PC, Friedberg EC, Fox CF (编). DNA Repair Mechanisms. New York: Academic Press. 1978: 449–452. ^ Galetzka D, Weis E, Kohlschmidt N, Bitz O, Stein R, Haaf T. Expression of somatic DNA repair genes in human testes. Journal of Cellular Biochemistry. April 2007, 100 (5): 1232–9. PMID 17177185 S2CID 23743474 doi:10.1002/jcb.21113 ^ Nowak MA, Ohtsuki H. Prevolutionary dynamics and the origin of evolution. Proc Natl Acad Sci U S A. 2008 Sep 30;105(39):14924-7. doi: 10.1073/pnas.0806714105. Epub 2008 Sep 12. PMID 18791073 ; PMCID: PMC2567469
^ Lehman N. A case for the extreme antiquity of recombination. J Mol Evol. 2003 Jun;56(6):770-7. doi: 10.1007/s00239-003-2454-1. PMID 12911039
^ Smail BA, Clifton BE, Mizuuchi R, Lehman N. Spontaneous advent of genetic diversity in RNA populations through multiple recombination mechanisms. RNA. 2019 Apr;25(4):453-464. doi: 10.1261/rna.068908.118. Epub 2019 Jan 22. PMID 30670484 ; PMCID: PMC6426292
公有领域材料 来自NCBI 的文档《Science Primer》 。
Michael J. McDonald, Daniel P. Rice, Michael M. Desai: Sex speeds adaptation by altering the dynamics of molecular evolution. In: Nature. 2016, doi:10.1038/nature17143