|
Inibitore enzimaticoCon il termine inibitore enzimatico si indica una molecola in grado di instaurare un legame chimico con un enzima, diminuendone così l'attività. L'inibitore infatti può intralciare l'enzima nella catalisi della sua reazione, per esempio impedendo al substrato di entrare nel sito attivo dell'enzima stesso. Il legame tra enzima ed inibitore può essere reversibile o irreversibile. Gli inibitori irreversibili solitamente reagiscono con l'enzima, modificando chimicamente residui amminoacidici fondamentali per l'attività. Al contrario, gli inibitori reversibili si legano non-covalentemente e producono diversi tipi di inibizione a seconda che si leghino all'enzima, al complesso enzima-substrato, o ad entrambi. Agli enzimi possono legarsi anche molecole in grado di potenziarne l'attività, dette attivatori enzimatici. Alcune di queste molecole non sono indispensabili per la catalisi, ma la loro presenza la facilita. Altri, come i cofattori (es. ioni metallici come calcio, ferro, manganese) e coenzimi (es. prodotti derivanti da vitamine), sono indispensabili e la loro assenza non rende possibile il meccanismo enzimatico. Quindi alcuni inibitori enzimatici possono anche competere per cofattori e coenzimi e ridurre l'attività catalitica in questo modo. Inibitori sintetici e naturaliDal momento che il blocco o l'inibizione dell'attività enzimatica può causare la morte selettiva di microrganismi patogeni o correggere uno sbilanciamento metabolico, la maggior parte dei farmaci in commercio sono proprio inibitori enzimatici. Per questo la loro scoperta e il loro miglioramento è un settore importante della ricerca nel campo della biochimica e della farmacologia. Un inibitore enzimatico di carattere medicinale è spesso giudicato in base alla sua specificità (la sua mancanza di legami con altre proteine) ed alla sua potenza (la sua costante di dissociazione, che indica la concentrazione necessaria ad inibire l'enzima). Un'elevata specificità e potenza assicurano che un farmaco avrà pochi effetti collaterali e pertanto minore tossicità. Inibitori comunemente usati in campo agro-alimentare sono molti fitofarmaci. Gli inibitori enzimatici non sono però esclusivamente molecole sintetiche: essi sono spesso composti naturali, coinvolti nella regolazione del metabolismo. Ad esempio, gli enzimi di un pathway metabolico spesso sono inibiti dai prodotti finali del pathway stesso. Questo tipo di feedback negativo rallenta il flusso attraverso un pathway quando i prodotti cominciano a formarsi ed è un'importante strategia per mantenere l'omeostasi in una cellula. Altri inibitori enzimatici cellulari sono proteine che si legano ed inibiscono uno specifico enzima bersaglio. Questo permette di controllare gli enzimi che potrebbero danneggiare la cellula, come le proteasi o le nucleasi. Un importante esempio è l'inibitore della ribonucleasi, che si lega a questo enzima in uno dei più stretti legami proteina-proteina conosciuti.[1] Inibitori enzimatici naturali possono anche essere veleni ed essere usati come difesa contro i predatori o come arma per uccidere una preda. Inibitori reversibiliSono molecole che si legano non covalentemente all'enzima motivo per cui dopo la loro rimozione l'enzima torna ad essere funzionante. Tipi di inibitori reversibili  Gli inibitori reversibili si legano agli enzimi con interazioni non covalenti, come il legame idrogeno, l'interazione idrofobica ed il legame ionico. I legami multipli deboli tra l'inibitore ed il sito attivo si combinano per produrre legami forti e specifici. Al contrario dei substrati e degli inibitori irreversibili, gli inibitori reversibili generalmente non danno luogo a reazioni chimiche quando si legano all'enzima e possono essere facilmente rimossi per diluizione o dialisi. Vi sono tre tipi di inibitori enzimatici reversibili, classificati in base all'effetto causato dalle variazioni in concentrazione del substrato enzimatico sull'inibitore.[2]
Descrizione quantitativa dell'inibizione reversibileL'inibizione reversibile può essere descritta quantitativamente nei termini del legame dell'inibitore all'enzima ed al complesso enzima-substrato, e dei suoi effetti sulle costanti cinetiche dell'enzima. Nel classico schema di Michaelis–Menten, un enzima (E) si lega al suo substrato (S) per formare il complesso enzima-substrato ES. Durante la catalisi, questo complesso si rompe per rilasciare il prodotto P e l'enzima libero. L'inibitore (I) si può legare a E o a ES con costanti di dissociazione Ki o Ki', rispettivamente.
Se un enzima ha vari substrati, gli inibitori possono mostrare tipi differenti di inibizione a seconda del substrato in questione. Ciò risulta dal sito attivo contenente due diversi siti di legame, uno per ciascun substrato. Per esempio, un inibitore può competere con il substrato A per il primo sito di legame, ma essere un inibitore non competitivo rispetto al substrato B nel secondo sito di legame.[3] Misurazione delle costanti di dissociazione di un inibitore reversibile Come già discusso, un inibitore enzimatico è caratterizzato dalle due costanti di dissociazione: Ki (relativa a E) e Ki' (relativa al complesso ES). La costante Ki può essere misurata direttamente con vari metodi; un metodo estremamente accurato è la calorimetria isotermica di titolazione, in cui viene misurata la concentrazione dell'inibitore in una soluzione con enzimi ed il calore rilasciato o assorbito.[4] La costante Ki', invece, è difficile da misurare direttamente, dal momento che il complesso enzima-substrato ha vita breve ed è suscettibile alla reazione chimica che forma il prodotto. Pertanto, la Ki' è talvolta misurata indirettamente, osservando l'attività enzimatica su vari substrati e le concentrazioni dell'inibitore, e interpolando i dati[5] in una equazione di Michaelis–Menten modificata: ove i fattori modificanti α e α' sono definiti dalla concentrazione dell'inibitore e dalle due costanti di dissociazione Così, in presenza dell'inibitore, l'effettiva Km e Vmax dell'enzima divengono rispettivamente (α/α')Km e (1/α')Vmax. In ogni caso, l'equazione di Michaelis-Menten modificata assume che il legame dell'inibitore con l'enzima abbia raggiunto l'equilibrio, il che può essere un processo molto lento per gli inibitori con costanti di dissociazione molto basse (sub-nanomolari). In questi casi, è più pratico trattare l'inibitore come un inibitore irreversibile (come descritto in seguito); tuttavia, può essere ancora possibile stimare cineticamente Ki' se Ki è misurabile indipendentemente. Gli effetti dei diversi tipi di inibitori enzimatici reversibili sull'attività enzimatica possono essere visualizzati con rappresentazioni grafiche dell'equazione di Michaelis–Menten, come il Lineweaver–Burk ed il diagramma di Eadie-Hofstee. Per esempio, nel diagramma Lineweaver-Burk le linee dell'inibizione competitiva si intersecano sull'asse y, mostrando che tali inibitori non influenzano Vmax. Similmente, le linee dell'inibizione non-competitiva si intersecano sull'asse x, in quanto tali inibitori non influenzano Km. Tuttavia, può essere difficile stimare Ki e Ki' accuratamente da tali grafici[6], motivo per cui si è soliti stimare queste costanti facendo uso della più affidabile regressione non lineare descritta in precedenza. Casi particolari
Esempi di inibitori reversibili Dal momento che gli enzimi hanno lo scopo di legarsi saldamente ai loro substrati, e la maggior parte degli inibitori reversibili si legano al sito attivo dell'enzima, non sorprende come taluni di questi inibitori siano molto simili per struttura al substrato dell'enzima bersaglio. Un esempio di questa mimetica è costituito dagli inibitori delle proteasi, come il ritonavir, una classe di farmaci antiretrovirali usati ad esempio per curare le infezioni da HIV.[9] Gli inibitori sono spesso in grado di imitare lo stato di transizione, l'intermedio di una reazione catalizzata dall'enzima. Questo assicura che l'inibitore sfrutti l'effetto stabilizzante dello stato di transizione dell'enzima, dando luogo ad una migliore affinità di legame (Ki inferiore alla Km) rispetto al substrato. Un esempio di tale inibitore dello stato di transizione è l'antivirale oseltamivir, che imita la natura planare dell'anello dello ione ossonio nella reazione dell'enzima virale neuraminidasi.  Tuttavia, non tutti gli inibitori reversibili mimano le strutture dei substrati. La struttura di un altro inibitore della proteasi dell'HIV come il tipranavir, ad esempio, non è di tipo peptidico e non ha evidenti similitudini strutturali con il substrato. Questi inibitori non-peptidici possono essere più stabili di quelli contenenti legami peptidici, in quanto non costituiscono substrato per le peptidasi e sono più difficilmente degradati nella cellula. Nell'ideazione dei farmaci occorre anche una valutazione attenta delle concentrazioni necessarie per indurre una competizione efficace. Ad esempio, è possibile inibire le protein chinasi attraverso molecole con struttura chimica simile all'ATP (uno dei substrati di tali enzimi), ma tali farmaci dovranno competere con le alte concentrazioni di ATP della cellula. In alcuni casi, dunque, si preferisce mettere a punto inibitori delle protein chinasi che competono per il loro sito di legame con i relativi substrati proteici, presenti nelle cellule a concentrazioni molto inferiori alla concentrazione standard di ATP. Basterà dunque una concentrazione molto minore di questo secondo tipo di inibitore per ottenere un risultato simile a quello indotto dal primo tipo di farmaco.[10] Inibitori irreversibiliTipi di inibizione irreversibile Gli inibitori irreversibili solitamente modificano covalentemente l'enzima, e l'inibizione non può essere pertanto successivamente neutralizzata. Spesso tali inibitori contengono gruppi funzionali reattivi come aldeidi, aloalcani o alcheni. Questi gruppi elettrofili reagiscono con le catene laterali degli amminoacidi addotti covalenti. I residui modificati sono quelli con catene laterali contenenti gruppi nucleofili come l'ossidrile o il sulfidrile (ad esempio la serina, bersagliabile dal diisopropilfluorofosfato, la cisteina, la treonina o la tirosina).[11] Il concetto di inibizione irreversibile è in ogni caso diverso da quello di inattivazione irreversibile dell'enzima. Gli inibitori irreversibili sono solitamente specifici per una classe di enzimi e non inattivano tutte le proteine; non funzionano distruggendo la struttura proteica ma alterando specificamente il sito attivo del loro bersaglio. Gli inibitori irreversibili, dunque, non sono correlati a fenomeni legati a livelli estremi di pH o di temperatura, che causano solitamente la denaturazione aspecifica di tutte le strutture proteiche presenti in soluzione, oppure all'idrolisi aspecifica mediata dall'aggiunta nel mezzo di acido cloridrico concentrato caldo, che genera una rottura dei legami peptidici ed un rilascio di amminoacidi liberi.[12] Analisi dell'inibizione irreversibile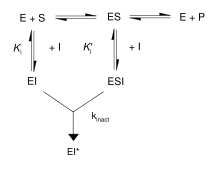 Gli inibitori irreversibili formano un complesso reversibile non-covalente con l'enzima (EI o ESI), che reagisce per produrre un complesso covalentemente modificato e definito EI* o dead-end (senza uscita). Il rapporto a cui si forma EI* è detto rapporto di inattivazione (o kinact). Dal momento che la formazione di EI può competere con ES, il legame degli inibitori irreversibili può essere evitato mediante competizione con un substrato o con un secondo inibitore reversibile. I meccanismi di legame e di inattivazione di questo tipo di reazione possono essere investigati incubando l'enzima e l'inibitore e misurando la quantità di attività rimanente al trascorrere del tempo. L'attività decresce in maniera dipendente dal tempo, di solito seguendo il decadimento esponenziale. Riportando questi dati in una equazione di rapporto si ottiene il tasso di inattivazione a quella concentrazione dell'inibitore. Questa operazione è ripetuta a diverse concentrazioni dell'inibitore. Se un complesso reversibile EI è coinvolto, il tasso di inattivazione sarà saturabile e, disegnando la curva, si otterrà la kinact e la Ki.[13] Un altro metodo largamente usato in queste analisi è la spettrometria di massa. In questo caso un'accurata misura della massa dell'enzima originario non modificato e dell'enzima inattivato permette di calcolare la crescita in massa causata dalla reazione con l'inibitore e mostra la stechiometria della reazione. Questo è indagabile usando uno spettrometro di massa MALDI-TOF. Una tecnica complementare, l'impronta digitale proteica, implica la digestione della proteina originaria e modificata mediante una proteasi come la tripsina. Ciò produce un insieme di peptidi che possono essere analizzati con uno spettrometro di massa. Il peptide la cui massa cambia dopo la reazione con l'inibitore è quello che contiene il sito di modificazione. Casi particolari Spesso alcuni inibitori reversibili sono in grado di legarsi così strettamente al loro bersaglio da apparire come irreversibili. Questi inibitori mostrano costanti cinetiche simili a quelle degli inibitori irreversibili covalenti. In questi casi, alcuni di questi inibitori si legano rapidamente all'enzima nel complesso a bassa affinità EI e questo successivamente va incontro ad un più lento riassestamento verso un complesso EI* legato molto saldamente. Tale comportamento cinetico è definito legame lento[15] Questo lento assestamento dopo il legame provoca spesso una modifica di conformazione, legata al fatto che l'enzima si stringe attorno alla molecola dell'inibitore. Esempi di inibitori a legame lento sono costituiti da importanti farmaci, come il metotrexato[16], l'allopurinolo[17] e la forma attivata dell'aciclovir.[18] Esempi di inibitori irreversibili Il diisopropilfluorofosfato (DFP) è un inibitore delle proteasi, che idrolizza il legame fosforo-fluoro lasciando il fosfato legato alla serina presente nel sito attivo, inibendo in modo irreversibile l'enzima.[19] In modo analogo, il DFP reagisce anche col sito attivo dell'acetilcolinesterasi nelle sinapsi dei neuroni, ed è quindi anche una potente neurotossina, la cui dose letale è fissata a meno di 100 mg.[20] Un altro esempio di inibitore irreversibile delle proteasi è la iodoacetamide, che si lega ad un residuo di cisteina dell'enzima. Lo zolfo contenuto nell'aminoacido reagisce con tale inibitore, si forma un tioetere liberando acido ioidrico (HI). L'enzima quindi perde la sua attività. L'inibizione suicida è un tipo inusuale di inibizione irreversibile che si ha quando l'enzima trasforma l'inibitore in una forma reattiva nel suo sito attivo. Un esempio è l'α-difluorometilornitina (o DFMO), inibitore della biosintesi delle poliammine ed analogo dell'amminoacido ornitina, usato per trattare la tripanosomiasi africana (febbre del sonno). L'ornitina decarbossilasi può catalizzare la decarbossilazione del DFMO invece che dell'ornitina. Conseguenza di tale reazione è l'eliminazione di un atomo di fluoro, che converte questo intermedio catalitico nell'immina coniugata, una specie altamente elettrofila, che reagisce con un residuo di cisteina o di lisina nel sito attivo per inibire irreversibilmente l'enzima.[14] Dal momento che l'inibizione irreversibile dà spesso luogo all'iniziale formazione di un complesso non-covalente EI, è talvolta possibile per un inibitore legarsi ad un enzima in più di un modo. Per esempio, un inibitore chiamato mostarda di chinacrina può inibire in due modi diversi la tripanotione reduttasi del protozoo Trypanosoma cruzi. In un caso può legarsi reversibilmente, nell'altro covalentemente, dal momento che reagisce con un residuo amminoacidico attraverso la sua azoiprite.[21] Scoperta e progettazione di inibitori I nuovi farmaci sono il prodotto di una lunga fase di sviluppo, il cui primo passaggio è spesso la scoperta di un nuovo inibitore enzimatico. In passato il solo modo per scoprire questi nuovi inibitori era l'approccio per "prova ed errore": si sottoponeva un enzima bersaglio all'azione di una gran varietà di composti sperando di scoprire qualche utile interazione inibitoria. Questo metodo è ancora utilizzato con successo ed è stato ampliato dall'approccio della chimica combinatoria, che produce rapidamente un gran numero di nuovi composti, e dalle tecnologie HTS, che permettono di sfruttare queste ampie librerie di composti chimici come inibitori utili. Recentemente, si sta applicando un approccio alternativo: la progettazione farmacologica razionale, che utilizza la struttura tridimensionale del sito attivo enzimatico per individuare le molecole che possono agire da inibitori. Queste ipotesi sono poi verificate e uno dei composti testati diventerà il nuovo inibitore. Questo nuovo inibitore viene poi usato per ottenere un complesso EI, che sarà quindi analizzato per determinare, e successivamente ottimizzare, il tipo di legame che si instaura tra la molecola e l'enzima all'interno del sito attivo. Questo ciclo di prove e miglioramenti può essere ripetuto fino a produrre un inibitore sufficientemente potente (con una costante di dissociazione di <10−9 M[22]). Usi degli inibitoriGli inibitori enzimatici si trovano in natura e sono anche sintetizzati artificialmente per usi nel campo della farmacologia e della biochimica. I veleni naturali sono anch'essi spesso degli inibitori enzimatici sviluppati per difendere piante e animali dai predatori. Queste tossine naturali includono alcuni dei composti più velenosi ad oggi conosciuti. Gli inibitori artificiali sono spesso usati come farmaci, ma possono anche essere insetticidi come il malatione, erbicidi come il glifosato, o disinfettanti come il triclosan. Farmacologia
L'uso più comune degli inibitori enzimatici è rappresentato dalla sintesi di farmaci. Molti di essi, infatti, hanno come bersaglio enzimi umani e possono correggere alcune situazioni patologiche. Tuttavia, non tutti i farmaci sono inibitori enzimatici. Alcuni, come i farmaci antiepilettici, alterano l'attività enzimatica causando una maggiore o minore produzione dell'enzima. Questi effetti sono chiamati induzione e inibizione enzimatica e derivano da alterazioni dell'espressione genica, che non sono legate all'inibizione enzimatica finora descritta. Altri farmaci interagiscono con bersagli cellulari diversi dagli enzimi, come i canali ionici o i recettori di membrana. Un esempio interessante di inibitore enzimatico medicinale è il sildenafil, principio attivo del Viagra, (figura a destra) un trattamento comune per le disfunzioni erettili maschili. Questo composto è un potente inibitore della fosfodiesterasi-5, l'enzima che degrada la molecola guanosina monofosfato ciclica.[23] Questa molecola, che svolge funzione di segnale, provoca il rilassamento della muscolatura liscia e consente al sangue di affluire all'interno del corpo cavernoso, causando l'erezione. Il farmaco, abbassando l'attività dell'enzima che blocca il segnale, mantiene quest'ultimo per un più lungo periodo di tempo. Un altro esempio della somiglianza strutturale di alcuni inibitori ai substrati degli enzimi bersaglio è rappresentato nella figura, dove si confronta il farmaco metotrexate con l'acido folico. L'acido folico è la forma ossidata del substrato del diidrofolato reduttasi, un enzima fortemente inibito dal methotrexate. Il Methotrexate blocca l'azione del diidrofolato reduttasi fermando quindi la biosintesi della timidina. Questo blocco della biosintesi nucleotidica è selettivamente tossica per le cellule che crescono rapidamente, e per questo motivo il methotrexate è spesso usato nella chemioterapia.[24] Gli inibitori usati in farmacologia servono anche ad inibire gli enzimi necessari alla sopravvivenza dei microorganismi patogeni. Ad esempio, i batteri sono protetti da una spessa parete cellulare formata da peptidoglicani. Molti antibiotici come la penicillina e la vancomicina inibiscono gli enzimi che li producono.[25] Questo provoca una perdita di resistenza della parete e la sua rottura. Nella figura, una molecola di penicillina si lega al suo bersaglio, la transpeptidasi del batterio Streptomyces R61. La ricerca sui farmaci è facilitata quando un enzima essenziale per la sopravvivenza di un patogeno è assente, o presente in una forma molto diversa, nell'uomo. Nell'esempio sopra, gli esseri umani non producono peptidoglicani, e quindi gli inibitori di questo processo sono selettivamente tossici per i batteri. La tossicità selettiva degli antibiotici può anche sfruttare le differenze nella struttura dei ribosomi batterici, o quelle relative alla biosintesi degli acidi grassi. Il concetto di tossicità selettiva risale agli studi pionieristici di chemioterapia condotti dal medico tedesco Paul Ehrlich nei primi anni del Novecento. Controllo metabolicoGli inibitori enzimatici sono molto importanti anche nel controllo metabolico. Molti pathways metabolici nella cellula sono inibiti da metaboliti che controllano l'attività enzimatica attraverso la regolazione allosterica o l'inibizione del substrato. Un esempio è costituito dal pathway glicolitico. Questo pathway catabolico consuma glucosio e produce ATP, NADH e piruvato. Un passaggio chiave nella regolazione della glicolisi è la reazione iniziale catalizzata dalla fosfofruttochinasi-1 (PFK1). Quando i livelli di ATP crescono, l'ATP si lega ad un sito allosterico nella PFK1 per abbassare la velocità di reazione dell'enzima; la glicolisi è inibita e la produzione di ATP scende. Questo controllo basato su un feedback negativo aiuta a mantenere una concentrazione stabile di ATP nella cellula. Tuttavia, i pathways metabolici non sono regolati solo attraverso l'inibizione in quanto l'attivazione è ugualmente importante. Rispetto alla PFK1, il fruttosio 2,6-difosfato e l'ADP sono esempi di attivatori allosterici.[26] L'inibizione fisiologica degli enzimi può anche essere prodotta da inibitori proteici specifici. Questo meccanismo avviene nel pancreas, che sintetizza molti precursori di enzimi digestivi noti come zimogeni. Molti di essi sono attivati dalla proteasi tripsina, perciò è importante inibire l'attività della tripsina nel pancreas per prevenire l'"autodistruzione" dell'organo. Un modo in cui l'attività della tripsina viene controllata è la produzione di uno specifico e potente inibitore nel pancreas. Questo inibitore si lega saldamente alla tripsina, bloccando l'attività che potrebbe danneggiare seriamente l'organo.[27] Nonostante l'inibitore della tripsina sia una proteina, essa evita la sua idrolisi, come substrato dalla proteasi, escludendo l'acqua dal sito attivo della tripsina e destabilizzando lo stato di transizione.[28] Altri esempi di inibitori enzimatici fisiologici proteici includono l'inibitore barstar della ribonucleasi batterica barnasi[29] e gli inibitori delle protein fosfatasi.[30] Inibitori dell'acetilcolinesterasiL'Acetilcolinesterasi (AChE) è un enzima presente in tutti gli animali, dagli insetti all'uomo. È essenziale per la funzionalità delle cellule nervose per la sua capacità di scindere il neurotrasmettitore acetilcolina nei suoi costituenti, l'acetato e la colina. Questo meccanismo è pressoché unico tra i neurotrasmettitori dal momento che la maggior parte, tra cui la serotonina, la dopamina, e la norepinefrina, non è scomposta, ma assorbita dalla membrana postisinaptica. Un gran numero di inibitori dell'AChE sono utilizzati sia in medicina che in agricoltura. Inibitori competitivi reversibili, come l'edrofonio, la fisostigmina, e la neostigmina, sono usati nel trattamento della Miastenia gravis ed in anestesia. Altri esempi di inibitori dell'acetilcolinesterasi sono gli insetticidi di terza generazione, quali i carbammati e gli organofosfati. Veleni naturali Animali e piante, nel corso della loro evoluzione, hanno sviluppato la capacità di sintetizzare un'ampia gamma di sostanze velenose, tra cui metaboliti secondari, peptidi e proteine che possono agire quali inibitori. Le tossine naturali sono di solito piccole molecole organiche e sono così varie e diffuse che, probabilmente, vi sono inibitori naturali per la maggior parte dei processi metabolici.[31] I processi metabolici cui si rivolgono i veleni naturali non interessano soltanto gli enzimi nei pathways metabolici, ma possono anche includere l'inibizione di recettori, proteine canale o strutturali nella cellula. Per esempio, il paclitaxel (taxolo), una molecola organica presente nel Taxus, si lega strettamente ai dimeri di tubulina e inibisce la loro unione nei microtubuli del citoscheletro.[32] Molti veleni naturali agiscono come neurotossine che provocano paralisi e possono quindi condurre alla morte. Tali veleni sono usati sia come mezzi di difesa contro i predatori, sia per cacciare e catturare le prede. Alcuni di questi inibitori naturali, nonostante i loro attributi tossici, a dosi più basse sono utili per usi terapeutici.[33] Un esempio di neurotossina sono i glucoalcaloidi, presenti nella famiglia delle Solanaceae (patate, pomodori e melanzane), che sono inibitori dell'acetilcolinesterasi. L'inibizione di questo enzima causa una produzione incontrollata di acetilcolina, la paralisi muscolare e quindi la morte. La neurotossicità può anche derivare dall'inibizione dei recettori; per esempio, l'atropina prodotta dall'Atropa belladonna funziona come un antagonista competitivo dei recettori dell'acetilcolina.[34] Nonostante molte tossine naturali siano metaboliti secondari, questi veleni includono anche peptidi e proteine. Un esempio di peptide tossico è l'α-amanitina, presente nell'amanita falloide e in specie correlate. Si tratta di un potente inibitore enzimatico, che impedisce la trascrizione del DNA da parte dell'RNA polimerasi II.[35] La tossina microcistina, presente nelle alghe, è anch'essa un peptide ed è un inibitore della protein fosfatasi.[36] Questa tossina può contaminare le fonti d'acqua dopo la fioritura algale ed è un noto agente cancerogeno che può anche causare emorragie epatiche acute e la morte, se assunta a dosi elevate.[37] Anche le proteine possono essere veleni naturali, come l'inibitore della tripsina che si trova in alcune lenticchie, come mostrato nella figura sopra. Una classe meno comune di tossine è rappresentata dagli enzimi tossici, che agiscono come inibitori irreversibili dei loro bersagli e modificano chimicamente i loro enzimi substrato. Un esempio è dato dalla ricina, una potentissima tossina proteica rilevata nei chicchi del Ricinus communis. Questo enzima è una glicosidasi che inibisce i ribosomi. Poiché la ricina è un inibitore catalitico irreversibile, una sola molecola di questo composto è in grado di uccidere una cellula.[38] Note
Voci correlate
Altri progetti
Collegamenti esterni
|
![{\displaystyle V={\frac {V_{max}[S]}{\alpha K_{m}+\alpha ^{\prime }[S]}}={\frac {(1/\alpha ^{\prime })V_{max}[S]}{(\alpha /\alpha ^{\prime })K_{m}+[S]}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/4a8f0a9dda1d308de7f090f99c2833f944f11a09)
![{\displaystyle \alpha =1+{\frac {[I]}{K_{i}}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/57fcf54938a9784f9313437681b220079ff43ee5)
![{\displaystyle \alpha ^{\prime }=1+{\frac {[I]}{K_{i}^{\prime }}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/8b7d75a61e059ecea4053a1c7afb91f9cf39a2ab)


