|
Western blot El Western blot, inmunoblot o electrotransferencia, es una técnica analítica usada en biología celular y molecular para identificar proteínas específicas en una mezcla compleja de proteínas, tal como la que se presenta en extractos celulares o de tejidos. La técnica utiliza tres etapas para lograr esto: separación por tamaño, transferencia a un soporte sólido y, finalmente, visualización mediante la marcación de proteínas con el uso de anticuerpos primarios o secundarios apropiados.[1] Esta técnica es hoy en día imprescindible en varios campos de la biología, como la biología molecular, la bioquímica, la biotecnología o la inmunología. Mediante una electroforesis en gel se separan las proteínas atendiendo al criterio que se desee: peso molecular, estructura, hidrofobicidad, etc. Hay casi tantas posibilidades como tipos de electroforesis existen. Luego son transferidas a una membrana adsorbente (típicamente de nitrocelulosa o de PVDF) para poder buscar la proteína de interés con anticuerpos específicos contra ella. Finalmente, se detecta la unión antígeno-anticuerpo por actividad enzimática o fluorescencia, entre otros métodos. De esta misma forma se puede estudiar la presencia de la proteína en el extracto y analizar su cantidad relativa respecto a las otras proteínas.[2][3] La técnica «Western blot» fue desarrollada en la Universidad de Stanford. El nombre (Western, occidental en inglés) le fue dado por W. Neal Burnette, y consiste en un juego de palabras con una técnica análoga pero que usa ADN, el Southern (sureño en inglés) blot, que en este caso debe su nombre a su descubridor, Edwin Southern. Otras técnicas que fueron nombradas siguiendo este criterio son el Northern (norteño en inglés) (en el que se separa e identifica ARN), el Eastern (oriental en inglés) blot y el Southwestern (del suroeste en inglés) blot.[2][4] Pasos del Western blotPreparación de la muestraLas muestras pueden ser tomadas de un tejido entero o de un cultivo celular:
 Electroforesis en gelLas proteínas de la muestra serán separadas mediante una electroforesis en gel en función de uno o varios (en las electroforesis bidimensionales) de estos criterios: punto isoeléctrico, peso molecular y carga eléctrica. La naturaleza de la separación depende del tratamiento de la muestra y de la naturaleza del gel. La electroforesis en gel más frecuente, conocida como SDS-PAGE, hace uso de gel de poliacrilamida y de tampón con dodecilsulfato (SDS). En esta técnica las proteínas sufren un tratamiento por agentes reductores que provocan la pérdida de las estructuras secundaria y terciaria y mantiene los polipéptidos en este estado desnaturalizado. De este modo, la estructura tridimensional de las proteínas no influye en la electroforesis, y pueden separarse únicamente en función del tamaño. 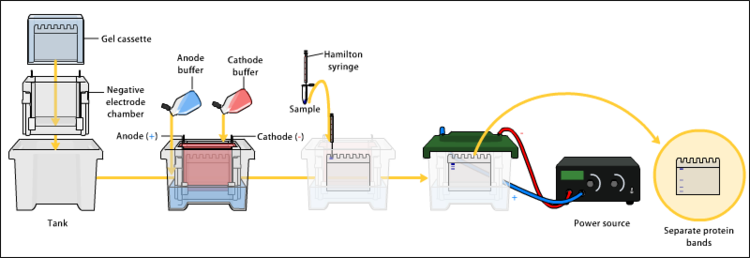 TransferenciaPara que las proteínas sean accesibles a la detección por anticuerpos, se las transfiere desde el gel de poliacrilamida a una membrana de nitrocelulosa o de PVDF. Esta transferencia puede realizarse por difusión, por vacío o por acción de un campo eléctrico (electrotransferencia). Las membranas que se emplean en el Western blot se caracterizan por su capacidad de unir proteínas de forma inespecífica (es decir, se adhieren a todas las proteínas con idéntica afinidad):
Las membranas de nitrocelulosa son más baratas que las de PVDF, aunque también son más frágiles, tienden a tornarse quebradizas cuando se secan y no pueden ser sometidas a varias pruebas consecutivas.[7] También pueden usarse otros soportes, como las membranas de nailon o el papel activado. Sin embargo, no son tan usados como los anteriores. Difusión simpleEn la transferencia por difusión simple se ponen en contacto las superficies del gel electroforético y de la membrana y, sobre ésta, se dispone un taco de papeles de filtro. Con el fin de facilitar el proceso, puede ponerse encima una placa de vidrio y un objeto pesado. Este montaje se coloca sobre un tampón, que ascenderá por capilaridad hacia el papel de filtro arrastrando consigo las proteínas. Al llegar a la membrana, quedarán retenidas en ella. Este protocolo no está muy extendido actualmente, puesto que la cantidad de proteína transferida a la membrana es muy baja, mucho menor que con la electrotransferencia. Sin embargo, ha ganado cierta popularidad por una modificación que permite obtener múltiples transferencias de un mismo gel, permitiendo de esta forma realizar varias pruebas sobre geles virtualmente idénticos. Esta modificación es viable cuando no es necesaria una transferencia cuantitativa de proteína.[8][7] Transferencia al vacíoEn esta técnica, se añade el poder de succión de una bomba conectada a un sistema de secado de planchas de gel, el cual lleva las proteínas desde el gel hasta la superficie de la membrana de nitrocelulosa. Este método es válido tanto para proteínas de alto como de bajo peso molecular.[9][7] ElectrotransferenciaEste método se basa en una corriente eléctrica y un tampón de transferencia para llevar las proteínas desde el gel hacia la membrana. Para ello, se apilan en el orden descrito los siguientes elementos (del polo negativo o ánodo al positivo o cátodo): esponja, varios papeles de filtro empapados en tampón de transferencia, gel, membrana, más papeles de filtro empapados y otra esponja. Este montaje, llamado coloquialmente sandwich, se dispone en el sistema de transferencia y se aplica una corriente eléctrica, de magnitud acorde a los materiales empleados, al tiempo disponible,... Las proteínas del gel se desplazan hacia el polo positivo y quedan atrapadas por la membrana.[3]  Tras la transferencia, se suele proceder a la tinción del gel con Azul de Coomassie (un colorante de proteínas) para comprobar que en efecto una parte importante del material proteico ha pasado a la membrana. La electrotransferencia es hoy en día la técnica de transferencia más usada gracias a la velocidad del proceso y al elevado porcentaje de proteína que se transfiere (especialmente en comparación con las otras técnicas). BloqueoPuesto que la membrana escogida necesita poder unirse a las proteínas de forma inespecífica, es preciso bloquear los lugares de unión que han quedado libres tras la transferencia. En caso contrario, el anticuerpo (de naturaleza proteica) empleado en la detección puede unirse a ellos, dificultando la distinción del complejo antígeno-anticuerpo que se forma con la proteína que se busca. En el bloqueo se incuba la membrana con una solución de proteínas, típicamente de albúmina de suero bovino o ASB (más conocida por sus siglas en inglés, BSA), leche en polvo o caseína, con una diminuta fracción de detergente, como Tween 20 o Tritón X-100. Las proteínas en solución se unirán a todos aquellos lugares de unión de la membrana que no estén ya ocupados por las transferidas desde el gel. De este modo, el anticuerpo sólo podrá unirse a su antígeno específico, reduciendo así el ruido de fondo y los falsos positivos. DetecciónEn la detección se comprueba la presencia en la membrana de una determinada proteína. Para ello se emplea un anticuerpo específico contra ella unido a una enzima que, en presencia de su sustrato, catalice una reacción colorimétrica (produce color). De esta forma, se hace patente la unión con el antígeno, la proteína, así como su localización. El proceso tradicionalmente se ha realizado en dos pasos, aunque ahora es posible la detección en un único paso, lo cual es útil en ciertos campos. Método en dos pasosLos dos pasos de los que consta este método de detección son la unión del anticuerpo primario y la del anticuerpo secundario.  Anticuerpo primarioEl primer paso es permitir la unión de un anticuerpo primario contra la proteína buscada. Este se consigue inoculando dicha proteína o uno de sus epítopos (regiones capaces de desencadenar la respuesta inmune) a un animal (como un conejo o una cabra) o a un cultivo celular. Además, como la proteína se ha desnaturalizado durante la electroforesis en gel, es preciso que el anticuerpo reconozca específicamente la proteína desnaturalizada. La presencia del elemento extraño debería desencadenar una respuesta inmune cuyo resultado incluya la producción del anticuerpo, primario en este caso, ya que reconoce la proteína. Así pues, tras el bloqueo se incuba en agitación moderada la membrana con una disolución de anticuerpo primario (0,5 - 5 μg/ml). La disolución consiste en un tampón salino, con cloruro de sodio por lo general, con un pH cercano a la neutralidad, una pequeña fracción de detergente y, en ocasiones, proteínas disueltas, como ASB o leche en polvo. La membrana junto con la disolución pueden ser introducidas en una pequeña bolsa de plástico. Esta incubación puede durar desde 30 minutos a una noche entera. La temperatura a la que puede tener lugar es igualmente variada; en general, las elevadas temperaturas favorecen las uniones más específicas.[cita requerida] Anticuerpo secundarioTras lavar la membrana para eliminar el anticuerpo primario que no se ha unido, se expone al anticuerpo secundario. Este reconoce de forma específica una región concreta del anticuerpo primario. Suelen estar marcados para ser detectables (unión a biotina, a una enzima reporter como la fosfatasa alcalina o la peroxidasa del rábano (HRP), etc.). Varios de estos anticuerpos se unirán a cada anticuerpo primario, amplificando la señal. Los anticuerpos secundarios proceden de animales o de cultivos de hibridomas de origen animal. Por ejemplo, un anticuerpo secundario "anti-ratón" es aquel capaz de reconocer casi todos los anticuerpos primarios obtenidos de ratones. Igualmente hay anticuerpos "anti-cabra", "anti-conejo"... Su uso presenta ciertas ventajas: económicas, por permitir a un laboratorio compartir una única fuente de anticuerpos secundarios; y dan resultados mucho más consistentes. También pueden emplearse proteínas no anticuerpos, como las proteínas A y G. Radiactividad Es una alternativa al uso de anticuerpos secundarios que consiste en proteínas de unión a anticuerpos marcadas radiactivamente. Esta proteína puede ser, por ejemplo, la proteína A de Staphylococcus aureus[4] o la proteína G[10] marcadas con 125I (un isótopo radiactivo de yodo). Las proteínas mencionadas tienen la capacidad de unirse a anticuerpos de forma inespecífica, por lo que se unirán al primario. Este método apenas se usa actualmente, por ser significativamente más caro, peligroso y lento que los otros. Sin embargo, presenta ventajas: es muy sensible, da bandas que permiten una precisa cuantificación; y la imagen autoradiográfica puede ser reproducida fácilmente y con gran precisión para su publicación.[7] Anticuerpo unido a una enzima Un mecanismo de detección simple y rápido consiste en unir al anticuerpo secundario enzimas que catalicen la transformación de un sustrato soluble en un producto insoluble. De esta forma, en el posterior análisis quedará una mancha allí donde esté la proteína de interés. Enzimas como la peroxidasa de rábano (HRP) o una fosfatasa alcalina son válidas para este mecanismo.[7] Por ejemplo, la peroxidasa puede catalizar la oxidación del 4-cloronaftol en presencia de un 1% de peróxido de hidrógeno. El resultado es que el primero forma una mancha de precipitado oscura.[11] Otras técnicas, como ELISA o ELISPOT, también emplean este método de detección. Otro método más sofisticado consiste en la unión de una enzima que catalice una reacción quimioluminiscente. Las anteriormente citadas enzimas también son válidas en este caso, aunque cada una usa un agente quimioluminiscente diferente. En el caso de la peroxidasa, cataliza la oxidación del luminol en presencia de peróxido de hidrógeno. La fosfatasa alcalina cataliza la defosforilación del adamantil-1-2-dioxetano fosfato, reacción que genera luz. Si se coloca una película fotográfica sobre la membrana, la exposición a la luz que se desprende en la reacción permite detectar la actividad enzimática. Marcaje fluorescenteOtro método basado en el mismo principio emplea anticuerpos unidos a fluorocromos del infrarrojo cercano, como el 2 - metoxi - 2,4 - difenil - 3 (2H) - furanona (MDPF) o el rojo Nilo. Estos compuestos emiten una cantidad de luz constante (estado estático) cuando se excitan de manera constante, permitiendo una detección muy precisa y una cuantificación del antígeno más exacta que la de la quimioluminiscencia, que se realiza durante un estado dinámico. El rojo Nilo no puede usarse para teñir bandas en membranas de PVDF; sin embargo es posible realizar la tinción en el gel SDS - PAGE y luego transferirlo a la membrana de PVDF. Esto último presenta una ventaja, y es que no es necesario realizar una tinción del gel para comprobar la transferencia proteica.[12] Sistema avidina/estreptavidinaOtra opción es emplear un anticuerpo primario unido covalentemente a moléculas de biotina. Después la detección se llevará a cabo con una proteína de unión a la misma, como la estreptavidina o la avidina. Detección con oroOtra técnica, conocida como Golden blot, consiste en la detección de los anticuerpos primarios con proteína A unida a partículas de oro. El resultado es una membrana con manchas rojas, causadas por el oro, allí donde está la proteína.[13] La sensibilidad del método puede incrementarse con una tinción fotoquímica de plata: el oro cataliza la reducción de los iones plata a plata metálica en presencia de otro agente reductor, como la hidroquinona; la plata forma de este modo unas manchas visibles bajo microscopio óptico. Se consigue así una sensibilidad comparable a la de las técnicas radiactivas.[7][14] Método en un pasoHistóricamente la detección se ha realizado en dos pasos por la relativa facilidad que supone producir anticuerpos primarios y secundarios en procesos separados. Esto ofrece a investigadores y empresas enormes ventajas en lo que a flexibilidad se refiere, y añade un efecto amplificador al proceso. Sin embargo, debido a la llegada de los análisis de proteínas de alto rendimiento y los menores límites de detección ha crecido el interés por desarrollar sistemas de detección en un solo paso que aumentarían la rapidez del proceso al tiempo que reducen los consumibles. Esto requiere un anticuerpo que reconozca al mismo tiempo la proteína de interés y una "etiqueta" detectable. Se incuba de manera similar al anticuerpo primario del proceso en dos pasos, y, tras una serie de lavados, ya se puede detectar directamente. AnálisisTras el lavado de las sondas marcadas no unidas, se procede a la detección de aquellas que sí se han unido a la proteína de interés. Detección colorimétricaLa detección colorimétrica depende de la incubación del Western blot con un sustrato que reacciona gracias a la enzima reporter (una peroxidasa, por ejemplo) unida al anticuerpo secundario. Pasa así de ser un compuesto soluble a una forma insoluble de otro color que precipita cerca de la enzima tiñendo así la membrana. Se procede después a un lavado en el que se elimina el tinte soluble. La cuantificación proteica se evalúa por densitometría (cuan intensa es la mancha) o por espectrometría. Detección quimioluminiscenteLa detección quimioluminiscente requieren la incubación de la membrana con un sustrato, que emitirá luminiscencia al ser expuesto al reporter que trae unido el anticuerpo secundario. La luz captada por una película fotográfica o, más recientemente, por cámaras CCD, que toman una imagen digital del Western blot. Se analiza la imagen por densitometría para evaluar la cantidad relativa de mancha y cuantifica el resultado en términos de densidad óptica. Actualmente hay programas que permiten realizar análisis más profundos si se aplican ciertos estándares, como la obtención del peso molecular.  Detección radioactivaLos marcadores radiactivos no requieren sustratos enzimáticos; en su lugar se coloca una película fotográfica contra el Western blot y la actividad radiactiva del marcador provoca la aparición en ella de regiones oscuras. Estas se corresponderán con las bandas de las proteínas de interés. Su alto precio y su riesgo para la salud y la seguridad han provocado su desuso en los últimos tiempos. Detección fluorescenteEn la detección con marcadores fluorescentes se procede a la excitación lumínica de éstos que les provoca una excitación. Ésta es detectable por un fotosensor, como una cámara CCD equipado con los filtros de emisión apropiados. Se consigue así una imagen digital del Western blot que puede ser analizado para obtener el peso molecular o la cuantificación proteica. La fluorescencia es considerada uno de los métodos de análisis más sensibles. Exploraciones secundariasUna característica de las membranas de PVDF, de la que carecen las de nitrocelulosa, es su capacidad de quitar los anticuerpos y volver a explorar la membrana con otros anticuerpos. Aunque hay protocolos que permiten hacer lo mismo en membranas de nitrocelulosa, la robustez de las de PVDF facilita la eliminación de los anticuerpos, permitiendo una mayor reutilización antes de que el ruido de fondo sea tan grande que impida continuar con los experimentos. Otra diferencia es que, a diferencia de la nitrocelulosa, el PVDF debe ser empapado en etanol, isopropanol o metanol al 95% antes de ser usado. Además, las membranas de PVDF tienden a ser más delgadas y más resistentes a los daños durante su uso. AplicacionesInvestigaciónActualmente, el Western blot es una de las técnicas más usadas en el estudio de la biología molecular. Diagnóstico
Protocolos
Véase también
Referencias
Enlaces externos |