|
PeptidomiméticoUn péptidomimético es una estructura similar a la proteína, diseñada para imitar un péptido, capaz de reemplazarlo en sus interacciones con receptores y enzimas.[1] Por lo general, surgen típicamente de la modificación de un péptido ya existente, o del diseño de sistemas similares que imitan péptidos, como los peptoides y los péptidos β, para alterar las propiedades de la molécula. Por ejemplo, pueden surgir de modificaciones para cambiar la estabilidad de la molécula o su actividad biológica, y ello puede desempeñar un papel en el desarrollo de compuestos farmacológicos a partir de los péptidos existentes. Estas modificaciones implican cambios sobre los péptidos que no suceden de forma natural (como alteraciones en la columna vertebral y la incorporación de aminoácidos no naturales). Según su similitud con el péptido precursor, los peptidomiméticos se pueden agrupar en cuatro clases (A - D) donde A presenta la mayor cantidad de similitudes y D. Las clases A y B involucran andamios similares a péptidos, mientras que las clases C y D incluyen moléculas pequeñas (Figura 1).[2] 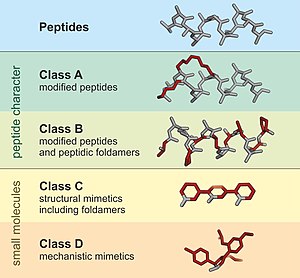 EstructuraLos peptidomiméticos son un grupo químicamente heterogéneo de compuestos orgánicos de bajo peso molecular. A pesar de una gran cantidad de cambios, generalmente se puede observar una similitud estructural más o menos pronunciada con los péptidos. Las modificaciones de la cadena principal del péptido incluyen, en particular, un intercambio de los grupos péptido amida por grupos bioisostéricos. Las modificaciones simples del esqueleto del péptido también incluyen alquilaciones del nitrógeno de la amida. Numerosos peptidomiméticos incluyen ciclaciones que se han introducido con el propósito de restricción conformacional y estabilización de la estructura tridimensional. D-péptidos Un D-péptido es una pequeña secuencia de D-aminoácidos. Dado que los ribosomas son específicos de los L-aminoácidos, los D-péptidos rara vez se encuentran naturalmente en los organismos y no se digieren ni degradan fácilmente. Los peptidomiméticos D-péptidos diseñados para imitar los L-péptidos naturales comúnmente tienen propiedades terapéuticas. Las enzimas responsables de la hidrólisis de péptidos no tendrán una alta afinidad por los D-péptidos dada la estereoquímica inversa de los péptidos que se encuentran normalmente en el cuerpo. También tienen una respuesta inmune débil, disminuyendo la inflamación.[3]  β-péptidosLos β-péptidos son péptidos que tienen dos carbonos entre el carbonilo de la amida y el nitrógeno de la amida precedente. La estructura helicoidal sería más estable que en los péptidos normales.[4]  PeptoidesEn general, los peptidomiméticos están hechos para imitar ciertos péptidos activos mientras tienen una vida media más larga en el cuerpo o una mayor afinidad.  EjemplosLos enfoques peptidomiméticos se han utilizado para diseñar moléculas pequeñas que matan selectivamente las células cancerosas, un enfoque conocido como quimioterapia dirigida, al inducir la muerte celular programada mediante un proceso llamado apoptosis. Los siguientes dos ejemplos imitan las proteínas involucradas en las interacciones proteína-proteína clave que reactivan la vía apoptótica en el cáncer pero lo hacen por mecanismos distintos. En 2004, Walensky y sus colaboradores informaron sobre un péptido alfa helicoidal estabilizado que imita las proteínas pro-apoptóticas.[5] Esta molécula fue diseñada para estabilizar la estructura helicoidal nativa formando un macrociclo entre cadenas laterales que no están involucradas en la unión. Este proceso, denominado péptido engrapado, utiliza aminoácidos no proteinogénicos para facilitar la macrociclación mediante la metátesis de olefina de cierre de anillo.[6] Los heterociclos se usan a menudo para imitar el enlace amida de los péptidos. Los tiazoles, por ejemplo, se encuentran en péptidos naturales y los investigadores los utilizan para imitar el enlace amida del péptido. Como análogos resistentes a la escisión de sustratos enzimáticos, los peptidomiméticos se usan como inhibidores enzimáticos. Un conocido ligando de receptor peptidomimético es la morfina de la adormidera, que es un análogo de la secuencia peptídica de las endorfinas del cuerpo y activa los receptores opioides. Debe su acción analgésica a que es capaz de unirse a los receptores de unos péptidos llamados encefalinas. Usos
Referencias
|