|
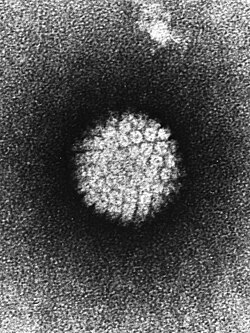
PapillomaviridaePapillomaviridae es una familia de virus que infectan vertebrados.[1][2][3] Además, se conocen más de 100 diferentes tipos de papilomavirus humanos (VPH). Tienen un genoma ADN bicatenario y por lo tanto pertenecen al Grupo I de la Clasificación de Baltimore. Su estructura se caracteriza por no presentar envoltura, la cápside es de simetría icosaédrica y mide unos 60 nm de diámetro. El virus del papiloma se identificó por primera vez a principios del siglo XX, cuando se demostró que las verrugas podrían transmitirse entre personas por un agente infeccioso filtrable. En 1935 Francis Peyton Rous, que había demostrado previamente la existencia de un cáncer que causa el sarcoma viral en pollos, demostró que el virus del papiloma puede causar cáncer de piel en conejos infectados. Esta fue la primera demostración de que un virus podía causar cáncer en los mamíferos. Papilomavirus humanosEl virus del papiloma se replica exclusivamente en la superficie de los tejidos corporales como la piel, mucosas, genitales, ano, boca o vías respiratorias.[4] La mayoría de los tipos de virus del papiloma se adaptan a la infección a una superficie corporal particular. Por ejemplo, los VPH de tipos 1 y 2 tienden a infectar las plantas de los pies o las palmas de las manos, respectivamente, donde pueden causar verrugas.[5] Los cánceres causados por una docena de tipos de VPH de "alto riesgo" matan varios cientos de miles de personas al año en todo el mundo y son uno de los focos importantes de investigación en salud pública.[6] Papilomavirus animales Las distintas especies de virus del papiloma tienden a ser muy específicas en la replicación, que suele realizarse en una sola especie animal. En un estudio los investigadores intercambiaron piel de una gran variedad de animales en un zoológico y utilizaron PCR para amplificar el ADN del virus que debería estar presente.[7] A pesar de la amplia variedad de secuencias del virus del papiloma que se identificaron en el estudio, los autores encontraron pocas pruebas de transmisión interespecífica. Curiosamente, un cuidador resultó positivo transitoriamente a la secuencia de un virus del papiloma específico del chimpancé. Sin embargo, los autores señalan que la secuencia detectada podría haber sido el resultado de contaminación superficial en la piel del cuidador, frente a una infección productiva. El virus del papiloma de la cola de algodón del conejo (CRPV) puede causar verrugas protuberantes en su huésped nativo, el conejo de América del Norte del género Sylvilagus. Los conejos domésticos europeos del género Oryctolagus pueden ser infectados en el laboratorio temporalmente con el CRPV. Sin embargo, puesto que los conejos domésticos Europea no producen progenie infecciosas del virus, se consideran como un "callejón sin salida" para el CRPV.[8] La transmisión interespecífica ha sido también documentada para el papilomavirus bovino de tipo 1.[9] En su huésped natural, el ganado, BPV-1 produce la piel grandes verrugas fibrosas en la piel. La infección por BPV-1 en el caballo, que es un hospedador accidental para el virus, puede conducir al desarrollo de tumores benignos conocidos como sarcoides. La importancia de BPV-1 en la ganadería ha conducido al éxito en los esfuerzos para desarrollar una vacuna contra el virus. En algunos estudios se han identificado virus del papiloma en pequeños roedores, tales como hámsteres dorados, ratas africanas y ratones de campo europeos.[10] Sin embargo, no se conocen virus del papiloma capaces de infectar al ratón de laboratorio. La falta de un virus modelo que infecte fácilmente al ratón ha sido una importante limitación en el laboratorio para la investigación de los virus del papiloma. Estructura Los virus del papiloma no presentan envoltura. Una sola proteína viral, denominada L1, es necesaria y suficiente para la formación de un cápside de 60 nm compuesta por 72 capsómeros con forma de estrella. Como la mayoría de los virus sin envoltura, la cápside es geométricamente regular y presentan simetría icosaédrica. Las partículas auto-ensambladas similares al virus compuestas de L1 son la base de un grupo de las vacunas profilácticacontra el virus, diseñados para generar anticuerpos neutralizantes del virus que protegen contra la infección inicial. El genoma es una molécula circular de ADN bicatenario de aproximadamente 8000 pares de bases de longitud. Es ensamblado dentro de la cubierta L1 junto con las proteínas histonas celulares, que envuelven y condensan el ADN. La cápside viral también contiene una proteína conocida como L2, que es menos abundante. Aunque no está claro cómo se organiza L2 en el virión, se sabe que realiza varias funciones importantes, en particular, facilita el ensamblado del genoma viral en los viriones nacientes, así como la entrada del virus en nuevas células huésped. L2 es interesante como un posible objetivo para vacunas de protección más amplia contra el virus. Evolución y taxonomíaLa evolución de los virus del papiloma es lenta en comparación con muchos otros tipos de virus. Esto probablemente se debe a que el genoma del virus del papiloma está compuesto por DNA de doble cadena genéticamente estable que es replicado con alta fidelidad por la maquinaria de la célula huésped. Se cree que el virus del papiloma generalmente co-evoluciona durante muchos años con una especie particular de huésped animal. Como ejemplo particular, el VPH-16 que afecta a los humanos ha evolucionado ligeramente y en la actualidad varía en diferentes regiones geográficas de una forma que probablemente refleja la historia de la migración humana.[11][12] Otros tipos de VPH, tales como VPH-13, varían relativamente poco en las diferentes poblaciones humanas. De hecho, la secuencia de VPH-13 se parece a un virus del papiloma de los bonobos (también conocidos como chimpancés pigmeos).[13] No está claro si esta similitud se debe a la reciente transmisión entre especies o porque el VPH-13, simplemente ha cambiado muy poco en los seis o más millones de años desde que los seres humanos y bonobos divergieron.[12] Los papilomavirus presentan organizaciones genómicas similares, y cualquier par de especies contiene al menos cinco genes homólogos, aunque la secuencia de nucleótidos puede diferir en más de un 50%. Los algoritmos filogenéticos que permitan la comparación de homologías llevado a árboles filogenéticos que tienen una topología similar independiente de los genes analizados. El análisis filogenético sugiere fuertemente que normalmente los virus evolucionan junto a las especies de mamíferos y aves que actúan como huéspedes, no cambian de especie huésped, no se recombinan, y han mantenido su organización base genómica, por un período superior a 100 millones de años. Esta comparación de secuencias han sentado las bases de una taxonomía de los papilomavirus, que es ahora reconocida oficialmente por el Comité Internacional de Taxonomía de Virus. Todos los papilomavirus se incluyen en la familia Papillomaviridae. Las principales ramas del árbol filogenético son considerados géneros, que se identifican por letras griegas. Las ramas inferiores se consideran especies y unen tipos que son genómicamente distintos pero no exhiben diferencias biológicas conocidas. Este nuevo sistema taxonómico no afecta a la tradicional identificación y caracterización de "tipos" y muestras con diferencias genómicas pequeñas, conocidas como "subtipos" y "variantes", todos los cuales son taxones por debajo del nivel de "especie".[14] Referencias
Véase también |