|
Estructura genéticaLa estructura genética es la organización de elementos de secuencia especializados dentro de un gen. Los genes contienen la información necesaria para que las células vivas sobrevivan y se reproduzcan.[1][2] En la mayoría de los organismos, los genes están hechos de ADN, donde la secuencia particular de ADN determina la función del gen. Un gen se transcribe (copia) del ADN al ARN, que puede ser no codificante (ncRNA) con una función directa, o un mensajero intermedio (ARNm) que luego se traduce en proteína. Cada uno de estos pasos está controlado por elementos de secuencia específicos, o regiones, dentro del gen. Por lo tanto, cada gen requiere múltiples elementos de secuencia para ser funcional. Esto incluye la secuencia que realmente codifica la proteína funcional o ncRNA, así como múltiples regiones de secuencia reguladora. Estas regiones pueden ser tan cortas como unos pocos pares de bases, hasta muchos miles de pares de bases de largo. Gran parte de la estructura genética es ampliamente similar entre eucariotas y procariotas. Estos elementos comunes resultan en gran medida de la ascendencia compartida de la vida celular en los organismos hace más de 2 mil millones de años.[3] Las diferencias clave en la estructura génica entre eucariotas y procariotas reflejan su maquinaria divergente de transcripción y traducción.[4][5] Comprender la estructura de los genes es la base para comprender la anotación, la expresión y la función de los genes.[6] Características comunesLas estructuras de los genes eucariotas y procariotas involucran varios elementos de secuencia anidados. Cada elemento tiene una función específica en el proceso de múltiples pasos de la expresión génica. Las secuencias y longitudes de estos elementos varían, pero las mismas funciones generales están presentes en la mayoría de los genes.[2] Aunque el ADN es una molécula bicatenaria, típicamente solo una de las cadenas codifica información que la ARN polimerasa lee para producir ARNm codificante de proteínas o ARN no codificante. Este 'sentido' o hebra 'codificación', se ejecuta en el 5' a 3' dirección, donde los números se refieren a los átomos de carbono de la cadena principal de azúcar ribosa. El marco de lectura abierto (ORF, del inglés open reading frame) de un gen, por lo tanto, generalmente se representa como una flecha que indica la dirección en la que se lee la cadena sensorial.[7] Las secuencias reguladoras se encuentran en las extremidades de los genes. Estas regiones de secuencia pueden estar al lado de la región transcrita (el promotor) o separadas por muchas kilobases (potenciadores y silenciadores).[8] El promotor está ubicado en el extremo 5 'del gen y está compuesto por una secuencia promotora central y una secuencia promotora proximal. El promotor central marca el sitio de inicio para la transcripción uniendo la ARN polimerasa y otras proteínas necesarias para copiar ADN en ARN. La región promotora proximal se une a factores de transcripción que modifican la afinidad del promotor central por la ARN polimerasa.[9][10] Los genes pueden estar regulados por múltiples secuencias potenciadoras y silenciadoras que modifican aún más la actividad de los promotores mediante la unión de proteínas activadoras o represoras.[11][12] Potenciadores y silenciadores pueden ubicarse distantemente del gen, a miles de pares de bases de distancia. La unión de diferentes factores de transcripción, por lo tanto, regula la velocidad de iniciación de la transcripción en diferentes momentos y en diferentes células.[13] Los elementos reguladores pueden superponerse entre sí, con una sección de ADN capaz de interactuar con muchos activadores y represores competidores, así como con la ARN polimerasa. Por ejemplo, algunas proteínas represoras pueden unirse al promotor central para evitar la unión de la polimerasa.[14] Para los genes con múltiples secuencias reguladoras, la tasa de transcripción es el producto de todos los elementos combinados.[15] La unión de activadores y represores a múltiples secuencias reguladoras tiene un efecto cooperativo en el inicio de la transcripción.[16] Aunque todos los organismos usan activadores y represores transcripcionales, se dice que los genes eucariotas están 'desactivados por defecto', mientras que los genes procariotas están 'activados por defecto'.[5] El promotor central de los genes eucariotas generalmente requiere una activación adicional por parte de los elementos promotores para que se produzca la expresión. El promotor central de los genes procariotas, por el contrario, es suficiente para una expresión fuerte y está regulado por represores. Se produce una capa adicional de regulación para los genes que codifican proteínas después de que el ARNm ha sido procesado para prepararlo para la traducción a la proteína. Solo la región entre los codones de inicio y parada codifica el producto proteico final. Las regiones no traducidas flanqueantes (UTR, del inglés flanking untranslated regions) contienen secuencias reguladoras adicionales.[17] El 3 'UTR contiene una secuencia de terminación, que marca el punto final para la transcripción y libera la ARN polimerasa.[18] El 5 'UTR se une al ribosoma, que traduce la región codificante de la proteína en una cadena de aminoácidos que se pliegan para formar el producto proteico final. En el caso de genes para ARN no codificantes, el ARN no se traduce, sino que se pliega para ser directamente funcional.[19][20]  Eucariotas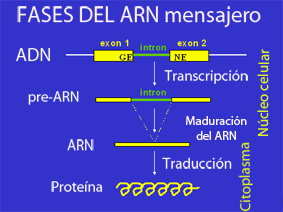 La estructura de los genes eucariotas incluye características que no se encuentran en los procariotas. La mayoría de estos se relacionan con la modificación postranscripcional de pre-ARNm para producir ARNm maduro listo para la traducción en proteína. Los genes eucariotas suelen tener más elementos reguladores para controlar la expresión génica en comparación con los procariotas.[5] Esto es particularmente cierto en eucariotas multicelulares, humanos, por ejemplo, donde la expresión génica varía ampliamente entre los diferentes tejidos.[11]  Una característica clave de la estructura de los genes eucariotas es que sus transcripciones generalmente se subdividen en regiones de exón e intrón. Las regiones de exón se retienen en la molécula de ARNm madura final, mientras que las regiones de intrón se empalman (se extirpan) durante el procesamiento post-transcripcional.[22] De hecho, las regiones intrónicas de un gen pueden ser considerablemente más largas que las regiones exónicas. Una vez empalmados, los exones forman una única región continua que codifica proteínas, y los límites de empalme no son detectables. El procesamiento post-transcripcional eucariota también agrega una tapa de 5' al comienzo del ARNm y una cola de poliadenosina al final del ARNm. Estas adiciones estabilizan el ARNm y dirigen su transporte desde el núcleo al citoplasma, aunque ninguna de estas características está codificada directamente en la estructura de un gen.[17] ProcariotasLa organización general de los genes procariotas es marcadamente diferente de la de los eucariotas. La diferencia más obvia es que los marcos abiertos de lectura (ORF) procarióticos a menudo se agrupan en un operón policistrónico bajo el control de un conjunto compartido de secuencias reguladoras. Todos estos ORF se transcriben en el mismo ARNm y, por lo tanto, se regulan conjuntamente y a menudo cumplen funciones relacionadas.[23][24] Cada ORF generalmente tiene su propio sitio de unión al ribosoma (RBS) de modo que los ribosomas traducen simultáneamente los ORF en el mismo ARNm. Algunos operones también muestran acoplamiento traduccional, donde las tasas de traducción de múltiples ORF dentro de un operón están vinculadas.[25] Esto puede ocurrir cuando el ribosoma permanece unido al final de un ORF y simplemente se transloca al siguiente sin la necesidad de un nuevo RBS.[26] El acoplamiento traduccional también se observa cuando la traducción de un ORF afecta la accesibilidad del siguiente RBS a través de cambios en la estructura secundaria del ARN.[27] Tener múltiples ORF en un único ARNm solo es posible en procariotas porque su transcripción y traducción tienen lugar al mismo tiempo y en la misma ubicación subcelular.[28] La secuencia del operador al lado del promotor es el principal elemento regulador en los procariotas. Las proteínas represoras unidas a la secuencia del operador obstruyen físicamente la enzima ARN polimerasa, evitando la transcripción.[29][30] Los riboswitches son otra secuencia reguladora importante comúnmente presente en las UTR procariotas. Estas secuencias cambian entre estructuras secundarias alternativas en el ARN dependiendo de la concentración de metabolitos clave. Las estructuras secundarias bloquean o revelan regiones de secuencia importantes, como los RBS. Los intrones son extremadamente raros en los procariotas y, por lo tanto, no juegan un papel importante en la regulación del gen procariota.[31] Referencias
Enlaces externos
|